Zusammenfassung des ersten Teils:
Im ersten Abschnitt wurde eine Arbeitshypothese aufgestellt. Der zweite Abschnitt widmete sich ausführlich der Pharmakokinetik der ionisierbar lipidischen Nanopartikel und der Lipidnanopartikel (LNP). Im dritten Abschnitt wurden die grundlegenden Eigenschaften und Unbekannten der modRNA untersucht.
Die Hypothese lautet:
Weder Biodistribution noch Transfektionsraten sind in vivo tatsächlich kontrollierbar.
LNPs sind durch ihre intrinsische Natur bioaktiv und verändern das gesamte Zellverhalten aufgrund tiefgreifender Eingriffe in das nichtlineare, zelltypabhängige Signaltransduktionsnetzwerk.
Bereits der Zellmembrankontakt mit LNPs löst die Signaltransduktionskaskaden rezeptorabhängig und -unabhängig in einer Art aus, die so unter physiologischen Bedingungen nicht zu beobachten ist.
Die daraus folgenden Konsequenzen sind weitreichend und unzureichend charakterisiert.
Darüber hinaus wird die modRNA weitere antagonistische und synergistische Prozesse forcieren.
Stilistische Limitation für diesen Teil ist, wie gehabt, die maschinelle Übersetzung, und neu dazu kommen die Fallberichte: Normalerweise würde man diese als “Fußnote x – n” deklarieren. (Substack-Limitation)
4. LNPs und modRNA – Die pharmakodynamische Realität
Dieser Abschnitt untersucht, ob die beobachteten Nebenwirkungen nach modRNA/LNP-Immunisierung, darunter Myokarditiden, idiopathische Autoimmunerkrankungen und Metastasenbildung, auf Kaskaden zurückzuführen sind, die sich mechanistisch und biologisch plausibel erklären lassen. Die Datenverfügbarkeit und Komplexität biologischer Prozesse in vivo begrenzen die Analyse. LNPs und modRNA sind in vivo nicht eindeutig trennbar. Dies stellt eine Einschränkung dar – insbesondere in realen Fallberichten und Datenerhebungen von modRNA-Transfizierten. Es werden in diesem ausführlichen Abschnitt diverse plausible und experimentell falsifizierbare mechanistische Perspektiven diskutiert. Diese rücken den Fokus auf die intrazellulare Perspektive.
4.1 Die Membran und LNPs
Um ein mechanistisches Grundverständnis zu entwickeln, werden zunächst, wie bereits in Abschnitt 1.1 skizziert, die Folgen der Membranüberwindung geklärt. Weiter wird untersucht, ob der endosomale Escape ohne Folgeschäden bleibt. Dies wird in diesem und den folgenden Unterabschnitten näher betrachtet.
Da die Membranüberwindung – wie in 1.1 und 2.1 ausgeführt – eine wesentliche Rolle spielt, werden zuerst die Auswirkungen auf die Membranreorganisation untersucht. Während sich die meisten Diskussionen vorrangig um das SP-Narrativ drehen, wird übersehen, dass auch ein alternatives Erklärungsmodell in Betracht gezogen werden sollte.
Unter diesem Aspekt ist es wichtig zu erörtern, wie die einzelnen Rezeptoren einer Zellmembran organisiert werden:1234 Lipid-Rafts sind spezielle Zellmembrandomänen, in denen veränderte Verhältnisse von Sphingolipiden, Cholesterin und gesättigten Phospholipiden vorliegen, wodurch sich eine andere Permeabilität ergibt. Durch die damit zusätzlich geschaffenen räumlichen und elektrischen Spannungsunterschiede – im Vergleich zur umgebenden Membran – dienen diese Domänen der Rezeptoranlagerung (receptor clustering) und verankern Signalmoleküle (Phosphatidylinositole [PtdIns] als erste intrazellulare Botenmoleküle [vgl. Abschnitt 4.2]).
4.1.1 Die Membranüberwindung
In Teil 1, Abschnitt 2 wurde darüber hinaus nahegelegt, dass sowohl rezeptorabhängige als auch rezeptorunabhängige endozytoseähnliche Aufnahmeprozesse der LNP/modRNA geschehen (Membranüberwindung). Wie in Teil 1, Abschnitt 2.3 gezeigt, können zahlreiche Zelltypen, inklusive B-Zellen, T-Zellen, Makrophagen, und dendritische Zellen transfiziert werden. Es ist daher anzunehmen, dass es bereits hier zu einer Rezeptorverschiebung kommen wird. Das heißt, dass sie eine veränderte Konformation, Anordnung (Translokation) oder ähnliche Effekte erfahren. Dies wird im Folgenden mechanistisch näher ausgeführt.
Wenn – wie von Ermilova und Swenson sowie Er-Rafik et al. bestätigt (Teil 1, Ermilova, 2023 [Fußnote 114]; Teil 1, Er-Rafik, 2022 [Fußnote 115]) – die Membran penetriert wird, hat es direkte Folgen für die Membranreorganisation und die Lipid-Rafts, in denen Rezeptorproteine, wie G-Protein-gekoppelte Rezeptoren (GPCRs) gebunden sind.
Lipid-Raft-Verschiebungen sind mit vielen pathogenen Mechanismen assoziert, darunter Alzheimer, Prionenerkrankungen und Muskeldegenerationen.5 6 7
Wie zahlreiche Fallberichte zeigen, gibt es Anzeichen für diese Dysregulationen.8 9 10 11 12 Es bilden sich Autoantikörper gegen diverse zellinterne Proteine wie MDA5, SRP und Interferon-1 (IFN-1)-Rezeptorproteine. Die erste Frage, die sich daraus ergibt, und die kurz erörtert werden muss: Gibt es Gemeinsamkeiten auf der zellulären Ebene? Dazu werden im Folgenden sowohl die GPCR sowie ihr regulatorischer Einfluss auf Autoantikörper als auch die Lipid-Rafts und deren Einfluss auf die GPCR untersucht.
GPCRs sind essenziell für die Migration und Regulation von T-Zellen und B-Zellen.13 14 15 16 Darüber hinaus – und das ist die eigentliche Gemeinsamkeit – stehen GPCRs häufig, wenn nicht sogar zumeist, in Wechselwirkung und wechselseitiger Regulation mit Immunrezeptoren wie den Toll-like-Rezeptoren (TLR) und Tyrosinrezeptoren. Dabei spielen nachgeschaltete Feedforward- und Feedbackmechanismen eine entscheidende Rolle.17 18 19 20 21 22
Wenn eine Lipid-Raft-Verschiebung oder Dysregulation stattfindet, ist zu erwarten, dass die GPCR mit als eine der ersten Rezeptorfamilien eine funktionelle Veränderung erfahren. Die wahrscheinlichsten Konsequenzen dieses Mechanismus sind klinisch heterogene Erscheinungsbilder, die sich durch fehlende Muster und heterogene Autoimmunreaktionen, von denen viele idiopathischer Natur sind, auszeichnen. Dies entspricht exakt den oftmals als “anekdotisch” klassifizierten Fallberichten, von denen mittlerweile mehr als 3 000 in Journals wie MDPI, Cureus, Oxford Academic, Springer, SAGE und anderen erschienen sind.23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 (Diese Zahl basiert auf der nicht-offiziellen, öffentlich zugänglichen Datenbank https://react19.org/1250-covid-vaccine-reports/, die ausschließlich auf peer-reviewte Fallberichte verweist; letzter Zugriff: 19. Juni 2025.)
Diese breite und scheinbar heterogene Anhäufung wurde bereits in Teil 1, Abschnitt 2.1 und 2.2 unter dem Aspekt der LNPs und Biodistribution eingeführt, ohne die pharmakodynamischen Aspekte zu diskutieren. Da LNPs eine intrinsische Nichtlinearität in vivo aufweisen, ist hier bereits einer der zentralen Schlüsselmechanismen verborgen, welcher eine nähere Betrachtung benötigt.
Abbildung 1
Schematische Darstellung der Interaktionsebenen; Crosstalk-Beispiele für Signalkaskaden, die nach Rezeptor-Interaktionen (Feedforward-/Feedback-Schleifen) und/oder sonstiger endozytoseähnlicher Aufnahme aktiviert werden [Mitogen activated protein Kinasen (MAPk), Janus Kinase (JAK) / Signal transducer and activator of signal transduction (STAT), Cyclisches Adenosinmonophosphat (cAMP) und mechanistical target of Rapamycin complex (mTORC)]; Endosomaler escape; downstream eIFs (3.1) [Regulation der ribosomalen Kontrolle] und miRNA-Regulation von modRNA Translation; Nuclear Faktor kappa B (NF-ϰB); Kontrollierte Meachanismen im Nukleus, durch NF-ϰB, MAPk, JAK/STAT, cAMP und mTORC1/2 [©F. Seger]
Falls bereits in der Lipid-Raft-Organisation eine Störung vorliegt, ist ebenfalls zu erwarten, dass sich in einigen Fällen GPCR-Autoantikörper bilden können.
Auch Autoantikörper, wie antinukleare Autoantikörper, erscheinen plausibel unter diesem Aspekt, da wie unter 1.1 bereits erwähnt, die Membran-Chemie eine Vielzahl an Kaskaden auslöst und nicht isoliert von LNPs betrachtet werden kann.
Fallahi-Sichani und Linderman schreiben:41
“In addition, microdomains on the cell membrane termed lipid rafts have been shown to play a role in GPCR localization. (…)"
"However, when both receptor dimerization and monomerization are fast, “partner switching”, i.e. alternating of bonds between neighboring receptors, occurs quickly, leading to the formation of oligomer-like clusters of receptors on the cell membrane (oligomer regime) (...)"
Wirklich bemerkenswert ist, dass "Some GPCRs undergo ligand-induced dimerization, while ligand stimulation has either no effect or decreases the level of dimerization in others [4], [11]."
Daraus ergibt sich, dass "(...) dimerization-mediated organization of receptors can be affected differently by ligand stimulation.”
An dieses Zitat anschließend muss die Auswirkung von LNPs auf die Lipid-Rafts erwähnt werden (siehe Abbildung 1 - Schematische Darstellung der Interaktionsebenen).
In ihrem Review merken Wang et al. zu den Lipid-Rafts an:42
”For example, these domains facilitated interactions between LNPs and cellular membranes, improving intracellular uptake via endocytosis (Herrera et al., 2021). Moreover, their ordered nature may mimic lipid rafts on cell membranes, enhancing recognition and internalization regulated by lipid rafts (Wang et al., 2023a, Wang et al., 2023b, Wang et al., 2022a, Wang et al., 2022b).”Cabral-marque et al. kommen zu dem Schluss, dass GPCRs eine treibende Rolle bei der Dysregulierung von Autoantikörperfunktionen spielen und Autoantikörper erst durch diese Dysregulation zu einer pathogenen Natur führen.43 Sie kommen zu dem Schluss, dass gestörte GPCR-Funktionen die Zellwanderung, das Gewebs-Homing und die Immunzellorganisation beeinträchtigen. Das eröffnet Raum für idiopathische Autoantikörperbildung. Sie schreiben dazu:
”Therefore, it is reasonable to assume that dysregulation of subgroups of GPCRs contribute to disease phenotypes by causing abnormal cellular infiltration into inflamed tissues and modulation of the residence time of immune cells at these sites.
In response to exogenous and endogenous factors, GPCRs (21–27) emit signals to the cytoskeleton and to adhesion molecules (e.g. integrins), thereby promoting cell trafficking and cell migration.
They do so under homeostatic as well as pathological conditions when signals from pathogen-associated molecular pattern (PAMPs) and damage-associated molecular patterns (DAMPs) induce the inflammatory response and trigger signal cascades such as those elicited by the interaction between chemokine/chemokine receptors that coordinate the recruitment of leukocytes.”Auch Shao et al. (Shao, 2025 [Fußnote 16]) kommen zu ähnlichen Schlussfolgerungen. Da GPCRs direkt, wie weiter oben ausgeführt, eine entscheidende regulatorische Rolle bei der B-Zell-Differenzierung und B-Zell Regulierung einnehmen, heißt dies im Umkehrschluss, dass sie auch die Reifung und Sezernierung von Autoantikörpern mitregulieren.
Zur Überprüfung, ob es Evidenz für Autoantikörper gibt, die über einzelne Fallberichte hinausgeht und ob diese Untersuchungen transparente Methodiken anwenden, werden noch folgende Studien prägnant diskutiert:
Die erste bemerkenswerte Studie, die eine systematische Übersicht liefern will, wurde von Yalcinkaya et al. verfasst.44 Diese konnte keine Autoantikörper gegen IFN-1 nachweisen. Dabei muss jedoch die Methodik beachtet werden. Zunächst orientierten sich die Autoren an Bastard et al. und setzten ein Detektionslimit von >1500 AUs als statistisch signifikant.45
“The initial multiplex screening (bead-based assay, Luminex) results were confirmed via optimized ELISA methods for several IFNs that were selected due to the presence of at least one sample with elevated response in the bead-based screening. That is, ELISA re-analysis was performed for a certain IFN if at least one ‘elevated response’ had been observed in either the AEFI or the BD group for said IFN (defined as >1500 AUs, based on Bastard et al.14).”Bastard et al. haben jedoch dieses Detektionslimit für Covid-Patienten mit lebensbedrohlicher Covid-19-Symptomatik gesetzt. Es ist fraglich, ob das gewählte Detektionslimit eine verallgemeinerbare Aussagekraft für tatsächliche Autoimmunfolgen besitzt, oder ob nicht vielmehr bereits eine wesentlich geringere Autoantikörperverschiebung bei Geimpften statistisch relevant wäre. Zudem clustern Yalcinkaya et al. in so viele Subgruppen, die sie getrennt analysieren, ohne eine mögliche Gesamttendenz zu berücksichtigen. Dies resultiert in einem doppelten negativen Bias – sowohl durch Übersegmentierung der Subgruppen als auch fehlende Berücksichtigung möglicher Gesamttendenzen. Hinzu kommt die weitere Limitation, dass sie nur 290 Patienten untersuchten und nicht nach Impfstoffart subgruppierten. Stattdessen wurde ausschließlich nach klinischen AEFI-Symptomgruppen (z. B. neurologisch, kardiovaskulär, hämatologisch) geclustert.
Im Gegensatz zu dieser Studie dokumentieren Gazitt et al.:46
"A value above 15 binding antibody units (BAUs) was considered positive, according to the manufacturer’s instructions [18]."Sie nutzten einen – nach WHO-Richtlinie empfohlenen (>15 BAUs = positiv) – Standardtest, um bindungsfähige Autoantikörper zu messen. Sie beobachteten Entzündungsschübe bei 5.8 % der gesunden Kontrollgruppe, die 2 – 3 Dosen BNT162b2 erhalten haben, mit nachgewiesenen Autoantikörpern und 7.2% bei AIIRD-Patienten (54 Gesunde vs. 463 Patienten).
Die Autoren folgern: ”In summary, in our prospective cohort of AIIRD patients and controls, incidences of autoantibody seroconversion after three doses of mRNA COVID-19 vaccination was low, with many of these autoantibodies appearing to be transient.”Diese Schlussfolgerung sollte jedoch aus mehreren Gründen hinterfragt werden:
"Data on the development of autoantibodies were analyzed based on the number of patients with available serology following each vaccine dose. When a baseline time-point was missing prior to obtaining mRNA COVID-19 immunization, the baseline pre-vaccination autoantibody was imputed to be negative only if negative serologic testing results for that particular autoantibody was observed following vaccination."Der erste und statistisch relevante Grund betrifft die Exklusionskriterien. Von den ursprünglich 55 Teilnehmern in der Kontrollgruppe wurden letztlich nur 54 Personen ausgewertet. Von den 686 AIIRD-Patienten blieben 463 in der Untersuchung. Die Kontrollgruppe war deutlich kleiner dimensioniert. Es zeigt sich eine auffällige Diskrepanz zwischen der zweiten und dritten Dosis. Von den 54 Personen der Kontrollgruppe wurden nur noch 32 ausgewertet. Dies entspricht einer Exklusion von 41%. Durch die Serokonversionsdaten entsteht ein methodisch intransparenter Bias, da unklar bleibt, nach welchen Kriterien die Auswahl getroffen wurde, wodurch eine erhebliche Gewichtungsverschiebung in beide Richtungen möglich ist.
Der zweite und mechanistische Grund: Es wurde nirgends erwähnt, ob die Autoantikörperspiegel wieder gesunken sind. Eine Nachverfolgungsdauer von einem Jahr mit stichpunktartigen Messungen nach den Impfdosen reicht für eine Schlussfolgerung von “transienten” Autoantikörpern nicht aus.
Ludwig et al. schreiben in einem Review 2017 zur Natur von Autoantikörpern:47
“The symptoms usually progress to other muscles during the first 2 years, resulting in generalized MG. In about 15% of MG patients, symptoms are restricted to the eye muscles, and this condition is termed ocular MG. In most cases, a specific cause of MG cannot be identified (157).”Während Johnson und Jiang anmerken:48
“Nonetheless, it may take many years to develop a pathogenic autoantibody due to disordered somatic hypermutation and class switch recombination 120.”Song, Yi & Wu bemerken ergänzend zum Sjögren’s Syndrom:49
”Autoantibodies detection is very sensitive and can be detected even 20 years before SS occurrence.229 Antinuclear antibodies (ANAs) are the most common for the majority of patients. Anti-RNA-related protein antibodies (anti-Ro/SSA antibodies) are representative of different clinical stages, histological changes and immunopathological changes.” Unter dieser Berücksichtigung erscheint das Wort “transient” nicht ausreichend belegt. Weder wurden zelluläre Untersuchungen durchgeführt, die diese Aussage stützen noch wurden spezifische Zelloberflächenmarker erfasst, die Aufschluss über die funktionelle Aktivität und den regulatorischen Zustand der B-Zellen hätten geben können. 50 51 52 53 54
Wie oben ausgeführt, kamen Shao et al. (Shao, 2025 [Fußnote 16]) und Cabral-marque et al. (Cabral-marque et al., 2019 [Fußnote 43]) zu dem Schluss, dass die Steuerung und korrekte Balancierung durch GPCRs über Immunität und Pathogenität entscheidet. Daraus lässt sich ableiten, dass nicht die absolute Anzahl der Autoantikörper, sondern deren Verhältnisse und Regulierung (Wo?, Wann?, Wie?) entscheidend über Gesundheit und Krankheit sind.
In diesem Abschnitt wurde ausgeführt, dass nicht die Autoantikörper der eigentlich ursächliche Treiber für zu erwartende pathogene Mechanismen nach einer modRNA/LNP-Transfektion sind. Die Ursache ist zunächst in gestörten Membran-Rezeptor-Interaktionen zu suchen. Dies führt – im Einklang mit den bereits in Teil 1, Abschnitt 2 beschriebenen LNP-Charakteristika – zu einer systemweiten Kettenreaktion.
In den folgenden Abschnitten wird diese Hypothese vertiefend untersucht. Zusammenfassend lässt sich bis hierher festhalten, dass es zu Feedforward-Interaktionen in der Membran kommen wird, welche direkten Einfluss auf die Lipid-Rafts nehmen. Daraus ergeben sich unvermeidbare Feedback-Schleifen.
4.1.2 Die Rezeptornatur am Beispiel der GPCR und der Mitogen Activated Protein Kinasen (MAPKs)
Um zu verstehen, warum Rezeptorclustering das Schicksal zwischen Gesundheit und Pathogenität entscheidet und Störungen dieses Prozesses, wie in 4.1.1. ausgeführt, zu vermuten sind, sollte ein grundlegendes Verständnis der Natur von membrangebundenen Rezeptoren vermittelt werden. Dies wird in diesem Abschnitt diskutiert.
GPCRs zeichnen sich durch sieben hochkonservierte transmembrane α-helikale Strukturmotive aus.55 Diese sieben Strukturmotive variieren in der Gesamtlänge jedes einzelnen GPCRs bis weit über 1.000 Nukleotide, wodurch eine Vielfalt entsteht, welche mit rekombinatorischen Prozessen vergleichbar ist. Daraus ergibt sich die Erkenntnis (Stand 2024), dass mehr als 800 identifizierte GPCRs im menschlichen Organismus existieren.56 GPCRs werden nach Aktivierung vorrangig am C-terminalen Tail an Serin (Ser)- und Threoninresten (Thr) phosphoryliert.57 Dadurch ergibt sich für jeden einzelnen spezifischen GPCR ein einmaliges Phosphorylierungsmuster.58
Rosenbaum et al. schreiben:59
“(GPCRs) mediate most of our physiological responses to hormones, neurotransmitters and environmental stimulants,(…)”Während die Charakteristik und Funktionalität eindeutig zu sein scheint, ist es aus der intrazellularen Perspektive komplex und noch nicht abschließend charakterisiert.
Zunächst wird der Feedforward-Aspekt beleuchtet:
Es verdichten sich seit 2005/2006 die Hinweise, dass ein GPCR multiple Bindungsstellen besitzt und verschiedene konformationelle Zustände einnehmen kann.60 Daraus ergibt sich eine kontext- und bindungsspezifische, nichtlineare Interaktionsvielfalt.
Wie Zhang et al. 2024 vermerken:61
“Human G proteins comprise four major families (Gs, Gi/o, Gq/11, and G12/13) and more than half of GPCRs activate two or more G proteins, each of which exhibits distinct efficacies and kinetics.49 50 The promiscuous coupling leads to fingerprint-like signaling profiles inside the cell, which contributes to the complexity of GPCR signaling.” Diese Erkenntnis legt die Überlegung nahe: GPCRs interagieren nicht ausschließlich mit einer einzelnen G-Protein-Klasse, sondern initiieren ein zelltyp- und kontextabhängiges Signalmuster.
Aus intrazellularer Perspektive – ausgehend von der Membran und wie eine Rezeptorverschiebung (Abschnitt 4.1.1) das gesamte Zellgeschehen maßgeblich beeinträchtigen, verändern und in Richtung Pathogenität verschieben wird – muss geklärt werden, welche Signalkaskaden betroffen sind. Für die GPCRs ist dies – wenn auch noch nicht abschließend geklärt – am besten charakterisiert.
Gavi et al. schreiben in ihrem Reviewartikel, dass GPCRs und Protein Tyrosin Kinasen (PTKs) überlappende Eigenschaften haben und integrativ in Knotenpunkten, wie den MAPKs münden.62 Sie fassen zusammen:
”Cross-talk between G-protein-coupled receptors and tyrosine kinases can occur at several levels, including the receptor-to-receptor level, and at crucial downstream points (e.g. phosphatidylinositol-3-kinase, Akt/protein kinase B and the mitogen-activated protein kinase cascade).”Budd et al. demonstrierten darüber hinaus, dass die MAPK-Aktivierung – nicht auf die Internalisierung von Rezeptoren angewiesen – durch Endozytose initiiert werden kann.63
Naor et al. fassen zusammen:64
”A variety of signaling mechanisms link GPCRs to the four known MAPK cascades (ERK, JNK, p38MAPK and BMK), and they operate simultaneously. Upon stimulation of the GnRHR, all four MAPK cascades are activated to various degrees by a pathway that involves Gq and PKC.”Zorkin und von Zastrow schreiben in ihrem Review:65
“However, it has been challenging to unequivocally show that the endocytosis of EGFR and other RTKs is necessary for the full activation of ERK. Some experiments using dominant-negative mutants and small interfering RNAs (siRNAs) to target proteins involved in endocytosis suggest that this process is required for ERK activation by several RTKs31–34, but many similar experiments reach the opposite conclusion35–37."
Zorkin und von Zastrow diskutieren, ob auch Tyrosinkinaserezeptoren (RTKs) durch endosomale Internalisierung zur Aktivierung der MAPKs beitragen, da einige Studien dies demonstrierten, während andere diesen Zusammenhang nicht bestätigen konnten. Sie schlussfolgern daraus:
"Although it is difficult to reconcile these data, it can be suggested that the contribution of signalling from RTKs to MAPKs in endosomes may depend on how fast internalized RTKs are sorted to degradative compartments in a particular cell type and on the specific experimental conditions.”Bereits aus diesen Beispielen lässt sich zusammenfassend verdeutlichen, dass die MAPKs (hier: kanonischer Extracellular signal-regulated Kinase(ERK)-Signalweg) ein zentraler Regulationshub wachstumsfaktorgesteuerter Signalwege (RTKs) mit Crosstalks zu den GPCRs in Feedforward-Orientierung (downstream) sind. Darüber hinaus steuern die GPCRs gleichzeitig die MAPKs direkt an.
Um das ganze Netzwerk zu verstehen, müssen auch die Feedbackmechanismen betrachtet werden. Shin et al. merken an:66
“In addition, RKIP is a physiological inhibitor of G-protein-coupled receptor kinase (GRK2). After stimulation of the G-protein-coupled receptor (GPCR), RKIP dissociates from Raf-1 and associates with GRK2 and subsequently blocks its activity. This switch is triggered by PKC-dependent phosphorylation of RKIP on S153, and serves to directly connect the ERK pathway with GPCR signaling.”Matsuda et al. Zeigen in ihrem Experiment eine überraschende Beobachtung:
”One of the clones (termed #5–8) encoded the C‐terminal fragment of LAT (Figure 1B)."
Sie fassen zusammen, dass gezeigt wurde "(...) that the tyrosine‐phosphorylated LAT associates with multiple molecules such as PLCγ1 (Finco et al, 1998; Zhang et al, 1998a, 2000)."
Darüber hinaus liefern sie neue Einblicke und öffnen weitere Fragen:
"(...) Since our data raised the possibility that LAT is phosphorylated by the ΔMEKK1‐induced Ser/Thr kinase cascade, we performed a phosphoamino acid analysis of [32P]LAT in response to TCR stimulation. We found that LAT is indeed phosphorylated at both Ser and Thr residues in addition to Tyr under physiological conditions (Figure 1C)."
Insbesondere ist die Beobachtung zu Nf-κB zu beachten. Sie notieren dazu:
"Although ΔMEKK1 can activate the NF‐κB pathway as well as the MAPK pathways (Karin and Delhase, 1998), coexpression of MKP5 (Tanoue et al, 1999), a negative regulator of MAPK family, completely diminished the effect of ΔMEKK1 on clone #5–8, demonstrating the involvement of MAPK(s) (Figure 1D). Consistent with these results, the amino‐acid sequence of LAT contains four possible MAPK phosphorylation sites (Figure 1B).”Bereits aus diesen Erkenntnissen ergeben sich erste mechanistische Schlussfolgerungen, die breite Implikationen für die Signallogik haben: die MAPKs (PKC-MAPK, JNK-MAPK, ERK-MAPK, P38-MAPK, etc.67 68 69) bilden ein komplexes Netzwerk aus kleinen GTPasen, welche in Feedforward- und Feedback-Loops sowohl das Zellschicksal, als auch die Rezeptorfunktion beeinflussen. Des Weiteren – wie schon unter Abschnitt 1.1 erwähnt – lässt sich hier klar eine kompetitive, vom Kontext (Milieus, Zellstatus, Eingangssignal) abhängige Natur der Signaltransduktion erkennen.
Lark et al. schreiben:
”ERK1/2 proteins have been reported to directly phosphorylate several receptor tyrosine kinases. For example, ERK1/2 MAPKs can phosphorylate the epidermal growth factor (EGF) receptor (EGFR) at T669 [7, 8], the major site of Ser/Thr phosphorylation of the receptor.”Da MAPKs nicht nur durch Rezeptorlandschaft aktiviert werden, sondern, wie ausgeführt, deren Schicksal beeinflussen, muss betrachtet werden, wo diese Kaskaden ihr funktionelles Schicksal erfüllen. Dies ist in den meisten Fällen die Aktivierung eines Transkriptionsfaktors. 70 71
Die hier abgeleitete Synthese lässt sich in einer zweischichtigen Darstellung wie folgt ausdrücken:
GPCR → PKC → RKIP → RAF-1 (freigeben) heißt MAPK-Aktivierung (ERK-Signalweg)
↧konkurrieren↥
GPCR → RKIP → GRK2 (blockieren) heißt GPCR bleibt sensitivAvraham & Yarden schreiben 2011: 72
"In contrast to the early phase, which largely engages pre-existing components involved in receptor endocytosis and EGF-induced phosphorylation events, newly induced RNAs and proteins drive the late regulatory mechanisms."
Sie teilen diesen Mechanismus in drei Phasen und schreiben weiter zur ersten Phase:
"The initial wave, up to 45 minutes from stimulation, consists of a limited set of genes, the IEGs(...)"
Für die zweite Phase notieren die Autoren:
"The group of delayed early genes (DEGs; 45–120 minutes) comprises both positively and negatively acting components(...)", und für die 3. Phase:
"The late, secondary response genes (SRGs; >120 minutes) confer stable phenotypes in a cell context-specific manner."
Einer der markantesten und bemerkenswertesten Sätze, der daraus folgt lautet:
"(...) IEGs are induced by a large range of stimuli, and oncogenic retroviruses encode aberrant forms of IEGs (for example, viral FoS and JUN), considerable effort has been devoted to characterizing IEG regulation.”Aus diesen Feststellungen ergibt sich ein Bild, welches sich mit den neusten Erkenntnissen und Diskussionen von Szischik et al., 2024 (Teil 1, [Fußnote 27]), Thiemicke & Neuert, 2023 ([Teil 1, [Fußnote 21]) und Parres-Gold et al., 2025 (Teil 1, [Fußnote 26]) deckt: Raum, Zeit, Input und Kontext sind die primären Reaktionstreiber.
Die hier diskutierte Natur der Rezeptoren und nachgeschalteten Signaltransduktionsnetzwerke (MAPKs, JAK/STAT, mTORC1/2, RHO, HH u.a.) – am Beispiel der GPCRs und MAPKs erläutert – sollte als potenziell dysreguliert in Größenordnungen von Millionen bis Milliarden transfizierter Zellen betrachtet werden. Eine genaue Zahl ist nur schwer abschätzbar (eventuell sogar Billionen), da keine Angaben zur Anzahl der verwendeten LNPs pro Gabe existieren. Wenn man die Logik von Avraham & Yarden – wie in Teil 1 Abschnitt 2 zur Natur der LNPs dargelegt – auf nicht näher definierte Zelltypen adaptiert (z.B. Immunzellen, Kupfferzellen, Epithelzellen u.a.), ergibt sich infolge weiterer parakriner und endokriner Kommunikationskaskaden eine nicht vorhersagbare Zeitspanne von Tagen bis Jahrzehnten, bevor pathomechanistische Konsequenzen zu erwarten sind. 73
4.2 Der Phosphatidylinositol (PtdIns)-Zyklus und MAPK
Die nächste Frage, die geklärt werden muss, ist, ob gestörte Signalwege plausibel sind – und im Kontext der ionisierbaren Lipidpartikel eine unausweichliche Realität darstellen.
Wie ebenfalls in Teil 1, Abschnitt 2 nahegelegt, definiert sich Transfektion durch die gezielte Zellmembranüberwindung, indem die Natur der phospholipiden Doppelschicht und Rezeptoren gezielt umgangen wird (siehe Teil 1, Ermilova und Swenson, 2023 [Fußnote 117] und Teil 1, Er-Rafik, 2022 [Fußnote 115])
Phosphatidylinositide bezeichnen die Derivate der Phosphatidylinositole (PtdIns), die sich durch die phosphorylierbaren Inositolkopfgruppen zur zytosolischen Seite der Membran orientieren.74 Sie sind amphiphile, elektrisch neutrale und integrale Bestandteile der Zellmembran und machen etwa 1%–5% (Van Meer, 2008 [Fußnote 337]) des gesamten Lipidmosaiks aus. Ihre phosphorylierten Derivate (PIPs) sind im Gegensatz dazu elektrisch negativ geladen und im Zytosol funktional wirksam. Trotz ihrer geringen Konzentration im Phospholipid-Membranmosaik ist ihre Rolle bei der Phagozytose, Endozytose, rezeptorvermittelten Endozytose und Makro- und Mikropinozytose essenziell.
Posor et al. beschreiben im Detail den gesamten PtdIns-Zyklus als ein fein abgestimmtes Netzwerk aus dynamischen Übergängen zwischen den einzelnen Phosphorylierungszuständen (PIP2, PIP3 etc.). Der PtdIns-Zyklus steuert die Verarbeitung und den Abbau der aus der Membran entstandenen Endosomen. Bereits während des Endosomeintritts markieren PIPs die Endsomen75, durch Bindung an die Endosom-Membran und fungieren dabei als dynamische Schlüsselmodulatoren der nachgeschalteten Signalwege (beispielsweise GTPasen). Da ihre Phosphorylierung durch Phosphatidylinositol3Kinase (PI3K), Phosphatase and TENsin homolog (PTEN) und nachgeschaltete Signale, wie Akt etc. gesteuert wird, befinden sie sich in einem zyklischen Übergang – etwa von PIP3 zu PIP2 – dessen Dynamik die einzelnen Reifungsprozesse der Endosomen und den endosomal-lysosomalen Übergang reguliert. Ihre Rollen im Gleichgewicht zwischen phospholipidbezogener Wiederverwertung (Recycling) und Abbau sind unerlässlich für die Membranrestrukturierung und Integrität.
Wenn PI3K als Effektorprotein von Rat-Sarkoma (RAS)-GTPasen (MAPK) in die Reorganisation involviert ist,76 77 78 folgt daraus, dass zwei der wichtigsten Signaltransduktionskaskaden bereits bei dem ersten LNP-Kontakt aus dem Fließgleichgewicht gebracht werden. Dabei spielt es keine Rolle, ob eine Rezeptorinternalisierung stattfindet.
4.2.1 PtdIns, Lipid-Rafts, GPCRs - Der rote Faden
Einen weiteren Hinweis liefern Torres et al. 2020:
”Some of the signals controlled by proteins non-permanently bound to membranes produce dramatic changes in the cell’s physiology. Indeed, changes in membrane lipids induce translocation of dozens of peripheral signaling proteins from or to the plasma membrane, which controls how cells behave. We called these changes “lipid switches”, as they alter the cell’s status (e.g., proliferation, differentiation, death, etc.) in response to the modulation of membrane lipids.”In ihrer Diskussion betonen die Autoren, dass funktionale Zustandsänderungen, die auf einer Neukonfiguration der Membranlipide beruhen – insbesondere durch PtdIns-abhängige Rekrutierung oder Ablösung peripherer Signalmoleküle - über das weitere Zellschicksal entscheiden.
Die Autoren liefern ein – für diese Hypothesenschrift – wichtiges Beispiel:
“For example, Akt is translocated from the cytosol to the plasma membrane (switched on) by binding to PtdIns(3,4,5)P3—a lipid formed by PI3 Kinase (PI3K)-mediated phosphorylation of PtdIns(4,5)P2. Interestingly, PI3K is mainly activated by receptor tyrosine kinases (RTKs) or GPCRs as a result of distinct extracellular anabolic cues, such as insulin, growth factors, cytokines and hormones. Alternatively, the tumor suppressor phosphatase and tensin homologue (PTEN) can switch off Akt growth promotion by dephosphorylating PtdIns(3,4,5)P3 to PtdIns(4,5)P2, translocating Akt from the plasma membrane back to the cytosol and terminating its downstream signaling.”Eine weitere Frage, die daraus folgend, geklärt werden muss, ist die nach dem “Dazwischen”. Wie in Teil 1, Abschnitt 2 nahegelegt, wird der endosomale Escape tief in den hier diskutierten PtdIns-Zyklus eingreifen – was sich unmittelbar aus der Natur des Recyclingprinzips dieses Zyklus ableitet.
Omo-Lamai demonstrieren in einem Experiment, bei dem sie spezifisch-endosomale Färbung nutzen, dass eine Endosomale Disruption mit dauerhaften Schäden geschieht.79 Sie dokumentieren:
”Over 80% of the ionizable lipids caused endosomal rupture severe enough to detect by the AO assay."
Die Autoren betonen, dass dies "(..) includes ionizable lipids such as Dlin-MC3-DMA and ALC-0315 which were not significantly inflammatory and did not lead to high mRNA expression."
Eine weitere bemerkenswerte Beobachtung ist, dass "There was thus a weak correlation between mRNA expression and AO-assessed endosomal rupture in our library of LNP formulations.”Bemerkenswert ist die Erkenntnis der Autoren, dass die Schädigung intrinsischer Natur ist – in einfachen Worten: unabhängig von der modRNA-Expressionseffizienz und dem endosomalen Escape (nach gängiger Definition, dass modRNA in das Zytosol entlassen wird). Eine Frage, die hier offen bleibt, was “nicht signifikant inflammatorisch” bedeutet. Denn auch eine unterschwellige chronische Entzündung ist eine dauerhafte Konsequenz. Dies steht im Einklang mit dem Review von Feng et al., die zu dem Schluss kommen, dass Nanopartikel prinzipiell die endosom-lysosomale Funktion verändern und beschädigen.80
Auch hier muss noch kurz die Frage nach der mechanistischen Plausibilität dahinter geklärt werden. Die Antwort darauf liefern Jörgensen et al. (Teil 1, Jörgensen et al., 2023, [Fußnote 35]):
"However, they are neither synthesized from endogenous or natural structures nor are they degraded into typical biocompatible building blocks."Daraus lässt sich eine kontinuierliche Störung des gesamten Recyclingzyklus ableiten.
4.3 “omische” Evidenz und weitere Daten, die auf eine MAPK-Dysregulierung hindeuten
In diesem Abschnitt wird die Verifizierbarkeit oder Falsifizierbarkeit der bis hierher geführten Bestandsaufnahme und Überlegungen, welche sich daraus ergeben, diskutiert und direkte transkriptomische und proteomische Evidenz und ähnliche Genexpressionsanalysen nach der modRNA-Injektion in vivo für die spätere Diskussion zusammengefasst und kurz besprochen.
4.3.1. “omische” Evidenz als Ausgangspunkt für Falsifizierbarkeit dieser Hypothesenschrift
Um diese theoretischen Gedanken in der Praxis messen und die hier aufgestellten Hypothesen falsifizieren zu können, muss demnach der Fokus auf “omischen” Shotgunanalysen liegen. Kurz: Wie weist man eine diffuse Reaktionskaskade nach, deren Effekte zunächst unauffällig verlaufen und Zeit benötigen, bis ihre kumulativen Synergien und Antagonismen ihre pathogenen Konsequenzen entfalten?
Es empfiehlt sich beispielsweise:
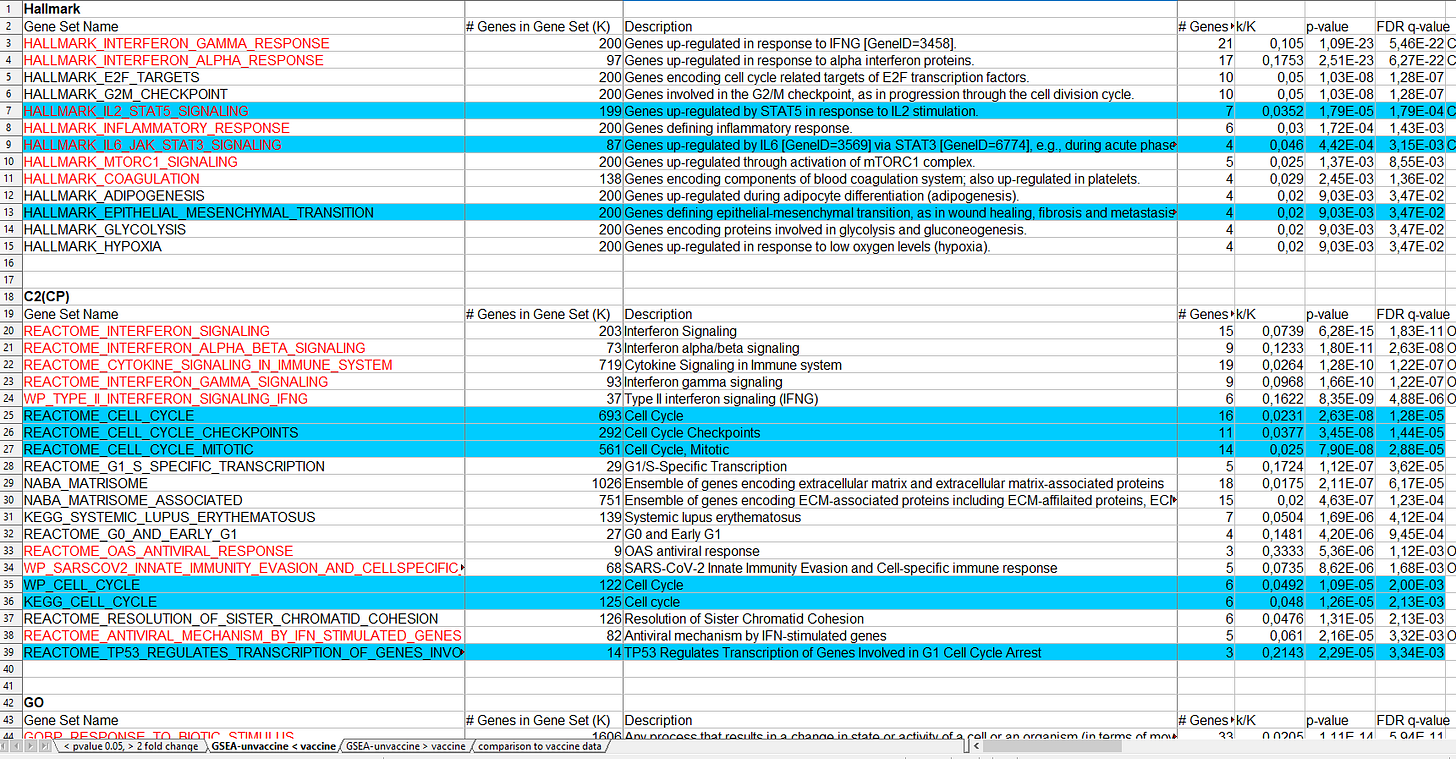
**Transkriptomik** → GSEA (KEGG, Reactome)
Ziel: Validierung von veränderten, signalwegausgelösten Expressionstrends
**Proteomik** → IPA; KEGG, Reactome, STRING (Gesamtprotein-Level)
Ziel: Aktivitätsvalidierung [nicht jede Expression heißt aktives Protein]
**Phosphoproteomik** → KSEA, PTM-SEA
Ziel: Signalwegaktivierung genau charakterisieren und Aktivitätsgrad bestimmen
**Single Cell Analyse** → FRET/FLIM, scWestern, CyTOF / Mass Cytometry
Ziel: heterogene Signalantworten im zellulären Kontext entschlüsseln
4.3.2. “omische” Evidenz die für Signaldysregulation spricht
Dieser theoretische Ansatz und weitere werden im Folgenden anhand Realität und Anwendung von BNT162b2 und mRNA-1273 näher betrachtet. Zudem wird hier weiter die Annahme, dass die MAPK-Signalwege (und weitere) dysreguliert sind verfolgt.
Ndeupen et al. liefern zur Analyse, wie ein biologisches System auf leere LNPs reagiert. Sie publizieren 2021 eine exemplarische Pionierarbeit in vivo.81 Ihre pharmakokinetische Studie, in der sie Wildtyp(WT)-C57BL/6(B6) Mäuse mit 10 μg transfizierten, zeigte, dass sich tausende von Genexpressionsmustern veränderten. Es handelt sich hierbei um ein gängiges pharmakodynamisches Studiendesign. Sie schreiben dazu:
”(...) thousands of genes were upregulated (Figure 2D) upon LNP injection. With p < 0.05 and FDR<0.05, 9,508 genes and 8,883 genes, respectively, were differentially expressed."
Und weiter heißt es
"(...)the genes associated with monocyte/granulocyte development, recruitment, and function (Cxcl1, Cxcl2, Cxcl5, Cxcl10, Ccl2, Ccl3, Ccl4, Ccl7, Ccl12, Csf2, and Csf3) and inflammation (Il1b and Il6) showed the highest fold increases over the control samples (...)"
Sie fanden in ihrer GSEA-Analyse "(...) large amounts of interleukin-1β (IL-1β), granulocyte-macrophage colony-stimulating factor (GM-CSF), and IL-6, the signature cytokines of inflammatory responses (...)”Abbildung übernommen aus Ndeupen, Sonia, Zhen Qin, Sonya Jacobsen, Aurélie Bouteau, Henri Estanbouli, and Botond Z. Igyártó. “The mRNA-LNP Platform’s Lipid Nanoparticle Component Used in Preclinical Vaccine Studies Is Highly Inflammatory.” iScience 24, no. 12 (December 2021): 103479. https://doi.org/10.1016/j.isci.2021.103479.; Lizenz: CC BY 4.0
In der GSEA-Analyse ist erwähnenswert, dass NF-ϰB um fast das dreifache anstieg, während der TCA Zyklus und PPAR-Signalweg komplett einbrachen und eine ~ dreifache Expressionsreduzierung zu beobachten ist. Auffällig ist die klare Übersteuerung von IL-17 und der Einbruch des AMPK-Signals. Die Pattern Recognition Rezeptoren (PRRs), TLR, Nod-Like und RIG-1-Like deuten auf eine klare systemweite immunologische Abwehrreaktion hin.
Bereits hier deutet sich – durch die beobachtete PPAR- und AMPK-Signatur – eine Dysregulation der MAPK-Signalwege an.82 83 84 85
Tahtinen et al. kommen auch zu ähnlichen Schlussfolgerungen.86 Jedoch lag ihr Fokus auf einem einzelnen Interleukin und dem antagonistischen Rezeptor (IL1/ IL1-ra). Die Autoren selbst schreiben:
”While we did not investigate the source of signal 1 in this context, it could be derived from direct activation of PRRs by the lipids themselves41,42,43, from the release of damage-associated molecular patterns following cell damage caused by LNPs44 or from the direct activation of caspase-11 (ref. 45).”Diese Limitation ist entscheidend: Ihre Beobachtungen erreichen weder eine statistische Signifikanz, noch liefern sie eine plausible Erklärung. Der Fokus auf einzelne Interleukine, wenn eine randomisierte Transfektion stattfindet und unvorhersagbare Zelltypen betroffen sind, ist nüchtern und streng logisch betrachtet nur das Beobachten einzelner Konsequenzen. Dadurch wird jedoch die Ursache eher maskiert, als aufgeklärt.
Forster et al. halten fest, dass lysomale Disruption die NLRP3-Inflammasomkaskade auslöst87 – ein Befund, der ebenfalls auf eine p38-MAPK-Dysregulierung hindeutet.88
Dies deckt sich ebenfalls mit den Beobachtungen von Zelkoski et al., die zeigen, dass leere LNPs NF-kB und IRF via TLR4 aktivieren.89 Interessanterweise zeigt sich in ihrer GSEA-KEGG-Analyse, dass sowohl JUN der JNK-MAPK eine 1.93-fache als auch JAK/STAT eine 1.13-fache Expressionsveränderung aufweisen (log2fold).
Eine weitere bemerkenswerte Arbeit, die die gesamte Realität von modRNA in LNPs verpackt zeigt, ist die von Knabl et al.:90
“Four patients had received the first dose of BNT162b2 about 11 days prior to the onset of COVID-19 symptoms and five patients were unvaccinated. Patients had received dexamethasone treatment. Immune transcriptomes were obtained at days 7-13, 20-32 and 42-60 after first symptomology.”Es ist auffällig, dass die Covid-19-Symptome nach der BNT162b2-Applikation auftraten. In den KEGG-GSEA-Daten dieser vier geimpften Patienten, von denen einer vor Experimentabschluss verstarb, zeigt sich ebenfalls klar eine akute Genexpressionsveränderung für unzählige Gene, wie JAK/STAT3, mTORC, KRAS (MAPK), OAS u.a.; viele davon erreichten eine statistische Signifikanz.
Abbildung übernommen aus Knabl, Ludwig, Hye Kyung Lee, Manuel Wieser, Anna Mur, August Zabernigg, Ludwig Knabl, Simon Rauch, et al. “BNT162b2 Vaccination Enhances Interferon-JAK-STAT-Regulated Antiviral Programs in COVID-19 Patients Infected with the SARS-CoV-2 Beta Variant.” Communications Medicine 2 (February 22, 2022): 17. https://doi.org/10.1038/s43856-022-00083-x.;Lizenz: CC BY 4.0
In diesem Ausschnitt aus den Rohdaten zeigt sich eine massive Dysregulation von JAK/STAT5 mit IL-2 sowie JAK/STAT3 mit IL-6. Es lässt sich nicht rekonstruieren, warum die GSEA-Daten über die verschiedenen Zeiträume inkonsistent mit den Gensets sind und die Anzahl der einzelnen Gene drastisch variiert. Auch lässt sich nicht rekonstruieren, warum die Autoren sich ausgerechnet für die 166 Gene, welche sie final auswerten, entschieden haben.
Weiterhin erwähnenswert ist folgende Beobachtung der Autoren:
”A significant increase of OAS1 and OAS2, but not OAS3, mRNA levels was observed upon vaccination (Fig. 2f). Since the induction of OAS expression in the vaccinated patients exceeded that seen in the vaccinated naïve individuals it is likely that SARS-CoV-2 infection and the development of COVID-19 triggered a booster.”Wenn OAS1 und OAS2 signifikant erhöht waren, liegt die Annahme nahe, dass sie auf dsRNA reagierten.91 92 93
Weiterhin untersuchten die Autoren 10 gesunde Personen und entnahmen nach der ersten Injektion an den Tagen 0, 7, 10 Blut. Auch hier ist der Blick in die Rohdaten entscheidend, da die Autoren nicht erwähnen, dass beispielsweise mTORC1 selbst am 10. Tag noch exprimiert wurde und sich statistisch signifikant verändert hat. Wie bereits unter Abschnitt 1.1 und 4.1.2 diskutiert, sind Vorstellungen von “transient und moderat” im Kontext der Signaltransduktion nicht ausreichend und entsprechen nicht deren Charakteristika.
Es gibt diverse Störfaktoren, die in dieser Studie als Limitation beachtet werden sollten: Komorbidität und Behandlung mit Dexamethason (Glucocortikoid), Heterogenität, Alter, Personenanzahl, Altersbereinigung, Inkonsistenz in den Datenpunkten u.a. Nichtsdestotrotz ist die grundlegende These von Genexpressionsveränderungen und Transfektion als Ausgangsmechanismus auch hier evident gezeigt. Dafür spricht insbesondere das verschobene mTORC1-Signal, da PI3K - wie bereits diskutiert - als Knotenpunkt der PtdIns-Regulation in Betracht gezogen werden sollte.94 Und es kann erneut gefragt werden, ob GPCRs betroffen waren.95
Ein Beispiel für eine Analyse auf single cell Basis liefern Knapp & Bhargava:
“T cells and monocytes showed the greatest number of sexually dimorphic genes. Platelet counts in the study population differed significantly before vaccination (3.6% in females vs 1.8% in males) but not after the second BNT162b2 dose (7.2% vs 7.3%).”Auch sie zeigen, dass sich diverse Genexpressionsmuster nach der Transfektion mit BNT162b2 extrem verschoben haben. Das Appendix, wo die Gene gelistet werden, findet man unter “Supplemental Figure legends and Tables 1-5”. Hier zeigen sich ebenfalls Verschiebungen in der MAPKAP1, MAPK6, MAPK8, MAPK1IP1L, MAPKAPK5. Besonders bemerkenswert ist MAPKAP1, da es als Schlüssel zwischen mTORC2 und MAPK via PI3K verstanden wird. MAPKAP1 ist auch als SIN1 bekannt. Ezine, Lebbe & Dumaz schreiben:96
”Moreover, the numerous interactions of SIN1 inside or outside mTORC2 connect it with other signaling pathways, which are often disrupted in human tumours such as Hippo, WNT, Notch and MAPK.”Hickey et al. lieferten - soweit bekannt ist - die erste Proteomicsanalyse von BNT162b2 und mRNA-1273.97
Abbildung übernommen aus Hickey, Thomas E., Uma Mudunuri, Heidi A. Hempel, Troy J. Kemp, Nancy V. Roche, Keyur Talsania, Brian A. Sellers, James M. Cherry, and Ligia A. Pinto. “Proteomic and Serologic Assessments of Responses to mRNA-1273 and BNT162b2 Vaccines in Human Recipient Sera.” Frontiers in Immunology 15 (January 27, 2025): 1502458. https://doi.org/10.3389/fimmu.2024.1502458.; Lizenz: CC BY 4.0
Die Proteomikdaten zeigen unter anderem, dass einen Monat nach BNT162b2 RAS-Signale durch “Erythropoietin” (Korrektur: Erythropoetin auch bekannt unter EPO) weiterhin “moderat” herunterreguliert waren. Signalkaskaden sollten jedoch im “Normalfall” maximal wenige Tage aktiv oder inaktiv sein, da sie als Schalter dienen.98 Die intrinsische Hydrolyse-Rate von RAS-GTPasen ist mit ca. 0.02 min⁻¹ extrem niedrig. Deshalb wird eine externe Regulation durch GTPase-aktivierende Proteine (GAPs) notwendig. Wenn das RAS-Signal, wie in diesem Fall, nach einem Monat weiterhin herunterreguliert ist, spricht das für eine Dysregulation und entgleiste GAP-Funktionen.
Weiterhin bemerkenswert ist die herunterregulierte Endozytoseaktivität und der “Endosomal Sorting Complex For Transport” (ESCRT). Beide Phänomene sind Anzeichen für einen akut gestörten PtdIns-Zyklus.99 100 101 Auch die erfassten RAB-Signalverschiebungen, sprechen für diese Annahme. Die Autoren belegen darüber hinaus, dass eine akute Immunreaktionsverschiebung stattfand. Ähnliche Verschiebungen waren auch für die weiblichen Teilnehmer mit BNT1262b2 sowie in beiden Gruppen der Studie nach der Impfung mit mRNA-1273 beobachtbar.
Die Limitationen liegen auch hier in einer inkonsistenten Genanalyse der Proteomik, wodurch ein direkter Vergleich erschwert wird und dem Umstand, dass alle Teilnehmer als Baseline bereits 2 Dosen der mRNA-Injektionen erhalten haben.
In einer weiteren aktuellen Proteomicsanalyse von Waters et al. zeigt sich ebenfalls eine gravierende Dysregulation.102
Diese Studie weist jedoch zu viele Limitationen auf, die ihre Aussagekraft einschränken und sie eher als weiteres Indiz denn als direkt belastbaren Beleg für dysregulierte Signaltransduktionskaskaden nach modRNA-Injektionen erscheinen lassen: Es wird nicht ersichtlich, welche Impfstoffe bei den Probanden zu welchen Ergebnissen führten. Postvaccination (PV) versus Post-Infektion (PIS) wurde nicht weiter aufgeteilt in PIS mit Covid-19-Injektion versus PIS ohne Injektion.
4.3.3. “omische” Evidenz Nachwort
Ohne umfangreiche Kohorten ist es in vivo unmöglich, mechanistische Grundlagen für eine Technologie zu etablieren, deren gesamte biologische und mechanische Natur auf direkte intrazellulare Interaktionen baut.
4.4 JAK/STAT und IgG4-Klassenswitch
Wie in Abschnitt 4.3 dargelegt, gibt es sich verdichtende Hinweise, dass STAT3 und STAT5 ebenfalls betroffen sind. Interessanterweise spielt STAT3 auch eine Rolle beim Lipid-Raft-Ratcheting. Unter Lipid Ratcheting wird die Reorganisation der Lipid-Rafts verstanden. Damit lässt sich auch hier erneut der Link zur Kernthese ableiten: LNPs werden ein ganzes Netzwerk aus Signaltransduktionskaskaden massiv stören.103 104 105 Es sind zahlreiche Crosstalks zwischen JAK/STAT- und MAPK-Signalwegen bekannt.106 107 108 Wenn STAT3 und STAT5 antagonistische und synergistische Wirkmechanismen durch einzelne Interaktionen der Zytokine mit den dazugehörigen Rezeptoren erfahren,109 110 und zudem auch noch synergistische und antagonistische Effekte zwischen den einzelnen Zytokinen herrschen, ist bereits hier eine erhebliche Limitation in der Erkennung von Reaktionsmustern gegeben.111 Francis & Porter merken zu dieser Erkenntnis an:
”These major lifestyle decisions require MKN regulation, as they cannot be made simply on the basis of the presence or absence of a single ligand and instead require multiple signals to be evaluated and factored into decision making.” Bereits daraus lässt sich eine Multidimensionalität in dem transmembran lokalsierten (*zum Zytosol ausgerichteten) JAK-/ intrazellularen STAT-Netzwerk erkennen, welche engmaschig reguliert wird.112 Es ist bekannt, dass IL-6 und STAT3 einen Feedback-Loop erzeugen können. Wang et al. zeigen, dass IL-6/IL-6-Rezeptor- Interaktionen mit EGF-Rezeptor-Aktivierung die STAT3-Expression verlängert.113 Diese Verlängerung führt zu einer verstärkten und persistierenden Genexpression. Dieser Loop versetzt beispielsweise Myofibroblasten in einen dauerhaft-inflammatorisch-aktiven Zustand. Dies könnte auch Kardiomyozyten betreffen.114 Darüber hinaus wird bei der Integration von NF-κB in den IL-6/STAT3-Loop erneut deutlich, dass es sich nicht um einfache, isolierte Pfade handelt, sondern um ein mehrdimensionales Regulationsnetzwerk.115 Hu et al. demonstrieren diesen Loop in tumorassoziierten Makrophagenpopulationen.116 Wenn – wie bereits unter 4.3 erwähnt – NF-κB durch einzelne Signalwegsdysregulationen unzureichend aktiviert wird (über welches auch das IL-6- Gen transkribiert117 ) und IL-6 als Feedback-Loop mit STAT3 interagiert, wird dies zwingend immunologische Folgen haben.
Was hat IL-6 mit IgG4-Klassenswitches zu tun?
McLoughlin et al. schreiben:118
"IL-6-mediated signaling is associated with the activation of a number of cellular events important in host defense and chronic disease progression."
Weiterhin betonen sie, dass "IL-6 activation of STAT3 promotes T cell recruitment"
und dass"sIL-6R-mediated trans-signaling promotes T cell expression of antiapoptotic regulators",
wobei die "blockade of sIL-6R signaling in experimental models suppresses the pathogenesis of Crohn's disease and rheumatoid arthritis."Das impliziert, dass STAT3-IL-6-Feedbackloops einen direkten Einfluss auf die B-Zell- und CD4⁺-T-Zell-Maturation nehmen. Dies wird auch von Kastner et al. in vivo bestätigt.(Kastner, 2010 [Fußnote 110]) Sie demonstrierten, dass IL-6 synergistische Effekte mit IL-4 erzeugt und diese die Maturation von FOXP3⁺-Treg-CD4⁺-T-Zellen einleiten. Dabei fanden die Autoren heraus, dass dies zu einer kompletten Destabilisierung der FOXP3⁺-Treg-Zellen führt. Interessanterweise notierten Klemm et al., dass die MAPK-Signalwege direkt in der Überproduktion von IL-6 und dessen Rekrutierung die entscheidende regulatorische Rolle spielen.119 Diese Schlussfolgerung ist deckungsgleich mit Costa-Pereira120 und Nyati et al.121 Wenn IL-6 und IL-4 eine synergistische Wirkung haben und – wie in Teil 1, Abschnitt 2.3 und Abschnitt 4.3 gezeigt – transfizierte T-Zellen eine MAPK-Dysregulation erfahren, ist genau dieser Effekt zu erwarten.122 Auch Deimel et al. kommen zu ähnlichen Schlussfolgerungen.123 Sie zeigen, dass STAT3 direkt über das “Schicksal” von IL4 in CD4⁺-T-Zellen bestimmt.
Liu et al. notieren:124
”Further studies showed that the levels of IL-4, IL-10, and Foxp3 were positively correlated with IgG4/IgG, suggesting that Treg-mediated immune response could promote IgG4 production and IgG4-RD progression.”Zusätzlich wird auch eine Verschiebung zu Th-17 - Zellen diesen Switch begünstigen.125 (Ndeupen, 2021 [Fußnote 81]; Knabl; 2022 [Fußnote 90]) Weiterhin halten De Sainte Marie et al. in ihrem Review fest:
”Studies have clearly shown a role for T cells and T cells cytokines. T follicular helper cells orientated functionally to Th2 cytokine production appear as central players, particularly the activated PD1high subset. CTLs and Treg are thought to participate to profibrotic cytokine production. B cell polyclonal activation is T cell driven, but the therapeutic efficacy of rituximab in the disease suggests that B cells participate to T cell activation and potentially to fibrosis.” Zongfei et al. legen in vitro nahe, dass IL-6 direkt an der Pathogenese von IgG4-Klassenswitches beteiligt ist.126
Dass es nach den modRNA-Injektionen zu einem IgG4-Klassenswitch kommt, ist inzwischen umfangreich dokumentiert. Im Folgenden werden die Studien dazu als Fußnoten gesetzt um diesen Befund zu belegen.127 128 129 130 131 132 133 134 135 136 137 138 139
In diesem Abschnitt wurde ein plausibler Mechanismus nahegelegt, wie diese IgG4-Switches zustande kommen. Wenn die Dynamik der Signaltransduktionsnetzwerke gestört und / oder dynamisch verändert ist, sind JAK/STAT und die MAPK die wahrscheinlichsten Treiber für eine dauerhaft veränderte Immunreaktion, inflammatorische Prozesse und eine für das biologische System zwingend notwendige Überlebenstaktik. IgG4 forciert eine Immuntoleranz.140141 142
4.5 Epigenetik
Dieser Abschnitt gliedert sich in zwei Unterabschnitte, da zwei Perspektiven berücksichtigt werden müssen, die zwar einen vergleichbaren Effekt erzielen, aber unterschiedliche kausale Ursachen haben können. Im ersten Unterabschnitt wird auf die Signaltransduktion mit Fokus auf ERK und IRF Bezug genommen. Es wird untersucht, ob Literatur existiert, die Nanopartikel als potenzielle epigenetische Schalter identifizieren und, ob die bis hierhin mechanistisch kausal und biologisch plausibel nahegelegten und auf Basis real erhobener Transkriptomik- und Proteomik-Daten belegten Annahmen der Signalverschiebung Einfluss auf epigenetische Regularien haben.
Im zweiten Unterabschnitt werden Realdaten von BNT162b2 und mRNA-1273 zur Untersuchung herangezogen. Die Trennung erfolgt rational, da sowohl die Kombination als auch die alleinigen LNPs in Betracht gezogen werden müssen. Dies ergibt sich aus den folgenden Gründen: Es kann nicht abschließend geklärt werden, wie viel mRNA in einer Injektion ist, da dies von der Charge und genutzten Dosis einer Flasche abhängig ist – wie bereits unter Teil 1, Abschnitt 2.5 erläutert.143 144 145 146 Des Weiteren ist entscheidend, welcher Prozess (1, 2, 3) zur Herstellung Anwendung fand,147 da die aktuellen Chargen zusätzliche DNA-Kontaminationen enthalten und der Puffer von BNT162b2 zwischenzeitlich ausgetauscht wurde. 148 149 150 151 152
Vor dem Hintergrund der jüngst entstandenen und kontrovers geführten Debatte um DNA-Verunreinigungen in der modRNA-Transfektionstechnologie, erscheint eine Würdigung der Arbeit von Kaiser et al. geboten.153 Sie weist als einzige signifikant geringere Messwerte der DNA-Menge auf. Daher muss die Methodik diskutiert werden.
Die Autoren berücksichtigten nicht die EtOH-Prinzipien, wie es beispielsweise Kurinomaru et al. 2017 demonstrierten.154 Dadurch wird fragmentierte DNA <200 Basenpaare kaum erfasst, was eine systematische Unterschätzung zur Folge hat. Dazu kommen Detektierungsschwierigkeiten bei RNA-Modifikationen.155 Darüber hinaus wurde nicht die Schwierigkeit des RNase-Verdaus bei 100% m1Ψ-modifizierter RNA berücksichtigt, wie in Teil 1, Abschnitt 3 expansiv diskutiert wurde. Dies wird erneut das Detektionslevel senken. Zudem wurde bei der Nutzung von Phenol-Chloroform und Nuclease die Ethanol-Fällungslimitation bei kleinen DNA-Fragmenten nicht berücksichtigt. Da der Farbstoff nicht explizit erwähnt wird, ist von Pico-Green bei der RNase auszugehen. Dies führt ebenfalls zu einer Unterschätzung.156 Die Frage nach dem Interkalator wurde in der Arbeit nicht beantwortet. Zusätzlich ist zu berücksichtigen, dass das genutzte Assay (Qubit), weder Fragmente, ssDNA noch mögliche RNA-DNA-Hybride zuverlässig detektieren kann. Noch eine weitere Limitation besteht darin, dass nicht sicher gesagt werden kann, wie viel RNA/DNA überhaupt verkapselt wurde.157
Zusammenfassend lässt sich festhalten: Wenn die modRNA intakt bleibt, kann sie immer noch Fluoreszenzfarbstoffe wie die im Qubit-Assay binden, die eigentlich DNA nachweisen sollen. Dadurch entsteht ein falsches Signal, das den scheinbaren DNA-Wert aufbläht. Kaiser et al. gehen jedoch davon aus, dass alle RNA verschwunden ist und sie nur „DNA“ messen werden. Dies verschleiert durch jede intakte modRNA die tatsächliche DNA-Fragment-Menge, was zu einer Unterschätzung der tatsächlichen DNA-Kontamination führt. Jedes einzelne Fragment sollte ein eigenes Signal erzeugen können, um eine tatsächliche Quantifikation zu ermöglichen. Der gegebene Hinweis auf Nukleoside und Verhältnisse gilt für LC-MS, wo intakte modRNA die RNA-Nukleosiddetektion reduziert und das RNA:DNA-Verhältnis verzerrt. Bei Qubit geht es um Fluoreszenzbindung, nicht um Nukleoside. Die Autoren ziehen damit einen nicht haltbaren Zirkelschluss.
Somit ist es um so wichtiger, die angesprochenen zwei Perspektiven einzunehmen, da auch Integrationsevents und/ oder modRNA-Interaktionen zusätzliche epigenetische Effekte auslösen können. Dies kann bei der durchschnittlich gemessenen Menge nicht ausgeschlossen werden, ohne finale Untersuchungen auf genomischer Ebene in vivo. Die Quantifizierung von Nukleosiden allein kann den Nukleotidkontext, die strangspezifische Integration oder den Phosphodiester-Inkorporationsstatus nicht aufklären und reicht daher nicht aus, um funktionelle epigenomische Konsequenzen abzuleiten.
4.5.1 Signaltransduktion, Epigenetik, leere LNPs
Wie bis hierhin gezeigt, verschieben die LNPs die gesamte Signaltransduktion. Insbesondere werden dabei verschiedene Wege der MAPKs (JNK, ERK, P38, etc.) sowie Mechanistic Target of Rapamycin1/2 (mTORC1/2) verändert. Demnach sollte untersucht werden, wie solche Veränderungen auch die epigenetische Regulierung der Histone und Chromatin beeinflussen können und ob es Hinweise im Kontext der LNPs auf solche Veränderungen gibt.
Wei et al. demonstrieren in 2023:158
“It has been reported that the ERK pathway is involved in the regulation of DNA methylation by histone deacetylase inhibitors and PP2Ac [22,23]. To confirm this, we treated F9 cells with the MEK/ERK inhibitor PD0325901 (1 μM, 12 h) (hereafter referred to as PD) and assessed genome-wide 5 mC levels by methylation staining. Our results showed that inhibition of ERK activity increased genome-wide 5 mC levels (Figure 2a,b).”Daraus ergibt sich bereits, dass ERK direkt in die DNA-Methylierungsregulation involviert sein muss. Cohen-Armon et al. stellten bereits 2007 fest, dass ERK2 eine direkte Rolle in der Histon-Acetylierung spielt.159 Sie fanden heraus, dass dies unabhängig von DNA-aktivierter Poly(ADP-Ribose)-Polymerase 1 (PARP1) geschieht und schrieben:
“In cortical neurons treated with nerve growth factors and in stimulated cardiomyocytes, PARP-1 activation enhanced ERK-induced Elk1-phosphorylation, core histone acetylation, and transcription of the Elk1-target gene c-fos. These findings constitute evidence for PARP-1 activity within the ERK signal-transduction pathway.”Navandar et al. kommen in ihrer computergestützten Analyse zu dem Schluss, dass ERK “eine Reihe charakteristischer Transkriptionsfaktormotive” reguliert, welche in distalen regulatorischen Regionen angereichert sind.160 ERK gestaltet dabei ihre epigenetische Struktur um.
Li et al. demonstrieren in Krebszellen, welche Konsequenz eine gestörte MEK-ERK-Signalgebung haben kann.161 Sie zeigen, dass es zu einer dauerhaft aktivierten Schleife in der UHRF1-DNMT1-Achse und einer anhaltenden Überexpression kommt. Bereits daraus ergibt sich die notwendige Frage, wie sich die Dynamiken nach modRNA-Applikation der MAPK-Signale verschoben haben.
Pradhan et al. zeigen zudem, dass auch die ERK/Snail-Achse entscheidend für die Epigenomregulation ist.162
Wie ebenfalls in 4.3 durch die “omischen” Daten gezeigt, wird nicht nur die MAPK-Signalgebung betroffen sein, sondern auch JAK/STAT3 und STAT5 und IRF.
Sobhiafshar et al. zeigen,163
dass IRF4 die “(...) DNA methylation (DNMT1, DNMT3B, UHRF1) and histone H3K27 methylation (EZH2)” reguliert.
Weiterhin halten Sie fest “(...) that IRF4 controls the expression of tumour suppressor genes known to be silenced by these epigenetic modifications, for instance cyclin‐dependent kinase inhibitors CDKN1A and CDKN1B, the PI3–AKT pathway regulator PTEN, and primary cilium components.” Eine interessante Beobachtung, die Mishra et al. in ihrer Arbeit beschreiben, ist, dass die epigenetischen Daten der Typ-1-dendritischen Zellen eine bevorzugte Reaktivität der TLR9 gegenüber den TLR3 zeigen, die über den Nuclear Receptor Co-repressor 1 (NCOR1) mittels verschiedener IRF3-Signatur reguliert wird. 164
Sie schreiben:
“We found IRF3, an important well-known SRTF under TLR3 activation, showed decreased phosphorylation as well as binding at key enhancer regions of important antiviral genes. Furthermore, the decrease in transcription of antiviral genes in pIC activated NCoR1 KD cells translated to an expected increase in Sendai Virus infection (SeV) load and decreased cytotoxic potential of CD8+ T-cells.”Darüber hinaus fanden Hu et al., dass IRF3 an ARID1A binden kann und dadurch direkt die Methyltransferase NSD2, welche H3K4 und H3K36 methyliert, mit regulieren wird.165
Aus den Beobachtungen von Mishra et al.[Fußnote 164] und Hu et al., 2021 [Fußnote 165] ergibt sich bereits eine nahelegende Überlegung: Da IRF3 nicht nur durch epigenetische Kontrolle die TLR3 und TLR9 reguliert, sondern selber auch epigenetisch-regulatorische Funktionen übernimmt, würde jeder Versuch die TLRs zu unterdrücken – oder durch Transfektion und Internalisierung – umfangreiche und/oder langfristige epigenetische Konsequenzen haben (siehe Teil 1, Abschnitt, 3.3.2). Wie von Lonez et al. (Teil 1, [Fußnote 91]) und Chaudhary et al. (Teil 1, [Fußnote 95]) gezeigt, können Liposome bzw. LNPs auch TLR4 binden. Es ist wichtig, festzuhalten, dass TLR3 und TLR4 nicht isoliert, sondern in Netzwerkdynamiken interagieren. 166 167 168 169 170 171 Eine ähnliche Regulationsdynamik lässt sich auch für TLR7 und 8 identifizieren.172 173 174 Wenn die TLR4 betroffen sind, ist zu erwarten, dass auch TLR3, 7 und 8 regulatorische Verschiebungen in ihrer Dynamik und Reaktivität (beispielsweise durch Supression) erfahren.
Zusätzlich zeigen Hu et al., dass IRF7 in Thymusepithelzellen epigenetische Regulationsfunktionen übernimmt.175 Sie schreiben:
”The absence of polycomb repressive complex 2 (PRC2) activity that catalyzes the repressive H3K27me3 epigenetic marks compromises mTECs development and TCR diversity of thymocytes through the transcription factor Irf7 and Ascl1 [13].”Die Konsequenzen von epigenetischen Veränderungen sind – Stand der Veröffentlichung dieser Hypothesenschrift – unzureichend verstanden.176 Breton et al. merken an:177
”Taken together, these results suggest that in utero exposures may program male fetal germ cells and/or Sertoli and Leydig cells, which are laid down in the fetal testes and are responsible for future sperm production31,32,33. The consequences of such effects on future progeny are largely unknown.”Welche Konsequenzen könnten aus einem Kontrollverlust epigenetischer Regulation resultieren? Langille et al. zeigen beispielsweise, “dass der Verlust der epigenetischen Regulation die Integrität der Zelllinie stört, eine abnorme Alveogenese induziert und Brustkrebs fördert”.178
Erneut sei erwähnt (Teil 1, Abschnitt 2), dass weder die Rate der Transfektionsevents, noch die Rate der Translationsevents, noch die genauen Verhältnisse, welche Zellen in welchem Umfang betroffen sind, ausreichend charakterisiert sind.
Lee et al. schreiben:
“In addition, we found that a TLR7/8 agonist, 3M-052, could induce long-lasting transcriptomic and epigenomic signatures in myeloid cells in mice. Specifically, myeloid cells, in particular monocytes, were found to retain increased gene expression and accessibility of interferon-stimulated genes (e.g. Cxcl10, Ifit2, Ddx58) and IRF and STAT loci accessibility, with these changes persisting up till 28 days after immunization [20]. We also found that a similar monocyte population with increased IRF/STAT and reduced AP-1 transcription factor gene expression was observed post-boost following BNT162b2 mRNA vaccination in human [57]. These findings suggest the induction of a distinct monocyte subset that could potentially be epigenetically reprogrammed with a heightened antiviral response following vaccination (Figure 3).”Monozyten können sowohl Vorläuferzellen von dendritischen Zellen als auch von Makrophagen sein.179 180
Musolino et al. diskutierten 2022 bereits unter allgemeinen Aspekten die epigenetischen Konsequenzen bei dem Einsatz von Nanopartikeln. 181
Connors et al. kommen zu einem für sie überraschenden Befund.182 In 2023 stellen sie fest, dass ihre leere LNP-Formulierung bereits aus Monozyten entstandene dendritische Zellen zur Maturation treibt. Weiterhin beobachteten sie daneben pro-TFH Zytokine inklusive IL-21 und IL-6. Sie weisen dies in vivo in sowohl jungen ( 18–29 Jahren) als auch in älteren (65–74 Jahren) Erwachsenen nach. Sie schreiben dazu:
“We also demonstrate that there is an age-specific difference between important innate immune cytokines and chemokines and show that eLNP initiate TGF-β production as a potential mechanism of human TFH cell differentiation19. Mechanistically, the capacity of eLNP to elicit robust induction of innate and adaptive responses shows evidence of being dependent on pathways that converge upon TBK-1 (phosphorylation of TBK-1), and/or IRF-7 though differences between younger and older adults was less noticeable. We also show that the addition of eLNP acts as a stimulator of phagocytosis. This study sheds light on the mechanism of the immunomodulatory component of the recent SARS-CoV-2 vaccines (...)”Zhang et al. weisen in 2023 auf unerwünschte epigenetische Rückprogrammierung durch Nanopartikel hin.183 Auch sanei et al. (vermutlich Sanei?) kommen 2024 zu ähnlichen Schlussfolgerungen.184
Pongma et al. untersuchten die transkriptomischen und epigenetischen Konsequenzen in leber-assoziierten F4/80 dendritischen Zellen in vivo in 6 Mäusen. 185
Sie berichten:
“On Day 7, we found a total of 554 genes that were considered differentially expressed, where more genes were downregulated. The number of upregulated genes (19 genes) was relatively small, possibly because the time of cell harvesting from the livers was past the acute phase.” (...)
Und stellen fest, dass die durchgeführte Gene Ontology "GO enrichment analysis identified the enrichment of pro-inflammatory cytokines (IL-6/JAK/STAT3, TNFα/NF-κB pathways) and cytokines related to cell-mediated immune response (interferon gamma response, interferon alpha response) in macrophages from the mRNA/LNP group."
Darüber hinaus berichten die Autoren, dass "several chemokines were upregulated (Cxcl1, Cxcl2, Cxcl13) (...)"
und sie in den "CUT&RUN" Sequenzierungsdaten "(...) two main clusters of cis-regulatory loci in which H3K4me3 was enriched at high and intermediate levels by mRNA/LNPs." fanden.Da – wie unter Teil 1, Abschnitt 2 diskutiert – Transfektion nicht mit mRNA-Expression gleichzusetzen ist, lässt sich nicht abschließend klären, was die genutzte RNA bewirkte und was von den Effekten der LNPs ausgelöst wurde. Doch wie bis hierher bereits belegt, werden die LNPs höchstwahrscheinlich einen epigenetischen Effekt bewirkt haben.
Mokhtari et al. demonstrieren, dass sie die Epigenetik mittels Transkriptionsrepressor auf der Grundlage von Zinkfingern in ihrer LNP-Formulierung in vivo veränderten.186 Wobei auch hier nicht eindeutig ableitbar ist, ob vorrangig ihr LNP-eingekapselter Transkriptionsfaktor diesen Effekt hervorrief oder die LNPs als solche.
Darüber hinaus zeigten Quin et al. in vivo, dass bereits die gesamte TLR-Reaktivität von der Exposition mit leeren LNPs betroffen ist und diese auf vererbbare Weise verändert wird.187 Dies deckt sich mit den vorgeschlagenen Mechanismen, dass ERK und IRF eine epigenetische Regulation annehmen können und vermutlich involviert sind. Quin et al. nutzten zur Verifizierung WT C57BL/6J- und Balb/c-Mäuse, wodurch sie ein bloßes Artefakt exkludierten. Diese Ergebnisse legen eine keimbahnvermittelte, generationsübergreifende Weitergabe der Umprogrammierung des Immunsystems nahe.
Es ist demnach nicht auszuschließen, dass bereits leere LNPs unvorhersehbare epigenetische Veränderungen in den sogenannten “Zielzellen” (Teil 1, Abschnitt 2.3) auslösen könnten. Und auch hier greifen erneut die Limitationen, dass zu wenige Daten tatsächlich erhoben und gezielt diese Möglichkeiten adressiert wurden und zudem nicht immer zwischen LNPs, modRNA und LNP/modRNA-Effekten unterschieden wurde.
4.5.2 Epigenetische Änderungen unter Betrachtung der Realweltdaten
Hellgren et al. zeigen Veränderungen in diversen Chemokinachsen, TNF-α und IL-6.188 Da es sich um keine longitudinale Studie handelt und eine nachvollziehbar definierte Kontrollgruppen-Baseline fehlt, ist es lediglich ein Indiz, welches dennoch erwähnt gehört. Die Autoren kommen zu interessanten Schlussfolgerungen.
Yamaguchi et al. liefern eine epigenetische Analyse über 49 Tage, in der sie die Monozytenpopulation beobachteten.189 Sie schreiben dazu:
”In this study, we reveal that the BNT162b2 mRNA+6
vaccine induces epigenetic changes in monocytes after the first vaccination, which result in an enhanced type I IFN response at the time of the second vaccination. Importantly, however, we show that this effect is transient and diminishes 28 days after the second vaccination.”Diese Studie hat einige Limitationen. Die Autoren verfolgen ausschließlich Monozyten. Es ist bekannt, dass Monozyten eine maximale Lebensspanne von drei Tagen haben.190 191 Die Autoren adressieren diesen Fakt nicht ausreichend. Sie verfolgen auch nicht die dynamische Regulation von Progenitorzellen.192 193 194 Diese Dynamiken haben systemweite Konsequenzen.195 196 Zudem differenzieren sie nicht, um welche Monozyten es sich handelt, sprich ob es kurzlebige, im Knochenmark produzierte Monozyten waren oder langlebige EMPs (Leber). Und was bedeutet “diminishes”? War der Effekt rückläufig, persistent, reversibel oder irreversibel? Die Autoren verfolgen auch nicht das “Schicksal” dieser Monozytenpopulation. Wurden sie in dendritische Zellen und/oder Makrophagen differenziert? (León, 2005 [Fußnote 179]; Italiani, 2014 [Fußnote 180]) Wie wirkte sich der Effekt auf die Immunmodulation der restlichen Immunzellen aus? Es bleiben zu viele Fragen offen, wodurch die Worte “transient” und “diminishes” nicht plausibel vertretbar erscheinen.
Weiter schreiben die Autoren:
“Blood samples were collected prior to the first vaccination (D0) and on D1, D10, D20, D22, D31, and D49 after the initial vaccination (Figure 1A).”Diese Zeitpunkte lassen keine ausreichende Auflösung hinsichtlich des Verhältnisses zwischen Progenitorzellen und Monozyten zu, und es bleiben zentrale Aspekte der zellulären Reprogrammierung sowie Immunmodulation unklar.
Eine Antwort auf diese Fragen lieferten jüngst Simonis et al.197 Sie halten fest, dass diese epigenetisch veränderte Subpopulation der Monozyten zu dauerhaft epigenetisch veränderten Makrophagenpopulationen führt. Simonis et al. fassen zusammen:
”•SARS-CoV-2 mRNA vaccination triggers H3K27ac changes in macrophages, indicating immune memory.
•Two vaccinations induce lasting epigenetic marks at promoter regions.
•H3K27ac is linked to G-quadruplex DNA structures in nucleosome-depleted regions."
Weiterhin schreiben sie, "(...) that several cytokines and chemokines (TNF-α, IL-36, CCL3, CCL20, CCL4, CXCL1) were significantly elevated in supernatants of macrophages at t2 compared to those isolated from unvaccinated individuals (...)”,
während "CCL7, CXCL13, and CXCL10 were not affected by SP stimulation in macrophages at t0 and t2 (...)".
Eine weitere wichtige Anmerkung, die die Autoren zu den G-Quadruplexinteraktionen treffen lautet:
"Notably, epigenetic remodeling extends beyond chromatin modifications and has also been linked to alternative DNA secondary structures. Human DNA can adopt G-quadruplex (G4) structures in repetitive elements, such as telomeres, as well as in nucleosome-depleted regions, particularly at highly transcribed genes (Biffi et al, 2013; Hansel-Hertsch et al, 2016; Hansel-Hertsch et al, 2020). G4 DNA has been associated with epigenetic remodeling and the regulation of innate immune responses (Guilbaud et al, 2017; Makowski et al, 2018; Miglietta et al, 2021)."Die Beobachtung, dass maßgebliche Chemokine wie CXCL13, CCL7 und CXCL10 nicht signifikant hochreguliert waren, während gleichzeitig proinflammatorische Mediatoren wie TNF-α, IL-36, CCL3, CCL20, CCL4, CXCL1 stark anstiegen, deutet auf eine funktionelle Umlenkung der Immunantwort und eine tiefgreifende homöostatische Zellveränderung hin.198 199 200 201 202 CXCL13 beispielsweise ist einer der dominantesten B-Zell-Attraktoren,203 204 205 206 während CCL7 (auch als MCP-3 bekannt) für die Mobilisierung von Natürlichen Killerzellen (NK), aktivierte T-Zellen und Monozyten bekannt und essenziell ist.207 208 209 210 Die Schlussfolgerungen der Autoren sind zwar konsequent, aber nicht zwingend.
Es sollte erwogen werden, ob nicht eine massive, epigenetisch verursachte Dysregulation der Makrophagen vorliegt. Die Autoren beantworten nicht abschließend, ob es sich um “klassische” (kanonische) Makrophagen handelt. Die Autoren klären auch nicht abschließend, welche G-Quadruplexe sie tatsächlich observierten und assoziieren lediglich, dass es sich ausschließlich um eine Immunmodulation handelt. Die Frage bleibt offen, welche und wie viele G-Quadruplexe vorlagen.
Eine weitere wichtige Beobachtung machten Pang et al. in ihrer longitudinalen Studie.211 Sie untersuchten periphere Blutproben von 21 Teilnehmern im Alter von 18 bis 73 Jahren, welche eine PCR-positive COVID-19-Diagnose hatten. Darüber hinaus untersuchten sie 36 Probanden vor und nach Pfizer BNT162b2- und Moderna mRNA-1273-Applikation.
Sie schreiben:
“Among the top Δβ-value methylation changes comparing pre- and post-COVID-19 time points, we observed two differentially methylated loci (cg02037503 and cg23712970) in a gene promoter transcription start site regulatory region of the apoptotic chromatin condensation inducer 1 (ACIN1) gene Supplementary Figure S2 that decreased in DNA methylation following COVID-19 by approximately 15% for both CpG sites (Figures 1D,E).”
Und weiter heißt es:
"Complementary longitudinal epigenetic clock analyses of 36 participants prior to and following Pfizer and Moderna mRNA-based COVID-19 vaccination revealed that vaccination significantly reduced principal component-based Horvath epigenetic clock estimates in people over 50 by an average of 3.91 years for those who received Moderna."Dies deutet ebenfalls auf eine Verschiebung und/oder einen Reset der beobachteten Zellen hin, da das Wort ‚jung‘ im Kontext von epigenetischen Clock-Messungen nicht mit ‚verjüngend‘ oder ‚gesund‘ gleichzusetzen ist. Hier ist als Limitation auch der Cutoff-Effekt – wie bei Krawcyk et al. (Teil 1, Abschnitt 3.1, [Fußnote 142], Teil 1 [Fußnote 143]) diskutiert – zu erwähnen. Es können sich an den Grenzen der p-Werte – durch Auswahl bedingte – Fehler einschleichen, die eine Signifikanz oder Nichtsignifikanz verschieben.
Gianotta et al. postulieren 2023 eine interessante und plausible Hypothese, bei der sie die epigenetische Zellprägung bereits als Ausgangspunkt der Reaktionen auf die modRNA-Injektionen betrachten.212
Sie gehen davon aus, dass die modRNA-Injektionen in “(...) from a limited pro-inflammatory condition to conditions of increasing expression of pro-inflammatory cytokines that can culminate in multisystem hyperinflammatory syndromes (...)” münden können.
Dabei wird angenommen, dass “(...) through epigenetic modifications, could have made the innate immune system of each individual tolerant or reactive to subsequent immune stimulations (...)”Daraus ergibt sich jedoch erneut die Frage, wie und in welchem Umfang die bisher gelieferten Beweise für epigenetische Verschiebungen den Populationsdurchschnitt betreffen werden. Darüber hinaus ist nicht klar, mit welchen Zeitspannen für pathomechanistische Entwicklungen, wie Autoimmunerkrankungen, Krebs und neurodegenerativen Pathomechanismen gerechnet werden sollte. Zudem ergeben sich auf diesem molekularen Level auch systematische epidemiologische Erfassungsschwierigkeiten, da die Vielzahl von möglichen Mustern keine feste Mustererkennung zulässt, wenn nicht eine breite Reaktionsmöglichkeit in Betracht gezogen wird.
Hirota et al. beobachten ebenfalls epigenetische Veränderungen nach BNT162b2 in den T-Zellen:213
“We observed that AP-1 expression in PBMCs rapidly decreased upon ex vivo stimulation with BNT162b2 mRNA, which is consistent with a recent report that expression of AP-1 genes such as FOS and ATF3 was diminished in CD14+ monocytes by BNT162b2 vaccination28. Interestingly, the AS3-adjuvanted H5N1 pre-pandemic influenza vaccine also induces a decrease of AP-1 gene expression in monocytes through epigenetic silencing, which is maintained for at least 28 days41. Vaccine-induced alteration of AP-1 expression levels might explain why we detected association between vaccine-induced T-cell responses and expression of FOS and ATF3 at baseline (T1), but not after vaccination (T4 or T5).”Die Autoren haben die T-Zell-Antworten in peripheren mononukleären Blutzellen (PBMCs) gemessen. Sie verwenden dazu ELIspot und ordnen sie anschließend in Beziehung zu Genexpressionsprofilen — darunter FOS und ATF3.
Die Autoren stellen eine weitere plausible Überlegung dazu an:
”These epigenetic mechanisms in monocytes or hematopoietic stem cells might be involved in changes in the frequency of monocytes that we observed at T5 after BNT162b2-induced inflammatory responses.”Daraus ergibt sich erneut die Frage nach den Progenitor-/Monozytzellverhältnissen und wo (lokal) diese epigenetische Prägung stattfand. Es ist plausibel – siehe auch Teil 1, Abschnitt 2.3 – anzunehmen, dass einige Knochenmarks-Stammzellen transfiziert wurden.
Kim et al. kartieren in ihrer longitudinalen Studie die epigenetische Landschaft in Immunzellen (CD14 Monozyten, zytotoxische NK-Zellen, CD8⁺-T-Zellen, Treg, CD4⁺ Gedächtniszellen und CD4⁺ naiven Zellen) nach BNT162b2-Applikation in Patienten, die eine Myokarditis erfuhren.214 Sie halten fest, dass IL-1, IL-6, IL-17 und IL-21 hochreguliert sind während IFN I und IFN III eine massive Runterregulierung aufweisen. Sie stellen zusätzlich fest, dass RUNX2/3 und die IFN-Signalgebung eine reziproke Expression erfahren. Eine weitere wichtige Erkenntnis, die Kim et al. festhalten – welche im Einklang mit der bisherigen Argumentation steht – ist, dass “IFN regulatory factors (IRFs) showed the highest degree of downregulation in the myocarditis state”.
Mele et al. demonstrieren eine persistente Aktivierungssteigerung der NK-Zellen und merken an:215
“Interestingly, epigenetic modifications have been observed in CIML NK cells, and similar modifications have been reported in the expanded NKG2C+ mNK cell subset in human CMV-infected subjects,8 suggesting that signaling through NKG2C mediated by CMV peptide-loaded HLA-E, with or without additional cytokine stimuli, also results in sustained functional modification of NK cells.”
Sollte sich, wie von Goel et al. demonstriert, ein Einbruch der CD8⁺-T-Zellen nach 6 Monaten bestätigen, müssen weitere Fragen geklärt werden, welche bisher unzureichend adressiert wurden:216
Abbildung übernommen aus Goel, Rishi R., Mark M. Painter, Sokratis A. Apostolidis, Divij Mathew, Wenzhao Meng, Aaron M. Rosenfeld, Kendall A. Lundgreen, et al. “mRNA Vaccines Induce Durable Immune Memory to SARS-CoV-2 and Variants of Concern.” Science 374, no. 6572 (December 3, 2021): abm0829. https://doi.org/10.1126/science.abm0829.; Lizenz: CC BY 4.0
Wurde auch die epigenetische Landschaft der NK-Zellen umstrukturiert und wie würde sich das auf die CD8⁺-T-Zellen auswirken?
Ivanova et al. halten dazu fest:217
”Based on the NK cell receptor phenotype we observed NKp46 and CD94-NKG2A cognate ligands were measured. Activating NKp46 (NCR1-ligand) ligand increased and NKG2A ligand Qa-1b expression was reduced on CD8+ T cells.”Eine weitere tiefe Analyse liefern Erdogan et al.218 Sie zeigen bei neonatalen männlichen Ratten autismusähnliche Verhaltensveränderungen, nachdem die weiblichen Ratten pränatal mit BNT162b2 behandelt wurden.
Die Autoren schreiben:
“Molecular techniques were applied to quantify WNT and m-TOR gene expressions, BDNF levels, and specific cytokines in brain tissue samples. The findings were then contextualized within the extant literature to identify potential mechanisms. Our findings reveal that the mRNA BNT162b2 vaccine significantly alters WNT gene expression and BDNF levels in both male and female rats, suggesting a profound impact on key neurodevelopmental pathways.”Diese Ergebnisse korrespondieren mit den speziell in Abschnitt 4.5.1 diskutierten Signaltransduktionsdysregulationen und deren Einfluss auf das Epigenom. Zwischen WNT- und mTOR-Signalwegen bestehen untereinander verschiedene Crosstalkmechanismen. Diese wiederum sind ebenfalls mit der MAPK verknüpft. Insgesamt zeigt sich, dass insbesondere der kanonische ERK-Arm der MAPK auch für die WNT-Regulierung als Knotenpunkt zu agieren scheint. 219 220 221 222 223
Weiterhin halten Erdogan et al. fest:
“Both genetic and hormonal elements play roles in these sex differences. Several studies have pinpointed sex-specific genetic variants linked to ASD, with particular genes on the X and Y chromosomes potentially causing a higher male prevalence [34]. The brain also exhibits sex-specific gene expression patterns and epigenetic changes, underlining the significance of sex chromosomes in brain function and growth.”Daraus lässt sich ableiten, dass es scheinbar geschlechtsspezifische epigenetische Veränderungen in den neonatalen Ratten gab.
Jin et al. entnahmen 260 Blutproben von 102 Probanden.224 Die Autoren identifizieren:
”A total of 94573 high-confidence A-to-I RNA editing sites were observed in 5723 genes in all recipients’ blood (Figure 1A). The editing levels of these sites ranged from 1% to 100%, and were widely observed across all chromosomes. Among these RNA editing sites, 92440 (97.7%) were shared by two groups, 158 (0.2%) and 1975 (2.1%) sites were uniquely detected in control (V0) and COVID-19 vaccines groups, respectively (Figure 1B). As for edited genes, 5623 (98.2%) were shared, 15 (0.3%) and 85 (1.5%) genes were uniquely edited in V0 and COVID-19 vaccines groups, respectively (Figure 1C). The functional categories of these RNA editing sites included 73227 (77.4%) intronic variants, 13351 (14.1%) 3’ -untranslated region (3’ UTR) variants, and 1228 (1.3%) missense variants (Figure 1D). Sorts intolerant from tolerant (SIFT) analysis predicted that 361 (29.4%) of these missense variants might have a potential functional impact on the encoded protein (Figure 1E). 61.6% of these edited sites overlapped with Alu repetitive elements (Figure 1F).”Sie kommen zu dem Schluss, dass wahrscheinlich Adenosin-zu-Inosin (A-zu-I)-RNA-Bearbeitung durch Adenosindeaminase, die auf RNA wirkt (ADARs) einer der plausibelsten Mechanismen ist, der diese epigenetischen Veränderungen posttranskriptional auslöst und diese Veränderungen dosisabhängig sind. Demnach sind die von den Autoren beschriebenen epigenetischen Veränderungen auf epitranskriptomische Modifikationen zurückzuführen.
Was ist zu erwarten , wenn nachhaltige epigenetische Umfunktionalisierungen der Immunzellen stattfinden?
Föhse et al.225 halten fest:
“In contrast, the production of IFN-α after stimulation with SARS-CoV-2, TLR3 ligand poly I:C, and TLR7/8 ligand R848 decreased after vaccination. Altogether, we observed that administration of the BNT162b2 vaccine modulated innate immune responses up to one year after the initial vaccination.”Die Autoren beobachten ein deutlich und dauerhaft verändertes Verhalten der Zytokinexpressionsmuster und eine Reduktion der TLR-Reaktivität. Daher scheint das Wort “modulated” im Lichte der Daten nicht zutreffend zu sein. Es wird eine deutliche Immunantwortverschiebung bzw. Reprogrammierung gezeigt.
Noé et al. zeigen ähnliche Ergebnisse bei Kindern nach 182 Tagen (V2+182).226 Bei V2+182 Tage zeigte sich, dass bei verschiedenen Stimuli eine völlige Verschiebung des Zytokin-Profils der Kinder entstand. Darunter war beispielsweise bei TLR-Stimulation mittels Hepatitis B Antigen eine Abnahme von IL-6, IL-15, TNF-α, GM-CSF, PDGF-BB, VEGF, FGF-basic, IL-10, IFN-γ, IL-2, IL-4, IL-5, IL-9, IL-13 und Eotaxin zu beobachten (Vergleich mit V1). Es handelt sich um eine longitudinale Studie. Die Schlussfolgerungen der Autoren sind nicht frei von Interpretationsspielraum.
Einen weiteren Beweis auf transgenerationale Effekte lieferten Estep et al.227 Die Autoren entnahmen Nabelschnurblut und observierten die CD34+ hämatopoetischen Progenitorzellen. Sie vermerken:
"Striking changes were observed in the CD34+ cell fraction: the total numbers of CD34+ cells drastically reduced 4-fold in the vaccinated donor group, in which this change was correlated with the induction of apoptosis in CD34+ cells, likely mediated by IFN-γ-related pathways as determined by a total transcriptome assay."Sie begründen die Deletion mit dem erhöhten IFN-γ-Transkriptom und Apoptose. Dies erscheint jedoch nicht im Einklang mit den beiden Observationen von Gur et al. und Rafaeli et al. Gur et al. beschreiben, dass TNF-α für die Deletion (Apoptose/ Zelltod) von CD34+ verantwortlich erscheint.228 Während laut Rafaeli et al. IFN-γ für die Deletion von CD8+-T-Zellen dominierend ist.229 Einen weiteren Hinweis liefern Zeng et al.:230
“Hematopoietic effects of interferon-γ (IFN-γ) may be responsible for certain aspects of the pathology seen in bone marrow failure syndromes, including aplastic anemia (AA), paroxysmal nocturnal hemoglobinuria (PNH), and some forms of myelodysplasia (MDS). Overexpression of and hematopoietic inhibition by IFN-γ has been observed in all of these conditions. In vitro, IFN-γ exhibits strong inhibitory effects on hematopoietic progenitor and stem cells.”Die Erkenntnisse von Zeng et al. decken sich auch mit der Zusammenfassung von Morales-Mantilla & King.231 Die Autoren halten in ihrer Untersuchung der Lektüre fest, dass
“(...) these studies suggest that IFN-γ only triggers HSC apoptosis in the presence of other stress stimuli.”Sollte IFN-γ, wie von Estep et al. vorgeschlagen, tatsächlich eine zentrale Rolle bei der beobachteten Zellreduktion spielen, erscheint es plausibler, dass seine Wirkung über reine Apoptose hinausgeht und auch regulatorische Effekte auf Differenzierung und klonale Expansion ausübt – beispielsweise als proinflammatorischer Inhibitor der Reifung. Es ist bekannt, dass IFN-γ das Gleichgewicht zwischen T-Zell-Apoptose und -Überleben stärker beeinflusst als direkt die hämatopoetischen Stammzellen. Darüber hinaus könnte man annehmen, dass der klonale Pool in einen quieszenten Zustand geriet, während die klonale Population die Apoptose erfuhr.
Es scheint, dass der transkriptomische Level des IFN-γ zwar eine Rolle spielt, aber andere wichtige Entzündungsmediatoren in der Genexpression für die Apoptose entscheidend sein müssen. Die Schlussfolgerung, IFN-γ sei direkt für die Apoptose der CD34⁺-Zellen verantwortlich, wirkt methodisch verkürzt und nicht ausreichend belegt.
Munje et al zeigten in ihrem Experiment:232
“Similar to the differences between freshly isolated and cytokine-induced CD34+ cells, the functional pathways that changed when cytokine-induced CD34+ cells were compared to AML were also related to cell cycle and cell division (Tables 2 and 3). Genes more highly expressed in AML blast samples compared to dividing HSPCs involved changes in regulation of transcription and gene expression (e.g., JMY, KLF7, PP2Aα, NF-AT5, MDM4, HMGB1, IRF1) or regulation of RNA and other cellular and metabolic processes (e.g., ZNF222, CCL5, TRIP8, CCND2).”In diesem Abschnitt wurden epigenetische Konsequenzen der modRNA/LNP-Technologie nach Injektion von BNT162b2 und mRNA-1273 untersucht. Es lässt sich ein klarer Trend erkennen. Daraus ergeben sich jedoch weitere offene Fragen. Zunächst muss berücksichtigt werden, dass sich alle hier diskutierten Arbeiten zu BNT162b2 und mRNA-1273 ausschließlich auf Blutproben und die Immunzellen bzw. Immunreaktionen fokussierten. Wie in Teil 1, Abschnitt 2 gezeigt, werden jedoch eine Vielzahl an unterschiedlichsten Zellen transfiziert. Ist dort auch eine epigenetische Umgestaltung zu erwarten? Wie werden sich die daraus resultierenden Zellverhaltensänderungen äußern? Die epigenetische Dynamik ist genauso plastisch und komplex wie die der Signaltransduktionsnetzwerke und darüber hinaus.233 234 235 236 237 238 Wie in Abschnitt 4.5.1 gezeigt, sind beide Aspekte nicht voneinander zu trennen.239 Wu et al. liefern dazu eine umfangreiche Übersichtsarbeit.240
4.6 modRNA, LNPs und unvorhersagbare Konsequenzen – Antagonismus und Synergismus
Wie in Teil 1, Abschnitt 3 diskutiert, haben die Modifikationen der modRNA zusätzliche biologische, biochemische und biophysikalische Effekte, wie eine veränderte sekundäre und tertiäre Struktur, andere Schmelztemperaturen, damit verbunden andere miRNA-Bindungsstellen, etc. In diesem Abschnitt werden unter den einzelnen Unterabschnitten verschiedene Aspekte möglicher synergistischer und antagonistischer Effekte der modRNA, die plausibel erscheinen, untersucht.
4.6.1 microRNA (miRNA) und Expressionsveränderungen als epigenetische Treiber
Im folgenden Abschnitt werden mögliche miRNA-Expressionsveränderungen nach der BNT162b2 und mRNA-1273-Transfektion untersucht. Anschließend folgt eine kompakte Diskussion, ob diese Veränderungen ebenfalls epigenetische Konsequenzen nach sich ziehen können.
Um die Gefahren, die sich hinter einer unkontrollierten miRNA-Maschinerie verbergen nachvollziehen zu können, muss auch unter diesen Aspekten zunächst die Netzwerkdynamik und Feedback- und Feedforward-Dynamik geklärt werden. Diese lassen sich wie folgt zusammenfassen:
miRNAs werden von Signaltransduktion reguliert.241 Saj und Lai fassen beispielsweise zusammen, dass die MAPK-Signalwege pri-miRs (das heißt die noch nicht via Drosha/Dicer-Komplex prozessierten Vorläufer der miRNA) aktivieren können.242 Dies wird über die Aktivierung von PREB1 oder AP1 initiiert, je nachdem, wie der kanonische Signalweg der MAPK (RAS-RAF-MEK-ERK) mündet. Dadurch wird pri-miR 143/145 oder pri-miR21 zur Prozessierung transkribiert. Es ist plausibel, eher davon auszugehen, dass wesentlich mehr miRs je nach Signalinitiierung und downstream Kaskadenregulation angestoßen werden. Bortoletto et al. demonstrieren in 2023, dass das Kirsten Ratsarcoma (KRAS) der MAPK-Signalwege bei Krebsentwicklung miRNAs “übernimmt” (multiple Schritte der Prozessierung verändert).243
miRNAs regulieren die Signaltransduktion und etablieren Feedbackschleifen.244 Bereits das Review von Saj und Lai (Fußnote 242) deutet an, dass miRNAs nicht nur von der Signaltransduktion angestoßen werden, sondern selbst regulatorische Rückkoppelungen auf diese ausüben. Paroo et al. demonstrieren in vitro, dass die MAPK die miRNA-Maschinerie reguliert aber auch zeitgleich vom Phosphorylierungszustand des Dicer/TRBP-Komplexes reguliert wird.245 Dies weist auf eine Feedbackschleife hin. Dabei identifizieren die Autoren Serinreste des DICER/TRBP-Komplexes als zentrale Phosphorylierungsstellen (S142, S152, S283, S286). Dies ist in Einklang mit dem Review von Copeland et al. welche eine direkte Regulation der Rezeptor- und Signalfunktionalität durch miRNA vorschlagen.246 Sie schreiben dazu:
”These findings support the idea that miRNAs play an essential role in regulating gene expression levels by acting in a combinatorial fashion to target multiple components of a signaling pathway.”
Eine weitere Vorbedingung, die Erwähnung finden soll, bevor miRNA-Veränderungen nach modRNA-Geninjektionen untersucht werden können, ist, zu verstehen dass miRNAs in Netzwerken kooperieren. Plaisier et al. demonstrieren beispielsweise, dass eine Alteration von miR-29 auch das Netzwerk um miR-767-5p stören kann.247 Mit anderen Worten, da miR-29 und miR-767-5p überlappende Seed-Sequenzen besitzen, agieren sie in sich teilweise überlappenden Netzwerkdynamiken, wodurch eine Dysregulation zu falschen Bindungsmustern führen kann.
Hill und Tran fassen für miR-21 zusammen, 248
dass “miRNAs may also regulate other miRNAs, with far-reaching influence on miRNA and mRNA expression. We explore this realm of small RNA regulation with a focus on the role of the oncogenic miR-21 and its impact on other miRNA species.”Daraus lässt sich festhalten, dass es sich um komplexe und dynamische Netzwerke handelt, welche – ähnlich den Signaltransduktionsnetzwerken – auf In- und Output kontextspezifisch reagieren.
Abbildung übernommen aus Jin, Daeyong, and Hyunju Lee. “A Computational Approach to Identifying Gene-microRNA Modules in Cancer.” PLOS Computational Biology 11, no. 1 (January 22, 2015): e1004042. https://doi.org/10.1371/journal.pcbi.1004042.; Lizenz: CC BY 4.0
Diese Netzwerkdynamiken sollten in einem vierdimensionalen Vorstellungsmodell verstanden werden. Jin & Lee publizierten dazu eine umfassende, computergestützte Arbeit.249 Die Autoren entwickelten ein Modell zur Identifikation von funktionellen miRNA-mRNA-Modulen in der Krebsprogression. Es erfasst spezifische Bindungsnetzwerke.
Gao et al. notieren 2025:250
”By developing and validating a miRNA discovery pipeline, we show that at least 2726 novel canonical miRNAs, majority of which represent novel miRNA families, exist in just one human cell line compared to just 1914 known miRNA loci.”Gibt es Hinweise auf solche Verschiebungen?
Eine bemerkenswerte Pionierarbeit liefert Fujii in 2021.251 Der Autor modellierte in silico potentielle miRNA Bindungsstellen in BNT162b2. Darunter befand sich miR-21. Er identifiziert insgesamt 16 mögliche CovS‑RNA‑abgeleitete miRNA-Bindungsstellen. Der Autor schreibt dazu:
”Further, CovS-miR-21 downregulated Rho associated coiled-coil containing protein kinase (ROCK2) and aryl hydrocarbon receptor nuclear translocator like (ARNTL), and CovSmiR- 3 decreased lysine methyltransferase 2C (KMT2C). Therefore, in the METS analysis, CovS-miR-21 suppressed the function of Ras homolog family member A (RhoA)/Rock2 signaling and circadian rhythm, and CovS-miR-3 inhibited histone H3-K4 methylation.”Wie bereits in diesem Abschnitt dargelegt, könnte dies ein ganzes Netzwerk von miRNA-Interaktionen betreffen. Da es sich um eine in silico Studie handelt, ist das Resultat jedoch nur als erster Hinweis und Möglichkeit in Betracht zu ziehen.
Ein erstes Indiz zu veränderten miRNA-Profilen liefern Miyashita et al.252 Sie entnahmen von 61 Probanden nach der 1. und 24 Probanden nach der 2. BNT162b2-Injektion Blut. Anschließend sortierten die Autoren diese in 2 Gruppen für lokale Nebeneffekte nach dem Schweregrad. Sie untersuchen 16 miRNAs in extrazellulären Vesikeln (Exosomen), die sie isolierten und anschließend mittels miRNA-Assay Profiling auswerten und halten fest:
”Additionally, miR-148a levels were positively correlated with systemic scores after the first dose (Fig. 6c). Unlike seasonal flu vaccination, miR-451a levels were not connected with any scores (Fig. 6b–e). Since EV miR-92a-2-5p levels were correlated with local and systemic scores, we investigated which symptoms were associated with miR-92a-2a-5p. Our statistical analyses suggested that miR-92a-2-5p levels in serum EVs were significantly decreased in subjects who had redness, headache, or joint-pain symptoms (Fig. 6f). Systemic scores of females were higher than those of males, but there was no difference in miR-92a-2-5p levels between females and males (Supplementary Fig. 5a, b), suggesting that miR-92a-2-5p is not associated with gender difference.”Diese Studie kann jedoch nur als weiteres Indiz betrachtet werden, da die Autoren kein Immunzellclustering machten und somit die zirkulierenden miRNA-Veränderungen nur als Resultat von intrazellularer miRNA-Regulationsveränderungen verstanden werden können, deren Profil sich nicht ableiten lässt. Die Autoren diskutieren zwar mögliche Mechanismen, liefern jedoch keine weiteren experimentellen Beweise. Des Weiteren ist nicht klar, wie sich die beobachteten zirkulierenden miRNAs verhalten, welche Zellen von den Exosomen transfiziert werden und welche systemischen Konsequenzen sich aus diesem Umstand ergeben. Zwar ist experimentell nachgewiesen, dass exosomale miRNAs Zell- zu Zellkommunikation über weite Strecken ermöglichen (endokrine Kommunikation), aber es bleibt unklar, welche Effekte sich aus den beobachteten Veränderungen ergeben.253 254
Abbildung übernommen aus Chetta, Massimiliano, Marina Tarsitano, Maria Oro, Maria Rivieccio, and Nenad Bukvic. “An in Silico Pipeline Approach Uncovers a Potentially Intricate Network Involving Spike SARS-CoV-2 RNA, RNA Vaccines, Host RNA-Binding Proteins (RBPs), and Host miRNAs at the Cellular Level.” Journal of Genetic Engineering and Biotechnology 20, no. 1 (September 6, 2022): 129. https://doi.org/10.1186/s43141-022-00413-5.; Lizenz: CC BY 4.0
Chetta et al. identifizieren in silico 39 potenzielle Host-miRNAs, die in der Lage sind, an das SP-RNA-Motiv zu binden.255 Sie schreiben zu ihrer Tabelle 1:
”The in silico sequences of the three motifs identified in S protein-encoding mRNA, the RBPs capable of binding the sequences, and the miRNAs with similar sequences to the motif are reported in the table” Dabei ist jedoch zu beachten, dass – wie unter Teil 1, Abschnitt 3.4.3 und 3.4.4 dargelegt – sich durch die m1Ψ-Substitutionen die Bindungsregeln verschoben haben könnten, da die gesamte sekundäre und tertiäre modRNA-Struktur andere Eigenschaften aufweist, als die der natürlichen Spike-RNA.(Teil 1, [Fußnote 197 - Fußnote 202])
Abbildung übernommen aus Lin, Yen-Pin, Yi-Shan Hsieh, Mei-Hsiu Cheng, Ching-Fen Shen, Ching-Ju Shen, and Chao-Min Cheng. “Using MicroRNA Arrays as a Tool to Evaluate COVID-19 Vaccine Efficacy.” Vaccines 10, no. 10 (October 8, 2022): 1681. https://doi.org/10.3390/vaccines10101681. ;Lizenz: CC BY 4.0
Lin et al. entnahmen von Schwangeren in verschiedenen Schwangerschaftsstadien maternale Blutproben und beobachteten die miRNA-Verschiebungen zwischen Frauen ohne mRNA-1273-Injektion und dreifach transfizierten Frauen.256 Sie beschriften die übernommene Tabelle wie folgt:
“MIRAscan was used to detect 83 microRNAs’ expression profiles in plasma samples collected from two pregnant women. Additionally, selection criteria to identify differentially expressed microRNAs were as follows: |ΔCq| ≥ 1 (including ΔCq ≥ 1 and ΔCq ≤ −1); microRNAs highlighted in orange indicate overexpression of microRNAs in the vaccinated, three-dose group (M1) while those highlighted in green represent overexpression of microRNAs in the unvaccinated, no-dose group (M2).”Es ist festzuhalten, dass die Legendenbeschriftung so nicht korrekt sein dürfte, da die positiven Werte eher – nach der Methodenbeschreibung – für eine Überexpression stehen würden. Es kann nicht ausgeschlossen werden, dass es sich um eine farbliche Verwechslung handelt oder aber sogar falsche Werte berechnet wurden. Hier wird von ersterer Möglichkeit ausgegangen. Die Autoren berufen sich unter anderem auf Légaré et al. und schreiben:257
“MicroRNAs have been found to regulate different mechanisms specific to pregnant women as substantial changes occur in the body to support the developing fetus[21].”Diese Einschätzung scheint jedoch nicht stimmig mit dem, was Légaré et al. zeigen: Aus der Tabelle von Lin et al. lässt sich beispielsweise eine klare positive ΔCq-Verschiebung von hsa-mir-21-5p zwischen den beiden Probandinnen erkennen, welche bei nicht mrna-1273-transfizierten Frauen auffällig niedriger exprimiert war. Dies bedeutet im Umkehrschluss, dass die Expressionsrate bei der untersuchten Schwangeren signifikant stieg, da es sich um PCR-Werte handelt und ein Faktor 2 berücksichtigt werden muss. Doch Légaré et al. untersuchten zwischen der 4. und 16. Schwangerschaftswoche und überprüften 2101 miRNA von denen sich 191 während der Schwangerschaft verschoben. hsa-miR-21-5p war nicht unter den Top10.
Abbildung übernommen aus Légaré, Cécilia, Andrée-Anne Clément, Véronique Desgagné, Kathrine Thibeault, Frédérique White, Simon-Pierre Guay, Benoit J. Arsenault, et al. “Human Plasma Pregnancy-Associated miRNAs and Their Temporal Variation within the First Trimester of Pregnancy.” Reproductive Biology and Endocrinology : RB&E 20 (January 14, 2022): 14. https://doi.org/10.1186/s12958-021-00883-1; Lizenz: CC BY 4.0
Da Lin et al. nur eine Schwangere pro Gruppe (mRNA-1273-transfiziert versus nicht transfiziert) zeigten, ist bereits daraus die Limitation gegeben, dass die Daten nur eine sehr schwache Aussagekraft haben. Demnach ist auch diese Studie nur als Hinweis modRNA-injektionsbedingter miRNA-Verschiebungen zu betrachten. Zusätzlich sollte berücksichtigt werden, dass der höhere BMI der nicht transfizierten Schwangeren (28.377 versus 21.797) eine höhere Entzündungsreaktion begünstigen kann.258 Zudem können auch die miRNA-Verschiebungen pathogene, transgenerationale Mechanismen auslösen.259 260 Es lässt sich klar festhalten, dass die hsa-miR-21-5p-Expressionsveränderung eher auf die Injektion zurückzuführen wäre. Ebenfalls bemerkenswert ist hsa- mir-16-5p. Diese zeigt einen vergleichbaren Trend. Und auch diese ist nicht unter den top-10 bei Légaré et al. Die Schlussfolgerungen und Assoziationen der Autoren sind interessant.
Stati et al. äußern ihre Bedenken und halten fest, dass261
“(…)studies have revealed that miRNA-451 (miR-451), downregulated after COVID-19 vaccination, is involved in various human physiological and pathological processes [45].”Brook et al. schreiben 2025:262
“Organs have different miRNA signatures, enabling the design and incorporation of multiple sequences in the untranslated region of an mRNA construct, dubbed multiorgan protection (MOP) sequences, which are complimentary to miRNA in tissues where transcript translation is not desired [8] (Figure 1D).”
Weiter schreiben die Autoren:
"Systemically distributed mRNA-LNPs and their encoded proteins can lead to reactogenicity, arising from innate immune responses to even low levels of LNP and mRNA components."
Und sie halten fest:
"Given that mRNA-LNP vaccination campaigns are generally an annual effort, we investigate potential areas for technological refinement, specifically in biodistribution and adjuvantation, to address reactogenicity and advance immunogenicity."Die Autoren vermerken zunächst korrekt, dass Organe über unterschiedliche und zellspezifische, teilweise sogar interaktionsabhängige miRNA-Signaturen verfügen. Erst dadurch wird eine gezielte posttranskriptionelle Regulation ermöglicht.263 Die Autoren schlagen jedoch vor, durch die Integration sogenannter “Multiorgan Protection (MOP) sequences” die 3'UTR von mRNA-Konstrukten zu modifizieren. Dadurch soll die Translation in bestimmten Geweben selektiv unterdrückt werden. Es bleibt von den Autoren völlig unbeachtet, dass die tatsächliche Kartierung zellspezifischer miRNA-Profile beim Menschen – insbesondere im Kontext dynamischer Pathomechanistik – noch keineswegs abgeschlossen ist. (Gao, 2025 [Fußnote 250]) Ebenso wird die derzeit nicht vollständig erfasste Vielzahl möglicher Off-Target-Interaktionen durch konkurrierende endogene RNAs (ceRNA), zirkulierende RNAs (circRNA) oder lncRNA-miRNA-Achsen vollständig ignoriert.264 265 266 267 268 Zudem scheint die Formulierung “Adjuvant” falsch gewählt. Wie in allen vorangegangenen Abschnitten belegt, sind LNPs weit mehr, als reine Adjuvantia und greifen tief in zelluläre Prozesse ein.
An dieser Stelle wird die Möglichkeit epigenetischer Verschiebungen, welche sich aus nicht kontrollierten miRNA-Verschiebungen ableiten lassen, kompakt diskutiert.
Chen et al. halten fest:269
“Furthermore, a previous study has reported that miR-148a-3p directly represses the expression levels of DNMT1 in human colon cancer cells (22). Therefore, DNMT1 expression was analyzed in cervical cancer tissues and a negative correlation was observed between the expression levels of DNMT1 and miR-148a-3p, implying that miR-148a-3p may regulate DNMT1 expression in cervical cancer.”Friedrich et al. diskutieren die Rolle zwischen miR-148 und miR-152 in Tumorentwicklungen.270
Drillis et al. schreiben:271
”In serum samples of patients with DLBCL, increased levels of miR-21, miR-155 and miR-210 have been identified, along with increased levels of miR-124, miR-532-5p miR-15a, miR-16, miR-29c and miR-155.”Daraus ergibt sich die Frage, wie sich stark herunterregulierte miR-Expressionen auswirken würden.
Zu dieser Frage dokumentieren Dionysiou et al.:272
“Compared to recipients of WT cells, allo-HCT recipients of miR-21 KO donors exhibit a distinct phenotype characterized by accelerated, severe, GVHD, as measured by survival and clinical GVHD score.”Während Brieño-Enríquez et al. in ihrem Experiment belegen:273
”We show that prenatal exposure to vinclozolin deregulates specific microRNAs in PGCs, such as miR-23b and miR-21, inducing disequilibrium in the Lin28/let-7/Blimp1 pathway in three successive generations of males.”Maccani et al. fassen 2011 die wichtigsten Rollen von mir-16 und mir-21 in Plazentagewebe wie folgt zusammen:274
“We have demonstrated the expression of key candidate miRNA in a large, population-based series of primary human placenta samples and their association with poor fetal growth, specifically identifying that reduced expression of miR-16 and miR-21 are significantly associated with growth restriction.”Dieser Abschnitt beleuchtete die potenziellen Auswirkungen von modRNA/LNP und LNPs auf miRNA-Verschiebungen anhand der aktuellen Literatur. Ein direkter Bezug zum SP besteht nicht, da es nicht der Schwerpunkt dieser Arbeit ist. Es lässt sich festhalten, dass die Möglichkeit von direkter miRNA-Interferenz plausibel erscheint und sich die Hinweise verdichten. Da jede mRNA bei der Translation reguliert werden muss, ist es bereits aus diesem Aspekt nachvollziehbar, davon auszugehen, dass die transfizierten Zellen ihren miRNA-Haushalt mit dem induzierten Gen teilen müssen und eine Dysregulation stattfindet. Die größte Limitation hierbei ist erneut, dass bisher ausschließlich Blutproben untersucht sind und keine intrazellularen Profile verschiedener Zelltypen vor und nach Transfektion existieren. Wie in Teil 1, Abschnitt 3.3 bereits gezeigt (Teil 1, Lockhart, 2018 [Fußnote 197]) verändern 100 Prozent Ψ-Substitution in einer mRNA die komplette Bindungsstellenaffinität. Und Orlandini Von Niessen et al. (Teil 1, Olandini Von Niessen, 2019 [Fußnote 139]) begründen die 3'UTR-Modifikation in BNT162b2 damit, die miRNA-Bindungsstellen minimieren zu wollen. Moderna schrieb, ohne weitere Beweise der Sicherheit nachzuliefern, dazu:275
”microRNA binding directly to mRNA effectively silences or reduces mRNA translation and promotes mRNA degradation. We design microRNA binding sites into the 3’-UTR of our potential mRNA medicines so that if our mRNA is delivered to cells with such microRNAs, it will be minimally translated and rapidly degraded.”4.6.2 “anekdotische Evidenz” – Fallberichte im Lichte der Kontroverse um das medial etablierte Wort “Turbokrebs”
Es gibt viele zu erwartende Nebenwirkungen, die sich im Laufe der diskutierten Signaldysregulation und miRNA-Veränderungen, zeigen können. Eine der wahrscheinlichsten, die sich bereits in epidemiologischen Daten ankündigt, ist Krebs. Erste Daten dazu wurden von einer japanischen Forschergruppe ausgewertet. Fujisawa et al stellen eine klare Korrelation zwischen Krebsratenzunahme und Impfkampagne her.276 In diesem Licht muss “Turbokrebs” als soziales Medienkonstrukt diskutiert werden.
Der Begriff “Turbokrebs” findet häufig Erwähnung auf sozialen Netzwerken. Und er wird in zugespitzten medialen Darstellungen erwähnt. Der Begriff ist jedoch aus wissenschaftlicher Perspektive nicht plausibel. Es gibt diverse Erklärungsmodelle, die diese real zu beobachtenden, ungewöhnlich aggressiven und sich rapide entwickelnden Krebse auslösen könnten. Dieser Abschnitt untersucht das Phänomen und listet eine Reihe von Fallberichten, um die mechanistische Plausibilität anhand real beobachteter Fälle zu stützen. Einige Studien werden (kursiv und in Klammern gesetzt) stichpunktartig kommentiert, da ihre Befunde auf den ersten Blick nicht auffällig erscheinen.
Zamfir et al.277
“Hematologic Malignancies Diagnosed in the Context of the mRNA COVID-19 Vaccination Campaign”
T-cell lymphoma and acute myeloid leukemia
Tachita et al.278
“Newly Diagnosed Extranodal NK/T-Cell Lymphoma, Nasal Type, at the Injected Left Arm after BNT162b2 mRNA COVID-19 Vaccination”
Extranodal NK/T-cell lymphoma, nasal type
Sogabe et al.279
“Warm autoimmune hemolytic anemia and IgM-monoclonal gammopathy following BNT162b2 COVID-19 vaccine in a patient with splenic marginal zone lymphoma“
Warm autoimmune hemolytic anemia and IgM-monoclonal gammopathy
( → Vakzininduzierte Progression oder Transformation eines indolenten B-Zell-Lymphoms?
→ Studie bleibt vage)
Sekizawa et al.280
“Rapid Progression of Marginal Zone B-Cell Lymphoma after COVID-19 Vaccination (BNT162b2): A Case Report”
marginal zone B-cell lymphoma
O'Sullivan et al.281
“Misinterpretation of Glioblastoma as ADEM: Potentially Harmful Consequences of over-Diagnosis of COVID-19 Vaccine-Associated Adverse Events”
Glioblastoma
Mizutani et al.282
“Two Cases of Axillary Lymphadenopathy Diagnosed as Diffuse Large B-Cell Lymphoma Developed Shortly after BNT162b2 COVID-19 Vaccination”
Diffuse large B-cell lymphoma
Kyriakopoulos et al.283
“Bell’s Palsy or an Aggressive Infiltrating Basaloid Carcinoma Post-mRNA Vaccination for COVID-19?”
Aggressive infiltrating basaloid carcinoma
Kim et al.284
“Drug-Induced Sarcoidosis-like Reaction Three Months after BNT162b2 mRNA COVID-19 Vaccination: A Case Report and Review of Literature”
Drug-induced sarcoidosis-like reaction
(→ DISR?
→ unspezifische Marker (sIL-2R)
→ Ausschlussdiagnostik → keine Reversibilität gezeigt
→ Malignität oder subklinische Transformation nicht sicher ausgeschlossen)
Kaimori et al.285
“Histopathologically TMA-like Distribution of Multiple Organ Thromboses Following the Initial Dose of the BNT162b2 mRNA Vaccine”
TMA-like distribution of multiple organ thromboses
( → “A 72-year-old woman with a history of diffuse large B-cell lymphoma in the stomach and hyperthyroidism received the first dose of the BNT162b2 mRNA vaccine and died 2 days later.”
→ Formulierung „mit vorbestehendem DLBCL“ ist retrospektiv und nicht eindeutig
→ siehe Babu & Bhat286 )Goldman et al.287
“Rapid Progression of Angioimmunoblastic T Cell Lymphoma Following BNT162b2 mRNA Vaccine Booster Shot: A Case Report”
Rapid progression of angioimmunoblastic T-cell lymphoma
Cohen et al.288
“Correlation between BNT162b2 mRNA Covid-19 Vaccine-Associated Hypermetabolic Lymphadenopathy and Humoral Immunity in Patients with Hematologic Malignancy”
Vaccine-associated hypermetabolic lymphadenopathy
Cavanna et al.289
“Non-Hodgkin Lymphoma Developed Shortly after mRNA COVID-19 Vaccination: Report of a Case and Review of the Literature”
Non-Hodgkin lymphoma
Ahmed et al.290
“Multiple Keratoacanthomas Following Moderna Messenger RNA-1273 COVID-19 Vaccination Resolved With 5-Fluorouracil Treatment: Case Report”
Multiple keratoacanthomas
Gentilini et al.
“A Case Report of Acute Lymphoblastic Leukaemia (ALL)/Lymphoblastic Lymphoma (LBL) Following the Second Dose of Comirnaty®: An Analysis of the Potential Pathogenic Mechanism Based on of the Existing Literature”
Acute Lymphoblastic Leukaemia (ALL)/Lymphoblastic Lymphoma (LBL)
Es gibt mindestens zwei Erklärungsmodelle, die die beobachteten Phänome erklären können. Zunächst wird das Konzept “Rapid onset Cancers” untersucht.
Bilger et al. merken an, dass: 291
“Human cancers are generally thought to develop over the course of decades. Such slow progression is well documented for a variety of cancers that we designate “slow-onset” cancers. “Rapid-onset” cancers, in contrast, can develop in a matter of months in humans or in as little as 9 days in mice.”
Und weiter halten die Autoren fest, dass die "Most neuroblastomas amplify MYCN or delete miRNAs that bind to MYCN mRNA (...)"
Und die "Expression of the pluripotent regulator LIN28B, which increases MYC expression, can initiate neuroblastoma development in transgenic mice [56]."
Darüber hinaus stellen Bilger et al fest, dass "(...) (Anaplastic Lymphoma Kinase) are associated with familial neuroblastoma, but this gene is rarely mutated somatically [50,51]."
Die für diese Hypothese wichtigste Anmerkung, die Bilger et al machen ist, dass die "Expression of Sonic Hedgehog (SHH) pathway mRNAs is also frequently upregulated." You et al. diskutieren die Signaltransduktionswege und deren direkte wechselwirkende Krebsmetaboliteneinflüsse. 292
Tartaglia & Gelb schreiben in ihrem Reviewartikel:293
“Activating somatic RAS gene mutations occur in approximately 30% of human cancers, but the upregulation of this signaling pathway can also result from enhanced function of upstream signal transducers or RAS effectors, as well as from inefficient function of feedback mechanisms.”Dies deckt sich auch mit den Feststellungen von Bahar et al.294 R. Seger merkt dazu an:295
”The most notable of these are the mitogen-activated protein kinase (MAPK) cascades that are involved in the regulation of essentially all stimulated processes, including proliferation, differentiation, motility, stress response, survival, and apoptosis (for a review see: [1,2,3]. Being such central signaling components, the dysregulation of the MAPK cascades often generates pathologies such as cancer, autoimmune diseases, neurological disorders, and others [4,5,6,7].” Wie zuvor dargelegt, ist eine akute Dysregulation der MAPK als Knotenpunkt verschiedenster Signalwege, wie WNT, SHH, RHO, JAK/STAT und den dahinter geschalteten Transkriptionsfaktoren, beispielsweise E2F, NF-κB und SMAD und Transkriptionsfaktoraktivatoren, wie ERK und Akt, zu vermuten.296 297 298 299 Es stellt sich zusätzlich das Problem, wie supprimierte TLRs (Teil 1, Abschnitt 3.3.1, 3.3.2 und 3.4.2.), welche ebenfalls eine zentrale Rolle im MAPK-Netzwerk einnehmen, sich auf diese Signaldysregulation auswirken werden (Abschnitt 4.5.1).
Li et al.: fassen zusammen, dass:300
"It was note that both differentiation and maturation of mDCs can be suppressed by imbalance of cytokines in the tumor microenvironment."Während Zheng et al. in ihrem Review schreiben:301
“Overall, TLR3 tends to play an antitumor role, but its protumor role should be mentioned in clinical applications.”Es sei nochmals erwähnt, dass TLRs ebenfalls untereinander Netzwerkdynamiken einnehmen und eine Dysregulation auch zellweite Konsequenzen haben wird.(Mishra, 2022 [Fußnote 164] – Assier, 2007 [Fußnote 174])
Wenn die Kombination aus verschiedenen Signaltransduktionswegen bidirektionale, divergente und entkoppelte Expressionsveränderungen erfuhren, erscheint es plausibel, daraus ein eher idiopathisches und sich schnell entwickelndes Krebsmilieu zu erwarten. Zusätzlich sollten auch lyso- und zytotoxische Effekte berücksichtigt werden. 302 303 304 305 306 Usmani & Mempel fassen zusammen, dass “Cancer cells relax and resist cytotoxic attack”.307
Eine zweite Möglichkeit, die in Betracht gezogen werden sollte, welche ebenfalls die zeitliche Diskrepanz von einer “normal erwartbaren” Krebsentwicklung erklären würde, ist die “Hyper Progressive Disease (HPD)”-Theorie:
Arasanz et al. schreiben in ihrem Review:308
“The first attempt to identify underlying immunological mechanisms of HPD was published in early 2019 and targeted myeloid populations. Lo Russo G et al. found a significant infiltration by M2-like CD163+ macrophages in all HPD patients in pretreatment tumor samples of NSCLC patients receiving immunotherapy.”
Die Autoren betonen, dass "(...) HPD is not a phenomenon with a single cause but a consequence of several events.", was im Falle der modRNA/LNP-Injektionen exakt die Ereigniskaskaden beschreibt.
Weiterhin schreiben Arasanz et al., dass "(...) that CTLA-4-, OX-40- or CCR4-targeted therapies could be strategies to prevent HPD by depleting Tregs (...)". Wenn also diverse Immunzelltypen, wie bis hier belegt wurde, eine epigenetische Reprogrammierung erfuhren, lässt sich folgende Überlegung aufstellen: T-Regulatorische Helferzellen(Treg) (eine Subspezies der CD4+-T-Zelllinie) wird unter anderem von Makrophagen maturiert und gelenkt.309 Guan et al. halten dazu fest:
”M2 macrophages, which are activated by IL-4 and IL-13, are responsible for inflammation resistance and tissue repair [60]. M2 macrophages produce anti-inflammatory cytokines, such as TGF-β or IL-10, which can also induce Treg differentiation.”Diese Erkenntnis deckt sich auch mit Ma et al.310
Was wäre demnach, wenn epigenetisch veränderte Makrophagenpopulationen gefunden wurden, für eine Konsequenz für die Treg zu erwarten? (Abschnitt 4.1.1, 4.5 und 4.5.2) Auch die Vorläuferzellen (Monozyten) spielen eine regulatorische Rolle bei der Treg-Entwicklung.311
Weiter halten Ding et. al fest:
”ICIs reverse the immunosuppressive state of T cells by disrupting the programmed cell death-1/programmed cell death-ligand 1 (PD-1/PD-L1) axis[40]. However, PD-1 receptors are present not only on the surface of T cells but also on the surface of many innate or acquired immune cells, including NK cells, monocytes, macrophages, Treg cells, and B cells[41]. Furthermore, immune cells have varying impacts on PD-1/PD-L1 axis disruption, boosting or inhibiting immune function.”Auch dieser Aspekt scheint wichtig, da es Hinweise auf eine gestörte Programmed Death1/Programmed Death-Ligand1 (PD-1/PD-L1)-Achse gibt.312 Barmade et al. stellten ein “unbiased” Immunprofil von Geimpften auf, die eine Myokarditis erfuhren und verglichen dieses mit Ungeimpften.313 Sie stellen fest:
”Upon examining the transcriptional signature of these expanded subsets, we noted the unique expression of various activation markers, including programmed cell death protein-1 (PD-1) (PDCD1) and CD38/HLA-DR (Fig. 4E), which was further validated by flow cytometry in CD8+ T cells (Fig. 4F).”Cuccaro et al. demonstrieren beispielsweise, dass PD-L1 übersteuerte Granulozyten zu klassischen Hodgkin Lymphomen führen können.314
Es müssen noch weitere Mechanismen, die sowohl für HPD als auch “rapid onset Cancer” greifen würden, in Betracht gezogen werden. Beispielsweise sollte auch die Möglichkeit, dass ein Codon Readthrough geschieht (Teil 1, Abschnitt 3.4.1), berücksichtigt werden. Was hätte ein translatierter Poly-A-Schwanz für Folgen?
Guydosh & Green zeigen in 2017:315
“Translation of the poly(A) tail leads to upstream mRNA cleavage and ribosome stalling at the 3′ ends of the resulting truncated fragments.”Diese Fragmente könnten demnach ebenfalls zusätzliche, biologisch paradoxe Effekte auslösen, die die Zellhomöostase beschleunigt aus der Balance bringen.316
Nishimura et al. liefern 2024 eine Erfassung vermuteter haupttreibender Dysregulationen bei HPD und halten fest, dass “the contribution of M2 macrophages, TGF-β signaling, and interleukin-8” die Pathogenität trieb und zur Letalität führte.317
Es ist in der gängigen Lektüre unstrittig, dass eine Überexpression des PD-L1 mit Tumorwachstum und der Inhibierung von Zelltodmechanismen in Verbindung steht.318 319 320
Roy et al. fassen zusammen: 321
”Our previous studies with intra-macrophage pathogen Leishmania donovani documented that during early infection, Leishmania inhibits the expression of death receptor, PD-1 to preventing host cell apoptosis [7]. Interestingly, Leishmania induced the expression of PD-1 at late hours but manage to escape apoptosis by inducing SIRT1, which inhibits cell death in a PD-1- independent manner [8]. The reason behind the increased PD-1 expression during late infection was to create an anti-inflammatory environment aiding in parasite survival [8].”Daraus lässt sich schließen, dass eine chronische Entzündung mit PD-L1-Überexpression zu unvorhersehbaren Zellwachstumsmustern führen und auch die parakrine und endokrine Kommunikation beeinflussen könnte.322
Wenn die MAPK, so wie plausibel nahegelegt, eine dauerhafte Dysregulation erfuhr (und sei es auch nur “moderater Natur") wäre eine Dysregulation des PD-L1 zu erwarten.323 324 325
Und auch hier scheint sich der Kreis zu einer zu erwartenden PtdIns-Dysregulation zu schließen. In einem jüngst erschienen Preprint zeigen Erdoğdu et al., dass ausschließlich bei BNT162b2-transfizierten, krebsbetroffenen Patienten folgende Werte höher waren als bei nichttransfizierten Krebspatienten:326
↑ Tetrahydrofolsäure
möglicherweise Ausdruck einer Modulation des Folatzyklus?
kompensatorische Veränderungen im Nukleotidstoffwechsel?
↑ Phosphorylcholin
Marker für einen veränderten Membranstoffwechsel?
Marker für Zellproliferation, ggf. zelluläre Umstrukturierung?
↑ N-Formyl-L-Glutaminsäure / N-Acetyl-L-Asparaginsäure
Mitochondrialer Stoffwechsel?
Übernommen aus Erdoğdu, Batuhan, Ozan Kaplan, Bilge Başak Fidan, Mustafa Çelebier, Ümit Yavuz Malkan, and Ibrahim C. Haznedaroglu. “Metabolomic Profiling of Leukemic Hematopoiesis: Effects of BNT162b2 mRNA COVID-19 Vaccine Administration.” Current Molecular Medicine, June 3, 2025. https://doi.org/10.2174/0115665240361878250601074746.; Lizenz: CC BY 4.0
Weiterhin bemerkenswert ist die leichte Erhöhung von sn-Glycero-3-phospho-1D-myo-inositol (Phosphatidylinositol). Auch wenn die statistische Signifikanz nicht erreicht wurde, sollte über eine aktive Beteiligung in der Regulation nachgedacht werden. Wie unter Teil 1, Abschnitt 1.1 diskutiert, sind Netzwerkdynamiken nicht statisch und besonders die Arbeit von Thiemicke & Neuert. spielt hier eine entscheidende Rolle. (Teil 1, Abschnitt 1.1, Thiemicke, 2023 [Fußnote 21]).
Thiemicke & Neuert schreiben dazu:
“In contrast, cells of any organism or in any natural environment experience a wide variety of gradual changes in external stimuli. Current treatments and therapies are based on observed phenotypes (purple plane) and current disease models (grey star) in such acute treatments, potentially missing or misinterpreting the response of a cell to the stimulus under physiological conditions.”Zudem scheinen Erdoğdu et al. (Fußnote 326) nicht die Subgruppen der Phosphatidylinositole identifiziert zu haben. Wenn – wie von Posor et al. (Posor, 2022 [Fußnote 74]) festgehalten – diese 2nd messaging Responder nur 1%–5% des gesamten Lipidmosaiks ausmachen und jede noch so kleine Varianz statistisch signifikante Feedbackloops erzeugen kann, ist jede Veränderung bereits relevant.337 338 339 340 341 Die Autoren Erdoğdu et al. (Erdoğdu, 2025 [Fußnote 326]) selbst betonen, dass sie keine Kausalität herstellen konnten. Sie legen jedoch weitere Untersuchungen nahe. Ihre Daten implizieren mechanistische Plausibilität.
Das zugrunde liegende Problem ist jedoch, dass Ärzte und Menschen sich nicht bewusst sind, dass sie Nebenwirkungen des Impfstoffs erleben. Die mechanistische Wahrscheinlichkeit, wie der Impfstoff auf Zellen wirkt und Nebenwirkungen verursacht, ist unbekannt. Hier wurden mit Schwerpunkt auf Krebs die wahrscheinlichsten Mechanismen diskutiert, die auch zu Autoimmunität, erhöhtem Infektionsrisiko und vielen anderen Komplikationen führen.
Es wurden zwei plausible Mechanismen vorgeschlagen, die das Phänomen des sogenannten „Turbo-Krebses” aus pharmakodynamisch-systembiologischer Sicht erklären.
Wir empfehlen, diese Terminologie zu vermeiden und stattdessen auf die tatsächlichen mechanistischen Möglichkeiten (HPD oder „rapid onset cancer”) Bezug zu nehmen. Dies erleichtert a) die Identifizierung und Klassifizierung, b) schafft eine einheitliche Datenbank, die es Forschern ermöglicht, schneller und tiefer auf vorhandene Literatur für weitere Untersuchungen zuzugreifen, und c) demonstriert wissenschaftliche Integrität.
4.6.3 Ungelöste Fragen und Beobachtungen – die Unbekannten
In diesem abschließenden Abschnitt der hypothesengetriebenen Arbeit werden bislang kaum adressierte Fragestellungen untersucht bzw. formuliert – insbesondere dort, wo nach aktuellem Kenntnisstand keine einschlägige Literatur existiert.
Die erste logische Frage, die sich aus den erwähnten Translationstemperaturunterschieden und strukturellen Alterationen der modRNA ergibt (Teil 1, Abschnitt 3.2), ist, wie sich diese Effekte auf die Ribosomen auswirken.
Tanaka et al. untersuchten als erste diese Frage und halten in ihrer Arbeit fest:342
“Unlike the destruction caused by chemotherapy with platinum (II) complex, progressive destruction in 18S ribosome was identified even at 6 months after vaccination.”Würden daraus weitere pathomechanistische Konsequenzen zu erwarten sein?343 344
Eine weitere naheliegende Frage betrifft die mögliche Beeinträchtigung der Mitochondrien durch Transfektion und endosomale Disruption.
Moghimi & Simberg merken zu dieser kaum adressierten Frage an:345
“Therefore, it is likely that SM-102 directly activates some intracellular pattern-recognition receptors, while both ionizable cationic lipids also trigger signals for the NLRP3 inflammasome, presumably, following transient disruption of plasma and/or endosomal membranes (e.g., through mitochondrial reactive oxygen species-mediated calcium flux and some caspases). Intriguingly, incorporation of the modified RNA in a liposome composed of the cationic lipid DOTMA neither induced IL-1β release nor any of its downstream cytokines.5”Dies deckt sich auch mit den Beobachtungen von Lonez et al. (Teil 1, Lonez, 2012, [Fußnote 91]), die vermerken, dass NLRP3 aktiviert wird, und den jüngsten Beobachtungen von Omo-Lamai et al. (Omo-Lamai, 2024 [Fußnote 79]). Im Umkehrschluss ergibt sich daraus die Überlegung, ob die freigesetzten reaktiven Oxygenspezies zusätzlichen Zellstress bewirken und in einer Feedbackschleife die Mitochondrien angreifen werden?
Auch die Frage, was genau mit den tertiären Aminen und deren Abbaublöcken geschieht, sollte untersucht werden (Teil 1, Jörgensen et al., 2023, [Fußnote 35] und Abschnitt 4.2.1): Wie werden die ionisierten tertiären Aminreste mit der modRNA interagieren? Wie wird sich der gestörte Recycling-Zyklus auf das gesamte Zellverhalten auswirken?
Werden – wie in Teil 1, Abschnitt 2.2 (Fußnoten 79 - 81) diskutiert – aldehydische sowie oxidative LNP-Verunreinigungen und kovalente LNP-Bindungen zusätzliche Off-Targets auslösen? In welchen Verhältnissen ist damit zu rechnen, wenn weniger als 60% (Schober, 2024 [Fußnote 157]) der modRNA und DNA-Fragmente effektiv eingeschlossen werden?
Welche synergistischen und antagonistischen Effekte sind zu erwarten, wenn zugleich eine hochinflammatorische TLR-abhängige Kaskade mit epigenetischen Konsequenzen ausgelöst wird und die Payload dafür sorgen soll, dass diese (TLRs) unterdrückt werden (Teil 1, Abschnitt 3.3 – 3.3.2)? Mit welchen synergistischen und antagonistischen Effekten ist zu rechnen, wenn es zur Störung des PtdIns-Zyklus kommt und somit zur Entkoppelung der zentralen Signaltransduktionswege? Diese regulieren auch die gesamte eukaryotische Initiationsmaschinerie (eIF).346 347 348
Eine weitere nicht gelöste Frage ist die Möglichkeit der Integration in das Wirtsgenom:
Wie häufig ist mit – durch Line-1-Retrotransposons angestoßenen – reversen Transkriptionsevents zu rechnen? 349 350 351 352 353
Es gäbe so viele weitere Fragen, die adressiert werden müssen, dass sich auch aus pharmakodynamisch-systembiologischer Perspektive die Natur der LNP/modRNA-Technologie zeigt (siehe Diskussion). Auch diese intensive Arbeit schließt mit mehr offenen Fragen als Antworten.
Diskussion
Abschließend wird zunächst geprüft, ob und inwieweit die hier 5 postulierten Hypothesen belegt wurden.
Die erste aufgestellte Hypothese ist:
”Weder Biodistribution noch Transfektionsraten sind in vivo tatsächlich kontrollierbar. “
Im ersten Teil (gesamter Teil 1, Abschnitt 2) wurde die intrinsische Natur der Lipidnanopartikel (LNPs) untersucht. Dabei wurden systematische Reviews und aktuelle Primärliteratur herangezogen. Es zeigt sich, dass die Lektüre durchgängig mit den Problemen von selektiver Datenauswertung und Missverständnissen geprägt ist. Diese Arbeit zeigt, dass bereits das Konzept Transfektion (Überwindung einer Zellmembran) und das Konzept des endosomalen Escapes (endosomal-lysomale Disruption) sowie die anschließende Translation in der LNP-Forschung verwässert und nicht einheitlich definiert sind. Durch ihre hochkomplexe Struktur, deren Inhalt methodisch nicht prüfbar ist (Limitation aufgrund aktueller technischer Möglichkeiten), ist es nicht möglich, die Verteilung in vivo ausreichend zu kontrollieren, so dass es generell zu fundamentalen Off-Targets kommt. Es wurde im zweiten Abschnitt anhand der Primärliteratur gezeigt, dass dies zum einen an kovalenten Bindungsproblemen liegt und zum anderen an der Proteincorona. Darüber hinaus wurde evident dargelegt, dass es Herstellungsprobleme gibt, welche bereits eine Vereinheitlichung der LNP-Formulierung eher unwahrscheinlich machen.
Damit ist die erste Hypothese — dass weder Biodistribution noch Transfektionsraten in vivo tatsächlich kontrollierbar sind — anhand der ausgewerteten Literatur vollumfänglich bestätigt.
Die zweite Hypothese heißt:
“LNPs sind durch ihre intrinsische Natur bioaktiv und verändern das gesamte Zellverhalten aufgrund tiefgreifender Eingriffe in das nichtlineare, zelltypabhängige Signaltransduktionsnetzwerk.”
Wie bereits erwähnt, wird häufig Transfektion mit Translation und Expressionsrate gleichgesetzt. Darüber hinaus werden oft Immunzellaktivierungen und Zytokinsekretion als direkte Reaktion einzig dem eingekapselten genetischen Material zugeschrieben. Dies wird in keiner Weise der Natur der LNP-Technologie gerecht. Außerdem bleibt bis heute offen, wie und unter welchen Umständen die Restbausteine der genutzten unterschiedlichen LNPs tatsächlich degradiert werden (Teil 1, Abschnitt 2, Jörgensen et al.).
Im 4. Abschnitt wurde dieser Logik folgend aus verschiedenen pharmakodynamischen und systembiologischen Aspekten untersucht, ob und inwiefern ein systemischer Eingriff in die Zellregulation durch die leeren LNPs bzw. LNP/modRNA unvermeidbar ist. Dazu wurde zunächst in den Abschnitten 4.1 bis 4.2.1 auf die biochemische und biophysikalische Natur von Zellmembranen und Rezeptoren sowie deren endosomale Regulations-, Reorganisations- bzw. Recyclingprozesse eingegangen.
Anschließend wurde eine Untersuchung der “omischen” Datenlage vorgenommen (Abschnitt 4.3). Es wurde gezeigt, dass sich die Datenlage zu nachhaltigen Signaltransduktionskaskadenverschiebungen verdichtet.
Darüber hinaus wurde in Teil 1, Abschnitt 2 nachgewiesen, dass bereits aufgrund der pharmakokinetischen Grundnatur der LNPs eine Bioreaktivität unvermeidbar ist.
Es wurde weiterhin in Abschnitt 4.4 einer der mittlerweile am besten dokumentierten Pathomechanismen bei transfizierten Menschen aus Zellperspektive untersucht. Abschließend wurde vorgeschlagen, welcher plausible Mechanismus zu einem IgG4-Klassenswitch aus Perspektive einer Signaltransduktionsentkoppelung/-verschiebung führt. Es wurden JAK/STAT-Feedbackloops mit IL-6 und IL-4 und zusätzlichen Synergien zwischen diesen und weiteren Interleukinen vorgeschlagen, welche gut in der Primärliteratur dokumentiert sind. Dies erscheint plausibel, da in Abschnitt 4.3 auch JAK/STAT-Verschiebungen aufgezeigt wurden.
Im Abschnitt 4.5 wurden epigenetische Konsequenzen untersucht und dargelegt, dass sich auch hier verdichtende Hinweise finden. Es wurde gezeigt, dass dies sowohl für LNP/modRNA als auch für leere LNPs gilt und bereits die Störung der Signaltransduktion epigenetische Folgen haben kann. Auch hier folgt die Untersuchung der Logik, die sich aus der “omischen Evidenz” ergibt. Es wurden IRF und ERK als zwei epigenetische Schalter betrachtet und weitere MAPK-Signalwege, Crosstalks und die Verknüpfung zu Toll Like Rezeptoren diskutiert.
Die zweite Hypothese ist durch viele Hinweise unterfüttert. Es lässt sich jedoch – aufgrund der Limitationen der genutzten Primärliteratur – nicht abschließend klären, ob diese Hypothese vollumfänglich greift oder nur teilweise. In anderen Worten: Es lässt sich nicht endgültig sagen, bei wie vielen Menschen in welchem Ausmaß die hier dargelegten Beobachtungen greifen werden und welche Bedingungen abweichende Phänomene begünstigen. Noch dazu ist ein epidemiologisches Problem zu beachten, das eine Erfassung noch weiter erschwert und sich auf eine akute Untererfassung in passiven Meldesystemen zurückführen lässt. Lazarus et al. vermerken 2011:359
“Likewise, fewer than 1% of vaccine adverse events are reported.”Baker et al. schreiben in 2015:360
“Although clinicians opened 94% of the AE reports we sent, they only elected to respond to 15%.”Zusätzlich wurde in Teil 1, Abschnitt 2 gezeigt, dass die Transfektion nichtlinear geschieht. Somit kann nur ein durchschnittlicher Off-Target-Score daraus abgeleitet werden, der keine Aussagen über kausale Ursachen zulässt.
Wir kommen zu dem Schluss, dass die zweite Hypothese nur durch großangelegte proteom,- transkriptom,- epigenom,- genom,- und Single Cell basierte Folgeuntersuchungen ex vivo, post mortem und in Humanprimaten endgültig abgeklärt werden kann. Dazu müssen möglichst eng gematchte Kohorten unter verschiedenen Prämissen untersucht werden. Somit ist die zweite Hypothese vorläufig durch die hier dargelegte Evidenz teilweise belegt.
Die dritte Hypothese ist:
“Bereits der Zellmembrankontakt mit LNPs löst die Signaltransduktionskaskaden rezeptorabhängig und -unabhängig in einer Art aus, die so unter physiologischen Bedingungen nicht zu beobachten ist.”
Auch diese Hypothese wurde durch die in den Abschnitten 4 und 2 diskutierten Mechanistiken teilweise gestützt. Insbesondere in Abschnitt 2.2 unter dem Aspekt der kovalenten Bindungsevents erscheint diese Aussage plausibel. Wie in den Abschnitten zur Membran- und Rezeptororganisation dargestellt wurde, hat jede Zellmembraninteraktion eine nicht abschließend verstandene Kaskadenflut an Interaktionen, welche in vierdimensionaler Art betrachtet werden müssen. (RAUM, ZEIT, INPUT, KONTEXT)
Diese Hypothese bedarf dringend weiterer Forschung, da dies zusätzlich potenzielle Risiken birgt. Somit ist auch diese Hypothese teilweise belegt.
Die vierte Hypothese lautet:
“Die daraus folgenden Konsequenzen sind weitreichend und unzureichend charakterisiert.”
Diese Hypothese stützt sich auf eine Vielzahl an Fallberichten, “omischen” Analysen und systembiologischen Untersuchungen zu leeren LNPs, die in der Literatur seit Beginn der modRNA-Injektionskampagne auffällig häufig dokumentiert wurden. Auch wenn sie teils als „anekdotische Evidenz“ klassifiziert wurden, ist ihre Häufung mittlerweile zu ausgeprägt, um sie weiterhin als rein anekdotisch abzutun. Weit über 3 000 dokumentierte Fälle zu vier verschiedenen Plattformen (modRNA und Vector) – von denen sich zwei technologisch stark ähneln und zwei bereits weitgehend vom Markt genommen wurden – sind aus historischer Perspektive zu zahlreich, um ignoriert zu werden.
Es wurden zwei verschiedene Mechanismen in Abschnitt 4.6.2 zum Phänomen der sich schnell und aggressiv entwickelnden Krebsfälle diskutiert. Weiterhin wurde auf scheinbar “nicht signifikante” Werte der PtdIns hingewiesen, auf eine falsche Interpretation dieser Werte aufmerksam gemacht und plausibel dargestellt, wieso entkoppelte Signaltransduktionsnetzwerke dieses Phänomen erklären können.
In den Abschnitten 4.1.1, 4.1.2 und 4.2.1 wurden anhand des GPCR-Beispiels idiopathische Mechanismen, die zu erwarten sind und sich auch in den Fallberichten wiederfinden, dargelegt.
Darüber hinaus wurden epigenetische Konsequenzen unter den Abschnitten 4.5 bis 4.6 und 4.6.1 diskutiert.
Diese Hypothese ist größtenteils belegt, da immer mehr idiopathische Krankheitsbilder zu beobachten sind und die Journalberichte kontinuierlich zunehmen. Es ist plausibel, davon auszugehen, dass viele neuere Berichte nicht den Impfstatus mit den Fällen in Verbindung bringen werden. So zeigte eine jüngste Studie von Pethő et al. eine Zunahme renaler Krankheitsbilder nach mRNA-Injektionen und halten fest:361
“Our hypothesis suggests that the development of kidney symptoms is closely linked to the administration of the mRNA vaccine. Before this, the patients were completely asymptomatic, and those with autoimmune diseases were stable.”Es ist zu erwarten, dass auch in den kommenden 20 bis 30 Jahren weitere Konsequenzen zu beobachten sein werden. Dies wurde in Abschnitt 4.1.2 am Beispiel der GPCRs und MAPK plausibel erklärt und mit gängiger Primärliteratur belegt.
Die fünfte und letzte Hypothese ist:
“Darüber hinaus wird die modRNA weitere antagonistische und synergistische Prozesse forcieren.”
Dies wurde anhand von Kariko et al., Sahin et al., Weissman et al. und weiteren Autoren, die diese Forschung maßgeblich für die Marktanwendung vorantrieben, diskutiert. In Teil 1, Abschnitt 3 wurde die intrinsische Motivation der Forscher erklärt, warum m1Ψ in die modRNA eingeführt wurde und wie sich diese Modifikation auf die Pattern Recognition Rezeptoren (PRR) auswirkt. Bereits daraus ergibt sich die logische Folgerung, dass die modRNA zusätzliche Effekte auf die bereits TLR-internalisierenden LNPs haben wird. Weiterhin wurden in 4.6. verschiedene Möglichkeiten und kaum untersuchte Interaktionswege der modRNA mit verschiedenen Zellkomponenten diskutiert.
Diese Hypothese scheint vollumfänglich durch gängige Primärliteratur belegt.
In dieser Arbeit wurde nahegelegt, warum LNPs auf jetzigem Forschungsstand und Verständnis ein potenzielles Risiko mit unvorhersagbaren Folgen sind und wieso jede Art von genetischem Material zusätzliche pathomechanistische Konsequenzen haben wird. Es wurde diskutiert, warum auch transgenerationale, epigenetische Konsequenzen zu erwarten sind. Diese Arbeit zeigte ferner rationale Mechanismen, die einige der idiopathischen Effekte mit der Wirkweise der LNPs in Einklang stellen und warum es plausibel erscheint, diese in kausalem Zusammenhang zu betrachten. Auf der einen Seite könnte man nun damit argumentieren, dass diese Technologie nur noch nicht optimiert ist. Auf der anderen muss dann aber die Frage lauten, wie lange eine vermeintliche Optimierung dauern soll, die seit 60 Jahren bis heute an den selben grundlegenden Problemen der Pharmakokinetik scheitert. Darüber hinaus ist spätestens seit den Reviews 2008 und 2012 von Lonez et al. bekannt, dass bereits Liposome so tief in das Zellgeschehen eingreifen, dass sie unkontrolliert Signaltransduktionswege verändern und proinflammatorische Prozesse auslösen.
Diese Arbeit fokussierte sich vorrangig auf die MAPK, da diese auch von Lonez et al. genannt wurde. Es ist jedoch zu berücksichtigen, dass auch viele weitere Signalwege und Crosstalks, beispielsweise CGAS/STING- und RHO-Signalwege existieren.362 363 364 365 366 367
Was ist daraus resultierend zu erwarten? Diese Frage kann nur spekulativ auf systembiologischen Überlegungen basierend beantwortet werden. Es ist anzunehmen, dass entweder kaum auffällige oder paradox erscheinende Blutparameter zu erwarten sind, welche sich über zunehmende Zeit verschieben oder sich normalisieren. Da Signaltransduktionswege sich gegenseitig ergänzen, ist zu vermuten, dass einige Prozesse temporär kompensiert werden können.368 369 370 371 Wie von Thiemicke & Neuert ([Teil 1, [Fußnote 21]) erwähnt, können die daraus resultierenden pathogenen Effekte bis zu Jahrzehnte später auftreten. Daher wird dringend empfohlen, die Perspektive von Blutproben auf die der intrazellularen Mechanismen zu verlagern, da diese Technologie eben nicht nur im Körper verteilt und metabolisiert wurde, sondern Gewebe und Zellen direkt nachhaltig verändert hat.
Schlussfolgerung
Wir empfehlen ein sofortiges Moratorium sämtlicher geplanten, auf LNP-Technologie basierenden Technologien, da zuerst all die hier adressierten Fragen und nicht ausreichend untersuchten Mechanismen falsifiziert werden müssen und genau betrachtet werden muss, was für Langzeitfolgen zu erwarten sind und wie schwer diese sich äußern. Weiterhin muss die Möglichkeit exkludiert werden, dass es zu epigenetischen, transgenerationalen Konsequenzen kommt.
Erst dieses Verständnis ermöglicht die Entwicklung angemessener Therapieoptionen für die durch diese Plattform induzierten Schäden.
Declaration of Competing Interest
The author declares no competing interests or financial conflicts of interestAuthor contributions
F. Seger wrote the manuscriptAcknowledgments
This work was supported by Maria Gutschi, N. Lorenzen and reviewed by M. Gutschi, N. Lorenzen and others; discussed with David Cowley and others.Special thanks to Dr. Hélène Banoun, Dr. Annelise Bocquet, David Cowley, Dr. Claire Craig, Dr. med. Johanna Deinert, Dr. Lynn Fynn, Maria Gutschi, Nicole Lorenzen, Dr. Jacobus Passer, Dr. Jens Pohl, Dr. Adriana Radler-Pohl, Pinsolle Typhaine, Dr. Richard Urso, the whistleblower “jikkyleaks”, LeBiochimiste and many others who made this work possible through their feedback and constructive discussions.Pike, Linda J. “Lipid Rafts: Bringing Order to Chaos.” Journal of Lipid Research 44, no. 4 (April 2003): 655–67. https://doi.org/10.1194/jlr.R200021-JLR200.
Zhu, Dan, Wen C. Xiong, and Lin Mei. “Lipid Rafts Serve as a Signaling Platform for Nicotinic Acetylcholine Receptor Clustering.” The Journal of Neuroscience 26, no. 18 (May 3, 2006): 4841–51. https://doi.org/10.1523/JNEUROSCI.2807-05.2006.
Guirland, Carmine, and James Q. Zheng. “Membrane Lipid Rafts and Their Role in Axon Guidance.” In Madame Curie Bioscience Database [Internet]. Landes Bioscience, 2013. https://www.ncbi.nlm.nih.gov/books/NBK6616/.
Myeong, Jongyun, Cheon-Gyu Park, Byung-Chang Suh, and Bertil Hille. “Compartmentalization of Phosphatidylinositol 4,5-Bisphosphate Metabolism into Plasma Membrane Liquid-Ordered/Raft Domains.” Proceedings of the National Academy of Sciences 118, no. 9 (March 2, 2021): e2025343118. https://doi.org/10.1073/pnas.2025343118.
Taylor, David R., and Nigel M. Hooper. “Role of Lipid Rafts in the Processing of the Pathogenic Prion and Alzheimer’s Amyloid-β Proteins.” Seminars in Cell & Developmental Biology, Membrane Lipid Microdomains: Roles in Signalling and Disease and 3D Chromatin, 18, no. 5 (October 1, 2007): 638–48. https://doi.org/10.1016/j.semcdb.2007.07.008.
Mesa-Herrera, Fátima, Lucas Taoro-González, Catalina Valdés-Baizabal, Mario Diaz, and Raquel Marín. “Lipid and Lipid Raft Alteration in Aging and Neurodegenerative Diseases: A Window for the Development of New Biomarkers.” International Journal of Molecular Sciences 20, no. 15 (August 4, 2019): 3810. https://doi.org/10.3390/ijms20153810.
Bryndina, Irina G., Maria N. Shalagina, Vladimir A. Protopopov, Alexey V. Sekunov, Andrey L. Zefirov, Guzalia F. Zakirjanova, and Alexey M. Petrov. “Early Lipid Raft-Related Changes: Interplay between Unilateral Denervation and Hindlimb Suspension.” International Journal of Molecular Sciences 22, no. 5 (February 24, 2021): 2239. https://doi.org/10.3390/ijms22052239.
Gazitt, Tal, Tali Eviatar, Jacqueline Shear, Roni Meidan, Victoria Furer, Joy Feld, Amir Haddad, et al. “Development of Autoantibodies Following BNT162b2 mRNA COVID-19 Vaccination and Their Association with Disease Flares in Adult Patients with Autoimmune Inflammatory Rheumatic Diseases (AIIRD) and the General Population: Results of 1-Year Prospective Follow-Up Study.” Vaccines 11, no. 2 (February 17, 2023): 476. https://doi.org/10.3390/vaccines11020476.
Syrmou, Vasiliki, Christos Liaskos, Niki Ntavari, Konstantinos Mitsimponas, Theodora Simopoulou, Ioannis Alexiou, Marianna Vlychou, Christina G. Katsiari, and Dimitrios P. Bogdanos. “COVID-19 Vaccine-Associated Myositis: A Comprehensive Review of the Literature Driven by a Case Report.” Immunologic Research 71, no. 4 (2023): 537–46. https://doi.org/10.1007/s12026-023-09368-2.
Ning, Wangbin, Wanli Xu, Xiaomei Cong, Hongkuan Fan, Gary Gilkeson, Xueling Wu, Heather Hughes, and Wei Jiang. “COVID-19 mRNA Vaccine BNT162b2 Induces Autoantibodies against Type I Interferons in a Healthy Woman.” Journal of Autoimmunity 132 (October 2022): 102896. https://doi.org/10.1016/j.jaut.2022.102896.
“COVID-19 vaccine-associated myositis: a comprehensive review of the literature driven by a case report | springermedizin.de.” Accessed June 6, 2025. https://www.springermedicine.com/link?doi=10.1007/s12026-023-09368-2.
Klein, Christian R., Annkristin Heine, Peter Brossart, Pantelis Karakostas, and Valentin Sebastian Schäfer. “Anti-MDA5 Autoantibodies Predict Clinical Dynamics of Dermatomyositis Following SARS-CoV-2 mRNA Vaccination: A Retrospective Statistical Analysis of Case Reports.” Rheumatology International 44, no. 10 (October 1, 2024): 2185–96. https://doi.org/10.1007/s00296-024-05683-5.
Cinalli, Ryan M., Catherine E. Herman, Brian O. Lew, Heather L. Wieman, Craig B. Thompson, and Jeffrey C. Rathmell. “T Cell Homeostasis Requires G Protein-Coupled Receptor-Mediated Access to Trophic Signals That Promote Growth and Inhibit Chemotaxis.” European Journal of Immunology 35, no. 3 (March 2005): 786–95. https://doi.org/10.1002/eji.200425729.
Wang, Dashan. “The Essential Role of G Protein-Coupled Receptor (GPCR) Signaling in Regulating T Cell Immunity.” Immunopharmacology and Immunotoxicology 40, no. 3 (May 4, 2018): 187–92. https://doi.org/10.1080/08923973.2018.1434792.
Lu, Erick, and Jason G. Cyster. “G-Protein Coupled Receptors and Ligands That Organize Humoral Immune Responses.” Immunological Reviews 289, no. 1 (May 2019): 158–72. https://doi.org/10.1111/imr.12743.
Shao, Yongqi, Yang Mei, Yixin Tan, Ming Yang, and Haijing Wu. “The Regulatory Functions of G Protein-Coupled Receptors Signaling Pathways in B Cell Differentiation and Development Contributing to Autoimmune Diseases.” Cell & Bioscience 15, no. 1 (April 30, 2025): 57. https://doi.org/10.1186/s13578-025-01398-7.
Natarajan, Kanchana, and Bradford C. Berk. “Crosstalk Coregulation Mechanisms of G Protein-Coupled Receptors and Receptor Tyrosine Kinases.” Methods in Molecular Biology (Clifton, N.J.) 332 (2006): 51–77. https://doi.org/10.1385/1-59745-048-0:51.
Lattin, Jane, David A Zidar, Kate Schroder, Stuart Kellie, David A Hume, and Matthew J Sweet. “G-Protein-Coupled Receptor Expression, Function, and Signaling in Macrophages.” Journal of Leukocyte Biology 82, no. 1 (July 1, 2007): 16–32. https://doi.org/10.1189/jlb.0107051.
Loniewski, Katie, Yuhui Shi, James Pestka, and Narayanan Parameswaran. “Toll-like Receptors Differentially Regulate GPCR Kinases and Arrestins in Primary Macrophages.” Molecular Immunology 45, no. 8 (April 2008): 2312–22. https://doi.org/10.1016/j.molimm.2007.11.012.
Abdulkhalek, Samar, Merry Guo, Schammim Ray Amith, Preethi Jayanth, and Myron R. Szewczuk. “G-Protein Coupled Receptor Agonists Mediate Neu1 Sialidase and Matrix Metalloproteinase-9 Cross-Talk to Induce Transactivation of TOLL-like Receptors and Cellular Signaling.” Cellular Signalling 24, no. 11 (November 1, 2012): 2035–42. https://doi.org/10.1016/j.cellsig.2012.06.016.
Zhang, Peng, Meirong Yang, Chunhua Chen, Liu Liu, Xinchuan Wei, and Si Zeng. “Toll-Like Receptor 4 (TLR4)/Opioid Receptor Pathway Crosstalk and Impact on Opioid Analgesia, Immune Function, and Gastrointestinal Motility.” Frontiers in Immunology 11 (July 8, 2020). https://doi.org/10.3389/fimmu.2020.01455.
Lažetić, Vladimir, Lakshmi E. Batachari, Alistair B. Russell, and Emily R. Troemel. “Similarities in the Induction of the Intracellular Pathogen Response in Caenorhabditis Elegans and the Type I Interferon Response in Mammals.” BioEssays 45, no. 11 (November 2023): 2300097. https://doi.org/10.1002/bies.202300097.
Mira, Filipe S, Jóni Costa Carvalho, Patrícia Amaral De Almeida, Ana Carolina Pimenta, Iolanda Alen Coutinho, Carolina Figueiredo, Luís Rodrigues, et al. “A Case of Acute Interstitial Nephritis After Two Doses of the BNT162b2 SARS-CoV-2 Vaccine.” International Journal of Nephrology and Renovascular Disease Volume 14 (December 2021): 421–26. https://doi.org/10.2147/IJNRD.S345898.
Santovito, Luca Spiro, and Graziano Pinna. “Acute Reduction of Visual Acuity and Visual Field after Pfizer-BioNTech COVID-19 Vaccine 2nd Dose: A Case Report.” Inflammation Research 70, no. 9 (September 1, 2021): 931–33. https://doi.org/10.1007/s00011-021-01476-9.
Autjimanon, Hathairat, Pongsakorn Champueng, Ritthideach Yorsaeng, and Yong Poovorawan. “ACUTE ENCEPHALOPATHY AFTER BNT162B2 COVID-19 VACCINATION: A CASE REPORT AND LITERATURE REVIEW: Acute Encephalopathy after mRNA COVID-19 Vaccine.” The Southeast Asian Journal of Tropical Medicine and Public Health 53, no. 3 (May 19, 2022): 217–31.
Grunenwald, Solange, Gabriel Lethellier, Philippe Imbert, Céline Dekeister, and Philippe Caron. “Orbital Inflammatory Disease Following mRNA SARS‐CoV‐2 Vaccine.” Clinical Case Reports 10, no. 6 (June 2022): e05926. https://doi.org/10.1002/ccr3.5926.
Báez-Negrón, Laisha, and Luis M. Vilá. “New-Onset Systemic Lupus Erythematosus after mRNA SARS-CoV-2 Vaccination.” Edited by Gregory J. Tsay. Case Reports in Rheumatology 2022 (February 11, 2022): 1–4. https://doi.org/10.1155/2022/6436839.
Adelmeyer, Jan, Joachim Nils Goebel, Alexander Kauka, and Peter Herbert Kann. “Two Case Reports of Subacute Thyroiditis after Receiving Vaccine for COVID-19.” Edited by J. Paul Frindik. Case Reports in Endocrinology 2022 (April 14, 2022): 1–9. https://doi.org/10.1155/2022/3180004.
Duran, M. “Bilateral Anterior Uveitis after BNT162b2 mRNA Vaccine: Case Report.” Journal Francais D’Ophtalmologie 45, no. 7 (September 2022): e311–13. https://doi.org/10.1016/j.jfo.2022.04.004.
Sagy, Iftach, Lior Zeller, Yael Raviv, Tzvika Porges, Amir Bieber, and Mahmoud Abu-Shakra. “New-Onset Systemic Lupus Erythematosus Following BNT162b2 mRNA COVID-19 Vaccine: A Case Series and Literature Review.” Rheumatology International 42, no. 12 (December 1, 2022): 2261–66. https://doi.org/10.1007/s00296-022-05203-3.
Raviv, Yael, Batya Betesh-Abay, Yuliya Valdman-Grinshpoun, Liora Boehm-Cohen, Michael Kassirer, and Iftach Sagy. “First Presentation of Systemic Lupus Erythematosus in a 24-Year-Old Male Following mRNA COVID-19 Vaccine.” Edited by Tsai Ching Hsu. Case Reports in Rheumatology 2022 (February 1, 2022): 1–4. https://doi.org/10.1155/2022/9698138.
Khanna, Urmi, Yasmine Oprea, Adnan Mir, and Caroline Halverstam. “New Diagnosis of Systemic Lupus Erythematosus after COVID-19 Vaccination: A Case Report and Review of Literature.” JAAD Case Reports 30 (December 1, 2022): 30–34. https://doi.org/10.1016/j.jdcr.2022.09.026.
Pattonieri, Eleonora Francesca, Marilena Gregorini, Maria Antonietta Grignano, Tefik Islami, Gioacchino D’Ambrosio, Gianluigi Ardissino, and Teresa Rampino. “Atypical Hemolytic Uremic Syndrome Associated with BNT162b2 mRNA COVID-19 Vaccine in a Kidney Transplant Recipient: A Case Report and Literature Review.” Infectious Disease Reports 17, no. 1 (February 11, 2025): 14. https://doi.org/10.3390/idr17010014.
Lemoine, Chantal, Cristina Padilla, Noah Krampe, Sean Doerfler, Adam Morgenlander, Brent Thiel, and Rohit Aggarwal. “Systemic Lupus Erythematous after Pfizer COVID-19 Vaccine: A Case Report.” Clinical Rheumatology 41, no. 5 (May 1, 2022): 1597–1601. https://doi.org/10.1007/s10067-022-06126-x.
Kim, Ji Hyoun, Hee Bok Chae, Sumin Woo, Myung Seop Song, Hyo-Jong Kim, and Chang Gok Woo. “Clinicopathological Characteristics of Autoimmune-Like Hepatitis Induced by COVID-19 mRNA Vaccine (Pfizer-BioNTech, BNT162b2): A Case Report and Literature Review.” International Journal of Surgical Pathology 31, no. 6 (September 2023): 1156–62. https://doi.org/10.1177/10668969231177877.
Moriyama, Masayori, Kentaro Noda, Haruyasu Ito, Takayuki Matsushita, and Daitaro Kurosaka. “Clinical Features of Newly Diagnosed Systemic Lupus Erythematosus after SARS-CoV-2 Vaccination.” Modern Rheumatology Case Reports 8, no. 1 (December 29, 2023): 63–68. https://doi.org/10.1093/mrcr/rxad036.
Huang, Lanhui, Min Liang, and Yuling He. “New-Onset Fulminant Type 1 Diabetes Following SARS-CoV-2 Protein Subunit Vaccine: A Case Report and Literature Review.” Journal of Korean Medical Science 38, no. 24 (May 12, 2023). https://doi.org/10.3346/jkms.2023.38.e209.
Pattonieri, Eleonora Francesca, Marilena Gregorini, Maria Antonietta Grignano, Tefik Islami, Gioacchino D’Ambrosio, Gianluigi Ardissino, and Teresa Rampino. “Atypical Hemolytic Uremic Syndrome Associated with BNT162b2 mRNA COVID-19 Vaccine in a Kidney Transplant Recipient: A Case Report and Literature Review.” Infectious Disease Reports 17, no. 1 (February 11, 2025): 14. https://doi.org/10.3390/idr17010014.
Iwamoto, Hideyuki, Tomohiko Kimura, Erina Nakao, Fuminori Tatsumi, Hideaki Kaneto, Hideyuki Iwamoto, Tomohiko Kimura, Erina Nakao, Fuminori Tatsumi, and HIdeaki Kaneto. “A Case of Graves’ Disease Exacerbation Triggered by BNT162b2 Vaccination.” Cureus 17, no. 5 (May 26, 2025). https://doi.org/10.7759/cureus.84863.
Mingot-Castellano, María Eva, Nora Butta, Mariana Canaro, María del Carmen Gómez del Castillo Solano, Blanca Sánchez-González, Reyes Jiménez-Bárcenas, Cristina Pascual-Izquierdo, Gonzalo Caballero-Navarro, Laura Entrena Ureña, and Tomás José González-López. “COVID-19 Vaccines and Autoimmune Hematologic Disorders.” Vaccines 10, no. 6 (June 16, 2022): 961. https://doi.org/10.3390/vaccines10060961.
Fallahi-Sichani, Mohammad, and Jennifer J. Linderman. “Lipid Raft-Mediated Regulation of G-Protein Coupled Receptor Signaling by Ligands Which Influence Receptor Dimerization: A Computational Study.” PLoS ONE 4, no. 8 (August 11, 2009): e6604. https://doi.org/10.1371/journal.pone.0006604.
Wang, Xinxin, Xin Shi, and Ruifeng Wang. “Regulating mRNA Endosomal Escape through Lipid Rafts: A Review.” International Journal of Pharmaceutics 675 (April 30, 2025): 125571. https://doi.org/10.1016/j.ijpharm.2025.125571.
Cabral-marque, Otavio, Alexandre henrique Marques, Lena F. Schimke, Harald Heidecke, and Gabriela Riemekasten. “Loss of Balance in Normal GPCR-Mediated Cell Trafficking.” Frontiers in Bioscience-Landmark 24, no. 1 (January 1, 2019): 18–32. https://doi.org/10.2741/4707.
Yalcinkaya, Ahmet, Marco Cavalli, Axel Cederholm, Maribel Aranda-Guillén, Anish Behere, Hedvig Mildner, Tadepally Lakshmikanth, et al. “No Link between Type I Interferon Autoantibody Positivity and Adverse Reactions to COVID-19 Vaccines.” Npj Vaccines 9, no. 1 (February 22, 2024): 1–6. https://doi.org/10.1038/s41541-024-00829-9.
Bastard, Paul, Lindsey B. Rosen, Qian Zhang, Eleftherios Michailidis, Hans-Heinrich Hoffmann, Yu Zhang, Karim Dorgham, et al. “Autoantibodies against Type I IFNs in Patients with Life-Threatening COVID-19.” Science (New York, N.y.) 370, no. 6515 (October 23, 2020): eabd4585. https://doi.org/10.1126/science.abd4585.
Gazitt, Tal, Tali Eviatar, Jacqueline Shear, Roni Meidan, Victoria Furer, Joy Feld, Amir Haddad, et al. “Development of Autoantibodies Following BNT162b2 mRNA COVID-19 Vaccination and Their Association with Disease Flares in Adult Patients with Autoimmune Inflammatory Rheumatic Diseases (AIIRD) and the General Population: Results of 1-Year Prospective Follow-Up Study.” Vaccines 11, no. 2 (February 17, 2023): 476. https://doi.org/10.3390/vaccines11020476.
Ludwig, Ralf J., Karen Vanhoorelbeke, Frank Leypoldt, Ziya Kaya, Katja Bieber, Sandra M. McLachlan, Lars Komorowski, et al. “Mechanisms of Autoantibody-Induced Pathology.” Frontiers in Immunology 8 (May 31, 2017): 603. https://doi.org/10.3389/fimmu.2017.00603.
Johnson, Douglas, and Wei Jiang. “Infectious Diseases, Autoantibodies, and Autoimmunity.” Journal of Autoimmunity 137 (May 2023): 102962. https://doi.org/10.1016/j.jaut.2022.102962.
Song, Yi, Jian Li, and Yuzhang Wu. “Evolving Understanding of Autoimmune Mechanisms and New Therapeutic Strategies of Autoimmune Disorders.” Signal Transduction and Targeted Therapy 9, no. 1 (October 4, 2024): 1–40. https://doi.org/10.1038/s41392-024-01952-8.
Khan, Farooq, and Mary Jane Brassill. “Subacute Thyroiditis Post-Pfizer-BioNTech mRNA Vaccination for COVID-19.” Endocrinology, Diabetes & Metabolism Case Reports 2021 (December 1, 2021). https://doi.org/10.1530/EDM-21-0142.
Liozon, Eric, Matthieu Filloux, Simon Parreau, Guillaume Gondran, Holy Bezanahary, Kim-Heang Ly, and Anne-Laure Fauchais. “Immune-Mediated Diseases Following COVID-19 Vaccination: Report of a Teaching Hospital-Based Case-Series.” Journal of Clinical Medicine 11, no. 24 (December 16, 2022): 7484. https://doi.org/10.3390/jcm11247484.
Tan, Elina, and Sam Salman. “Unusual Case of Painful Glossitis and Xerostomia Following Vaccination with Pfizer-BioNTech SARS-CoV-2 (BNT162b2).” American Journal of Case Reports 23 (July 8, 2022). https://doi.org/10.12659/AJCR.937212.
Tseng, Chia-Wei, Jing-Huan Liao, Tai-Kuang Chao, and Shun-Neng Hsu. “New Onset of Primary Membranous Nephropathy After COVID-19 mRNA Vaccination in Affected Sjögren’s Syndrome.” Nephrology (Carlton, Vic.) 30, no. 5 (May 2025): e70048. https://doi.org/10.1111/nep.70048.
Komori, Yuta, Satomi Kobayashi, Keiko Hatano, Yuko Saito, Tomio Arai, and Kanae Kubo. “Elderly-Onset Inflammatory Myopathy Associated with Sjögren’s Syndrome Following SARS-CoV-2 Vaccination.” Modern Rheumatology Case Reports 9, no. 1 (January 16, 2025): 104–9. https://doi.org/10.1093/mrcr/rxae055.
Xue, Chaoyang, Yen-Ping Hsueh, and Joseph Heitman. “Magnificent Seven: Roles of G Protein-Coupled Receptors in Extracellular Sensing in Fungi.” FEMS Microbiology Reviews 32, no. 6 (November 2008): 1010–32. https://doi.org/10.1111/j.1574-6976.2008.00131.x.
Liu, Samuel, Preston J. Anderson, Sudarshan Rajagopal, Robert J. Lefkowitz, and Howard A. Rockman. “G Protein-Coupled Receptors: A Century of Research and Discovery.” Circulation Research 135, no. 1 (June 21, 2024): 174–97. https://doi.org/10.1161/CIRCRESAHA.124.323067.
Verweij, Eléonore W. E., Betty Al Araaj, Wimzy R. Prabhata, Rudi Prihandoko, Saskia Nijmeijer, Andrew B. Tobin, Rob Leurs, and Henry F. Vischer. “Differential Role of Serines and Threonines in Intracellular Loop 3 and C-Terminal Tail of the Histamine H4 Receptor in β-Arrestin and G Protein-Coupled Receptor Kinase Interaction, Internalization, and Signaling.” ACS Pharmacology & Translational Science 3, no. 2 (April 10, 2020): 321–33. https://doi.org/10.1021/acsptsci.0c00008.
Latorraca, Naomi R., Matthieu Masureel, Scott A. Hollingsworth, Franziska M. Heydenreich, Carl-Mikael Suomivuori, Connor Brinton, Raphael J. L. Townshend, Michel Bouvier, Brian K. Kobilka, and Ron O. Dror. “How GPCR Phosphorylation Patterns Orchestrate Arrestin-Mediated Signaling.” Cell 183, no. 7 (December 23, 2020): 1813-1825.e18. https://doi.org/10.1016/j.cell.2020.11.014.
Rosenbaum, Daniel M., Søren G. F. Rasmussen, and Brian K. Kobilka. “The Structure and Function of G-Protein-Coupled Receptors.” Nature 459, no. 7245 (May 21, 2009): 356–63. https://doi.org/10.1038/nature08144.
Baker, Jillian G., and Stephen J. Hill. “Multiple GPCR Conformations and Signalling Pathways: Implications for Antagonist Affinity Estimates.” Trends in Pharmacological Sciences 28, no. 8 (August 2007): 374–81. https://doi.org/10.1016/j.tips.2007.06.011.
Zhang, Mingyang, Ting Chen, Xun Lu, Xiaobing Lan, Ziqiang Chen, and Shaoyong Lu. “G Protein-Coupled Receptors (GPCRs): Advances in Structures, Mechanisms and Drug Discovery.” Signal Transduction and Targeted Therapy 9, no. 1 (April 10, 2024): 1–43. https://doi.org/10.1038/s41392-024-01803-6.
Gavi, Shai, Elena Shumay, Hsien-yu Wang, and Craig C Malbon. “G-Protein-Coupled Receptors and Tyrosine Kinases: Crossroads in Cell Signaling and Regulation.” Trends in Endocrinology & Metabolism 17, no. 2 (March 2006): 48–54. https://doi.org/10.1016/j.tem.2006.01.006.
Budd, David C., Angela Rae, and Andrew B. Tobin. “Activation of the Mitogen-Activated Protein Kinase Pathway by a Gq/11-Coupled Muscarinic Receptor Is Independent of Receptor Internalization*.” Journal of Biological Chemistry 274, no. 18 (April 30, 1999): 12355–60. https://doi.org/10.1074/jbc.274.18.12355.
Naor, Zvi, Outhiriaradjou Benard, and Rony Seger. “Activation of MAPK Cascades by G-Protein-Coupled Receptors: The Case of Gonadotropin-Releasing Hormone Receptor.” Trends in Endocrinology & Metabolism 11, no. 3 (April 1, 2000): 91–99. https://doi.org/10.1016/S1043-2760(99)00232-5.
Sorkin, Alexander, and Mark von Zastrow. “Endocytosis and Signalling: Intertwining Molecular Networks.” Nature Reviews. Molecular Cell Biology 10, no. 9 (September 2009): 609–22. https://doi.org/10.1038/nrm2748.
Shin, Sung-Young, Oliver Rath, Sang-Mok Choo, Frances Fee, Brian McFerran, Walter Kolch, and Kwang-Hyun Cho. “Positive- and Negative-Feedback Regulations Coordinate the Dynamic Behavior of the Ras-Raf-MEK-ERK Signal Transduction Pathway.” Journal of Cell Science 122, no. 3 (February 1, 2009): 425–35. https://doi.org/10.1242/jcs.036319.
Jubaidi, Fatin Farhana, Satirah Zainalabidin, Izatus Shima Taib, Zariyantey Abdul Hamid, Nur Najmi Mohamad Anuar, Juriyati Jalil, Nor Anizah Mohd Nor, and Siti Balkis Budin. “The Role of PKC-MAPK Signalling Pathways in the Development of Hyperglycemia-Induced Cardiovascular Complications.” International Journal of Molecular Sciences 23, no. 15 (August 2, 2022): 8582. https://doi.org/10.3390/ijms23158582.
Yan, Yaping, Tengkun Dai, Mengmeng Guo, Xu Zhao, Chao Chen, Ya Zhou, Ming Qin, Lin Xu, and Juanjuan Zhao. “A Review of Non-Classical MAPK Family Member, MAPK4: A Pivotal Player in Cancer Development and Therapeutic Intervention.” International Journal of Biological Macromolecules 271 (June 1, 2024): 132686. https://doi.org/10.1016/j.ijbiomac.2024.132686.
Ng, Gavin Yong-Quan, Zachary Wai-Loon Loh, David Y. Fann, Karthik Mallilankaraman, Thiruma V. Arumugam, and M. Prakash Hande. “Role of Mitogen-Activated Protein (MAP) Kinase Pathways in Metabolic Diseases.” Genome Integrity 15 (January 17, 2024): 20230003. https://doi.org/10.14293/genint.14.1.004.
Falk, Martin, Michael Klann, Matthias Reuss, and Thomas Ertl. “Visualization of Signal Transduction Processes in the Crowded Environment of the Cell.” In 2009 IEEE Pacific Visualization Symposium, 169–76, 2009. https://doi.org/10.1109/PACIFICVIS.2009.4906853.
Nadal-Ribelles, Mariona, Carme Solé, Gerard Martínez-Cebrián, Francesc Posas, and Eulàlia de Nadal. “Shaping the Transcriptional Landscape through MAPK Signaling.” In Gene Expression and Control. IntechOpen, 2018. https://doi.org/10.5772/intechopen.80634.
Avraham, Roi, and Yosef Yarden. “Feedback Regulation of EGFR Signalling: Decision Making by Early and Delayed Loops.” Nature Reviews Molecular Cell Biology 12, no. 2 (February 2011): 104–17. https://doi.org/10.1038/nrm3048.
Sellegounder, Durai, Luigi Ferrucci, Rajakumar Anbazhagan, and Nathan Basisty. “Editorial: Molecular Crosstalk between Endocrine Factors, Paracrine Signals, and the Immune System during Aging.” Frontiers in Endocrinology 14 (April 21, 2023): 1203755. https://doi.org/10.3389/fendo.2023.1203755.
Posor, York, Wonyul Jang, and Volker Haucke. “Phosphoinositides as Membrane Organizers.” Nature Reviews. Molecular Cell Biology 23, no. 12 (2022): 797–816. https://doi.org/10.1038/s41580-022-00490-x.
Lolicato, Fabio, Walter Nickel, Volker Haucke, and Michael Ebner. “Phosphoinositide Switches in Cell Physiology - From Molecular Mechanisms to Disease.” Journal of Biological Chemistry 300, no. 3 (March 2024): 105757. https://doi.org/10.1016/j.jbc.2024.105757.
Erijman, Ariel, and Julia M. Shifman. “RAS/Effector Interactions from Structural and Biophysical Perspective.” Mini-Reviews in Medicinal Chemistry 16, no. 5 (n.d.): 370–75. https://doi.org/10.2174/1389557515666151001141838.
Aksamitiene, Edita, Anatoly Kiyatkin, and Boris N. Kholodenko. “Cross-Talk between Mitogenic Ras/MAPK and Survival PI3K/Akt Pathways: A Fine Balance.” Biochemical Society Transactions 40, no. 1 (February 2012): 139–46. https://doi.org/10.1042/BST20110609.
Fleming, Ian R., Jonathan P. Hannan, George Hayden Swisher, Corey D. Tesdahl, Justin G. Martyr, Nicholas J. Cordaro, Annette H. Erbse, and Joseph J. Falke. “Binding of Active Ras and Its Mutants to the Ras Binding Domain of PI-3-Kinase: A Quantitative Approach to KD Measurements.” Analytical Biochemistry 663 (February 15, 2023): 115019. https://doi.org/10.1016/j.ab.2022.115019.
Omo-Lamai, Serena, Yufei Wang, Manthan N. Patel, Eno-Obong Essien, Mengwen Shen, Aparajeeta Majumdar, Carolann Espy, et al. “Lipid Nanoparticle-Associated Inflammation Is Triggered by Sensing of Endosomal Damage: Engineering Endosomal Escape Without Side Effects.” bioRxiv, April 18, 2024. https://doi.org/10.1101/2024.04.16.589801.
Feng, Yawen, Hongying Fu, Xing Zhang, Suqin Liu, and Xiaoran Wei. “Lysosome Toxicities Induced by Nanoparticle Exposure and Related Mechanisms.” Ecotoxicology and Environmental Safety 286 (November 1, 2024): 117215. https://doi.org/10.1016/j.ecoenv.2024.117215.
Ndeupen, Sonia, Zhen Qin, Sonya Jacobsen, Aurélie Bouteau, Henri Estanbouli, and Botond Z. Igyártó. “The mRNA-LNP Platform’s Lipid Nanoparticle Component Used in Preclinical Vaccine Studies Is Highly Inflammatory.” iScience 24, no. 12 (December 2021): 103479. https://doi.org/10.1016/j.isci.2021.103479.
Yuan, Jimin, Xiaoduo Dong, Jiajun Yap, and Jiancheng Hu. “The MAPK and AMPK Signalings: Interplay and Implication in Targeted Cancer Therapy.” Journal of Hematology & Oncology 13, no. 1 (August 17, 2020): 113. https://doi.org/10.1186/s13045-020-00949-4.
Wang, Xinyue, Xiao Tan, Jinping Zhang, Jiaping Wu, and Hongjuan Shi. “The Emerging Roles of MAPK-AMPK in Ferroptosis Regulatory Network.” Cell Communication and Signaling 21, no. 1 (August 14, 2023): 200. https://doi.org/10.1186/s12964-023-01170-9.
Meng, Rongsen, Zhaohui Pei, Aixia Zhang, Yutian Zhou, Xingming Cai, Baolin Chen, Guanjie Liu, Weiyi Mai, Jianrui Wei, and Yugang Dong. “AMPK Activation Enhances PPARα Activity to Inhibit Cardiac Hypertrophy via ERK1/2 MAPK Signaling Pathway.” Archives of Biochemistry and Biophysics 511, no. 1 (July 1, 2011): 1–7. https://doi.org/10.1016/j.abb.2011.04.010.
Yin, Ran, Yu-gang Dong, and Hong-liang Li. “PPAR? Phosphorylation Mediated by JNK MAPK: A Potential Role in Mac-Rophage-Derived Foam Cell Formation.” Acta Pharmacologica Sinica 27, no. 9 (September 2006): 1146–52. https://doi.org/10.1111/j.1745-7254.2006.00359.x.
Tahtinen, Siri, Ann-Jay Tong, Patricia Himmels, Jaehak Oh, Andres Paler-Martinez, Leesun Kim, Sara Wichner, et al. “IL-1 and IL-1ra Are Key Regulators of the Inflammatory Response to RNA Vaccines.” Nature Immunology 23, no. 4 (April 2022): 532–42. https://doi.org/10.1038/s41590-022-01160-y.
Forster Iii, James, Dipika Nandi, and Ashish Kulkarni. “mRNA-Carrying Lipid Nanoparticles That Induce Lysosomal Rupture Activate NLRP3 Inflammasome and Reduce mRNA Transfection Efficiency.” Biomaterials Science 10, no. 19 (September 27, 2022): 5566–82. https://doi.org/10.1039/d2bm00883a.
Blevins, Hallie M., Yiming Xu, Savannah Biby, and Shijun Zhang. “The NLRP3 Inflammasome Pathway: A Review of Mechanisms and Inhibitors for the Treatment of Inflammatory Diseases.” Frontiers in Aging Neuroscience 14 (June 10, 2022). https://doi.org/10.3389/fnagi.2022.879021.
Zelkoski, Amanda E., Zhongyan Lu, Gauthaman Sukumar, Clifton Dalgard, Hooda Said, Mohamad-Gabriel Alameh, Edward Mitre, and Allison M. W. Malloy. “Ionizable Lipid Nanoparticles of mRNA Vaccines Elicit NF-κB and IRF Responses through Toll-like Receptor 4.” Npj Vaccines 10, no. 1 (April 17, 2025): 1–13. https://doi.org/10.1038/s41541-025-01124-x.
Knabl, Ludwig, Hye Kyung Lee, Manuel Wieser, Anna Mur, August Zabernigg, Ludwig Knabl, Simon Rauch, et al. “BNT162b2 Vaccination Enhances Interferon-JAK-STAT-Regulated Antiviral Programs in COVID-19 Patients Infected with the SARS-CoV-2 Beta Variant.” Communications Medicine 2 (February 22, 2022): 17. https://doi.org/10.1038/s43856-022-00083-x.
Donovan, Jesse, Matthew Dufner, and Alexei Korennykh. “Structural Basis for Cytosolic Double-Stranded RNA Surveillance by Human Oligoadenylate Synthetase 1.” Proceedings of the National Academy of Sciences of the United States of America 110, no. 5 (January 29, 2013): 1652–57. https://doi.org/10.1073/pnas.1218528110.
Schwartz, Samantha L., Debayan Dey, Julia Tanquary, Camden R. Bair, Anice C. Lowen, and Graeme L. Conn. “Role of Helical Structure and Dynamics in Oligoadenylate Synthetase 1 (OAS1) Mismatch Tolerance and Activation by Short dsRNAs.” Proceedings of the National Academy of Sciences 119, no. 3 (January 18, 2022): e2107111119. https://doi.org/10.1073/pnas.2107111119.
Merold, Veronika, Indra Bekere, Stefanie Kretschmer, Adrian F. Schnell, Dorota Kmiec, Rinu Sivarajan, Katja Lammens, et al. “Structural Basis for OAS2 Regulation and Its Antiviral Function.” Molecular Cell 85, no. 11 (June 5, 2025): 2176-2193.e13. https://doi.org/10.1016/j.molcel.2025.05.001.
Dibble, Christian C., and Lewis C. Cantley. “Regulation of mTORC1 by PI3K Signaling.” Trends in Cell Biology 25, no. 9 (September 2015): 545–55. https://doi.org/10.1016/j.tcb.2015.06.002.
Martelli, Alberto M., Camilla Evangelisti, Francesca Chiarini, and James A. McCubrey. “The Phosphatidylinositol 3-Kinase/Akt/mTOR Signaling Network as a Therapeutic Target in Acute Myelogenous Leukemia Patients.” Oncotarget 1, no. 2 (May 27, 2010): 89–103. https://doi.org/10.18632/oncotarget.114.
Ezine, Emilien, Céleste Lebbe, and Nicolas Dumaz. “Unmasking the Tumourigenic Role of SIN1/MAPKAP1 in the mTOR Complex 2.” Clinical and Translational Medicine 13, no. 10 (October 2023): e1464. https://doi.org/10.1002/ctm2.1464.
Hickey, Thomas E., Uma Mudunuri, Heidi A. Hempel, Troy J. Kemp, Nancy V. Roche, Keyur Talsania, Brian A. Sellers, James M. Cherry, and Ligia A. Pinto. “Proteomic and Serologic Assessments of Responses to mRNA-1273 and BNT162b2 Vaccines in Human Recipient Sera.” Frontiers in Immunology 15 (January 27, 2025): 1502458. https://doi.org/10.3389/fimmu.2024.1502458.
Savelsbergh, Andreas, Dagmar Mohr, Berthold Wilden, Wolfgang Wintermeyer, and Marina V. Rodnina. “Stimulation of the GTPase Activity of Translation Elongation Factor G by Ribosomal Protein L7/12*.” Journal of Biological Chemistry 275, no. 2 (January 14, 2000): 890–94. https://doi.org/10.1074/jbc.275.2.890.
Gulluni, Federico, Miriam Martini, and Emilio Hirsch. “Cytokinetic Abscission: Phosphoinositides and ESCRTs Direct the Final Cut.” Journal of Cellular Biochemistry 118, no. 11 (November 2017): 3561–68. https://doi.org/10.1002/jcb.26066.
Tan, Xiaojun, Narendra Thapa, Suyong Choi, and Richard A. Anderson. “Emerging Roles of PtdIns(4,5) P 2 – beyond the Plasma Membrane.” Journal of Cell Science 128, no. 22 (November 15, 2015): 4047–56. https://doi.org/10.1242/jcs.175208.
Ajazi, Arta, Christopher Bruhn, Ghadeer Shubassi, Chiara Lucca, Elisa Ferrari, Angela Cattaneo, Angela Bachi, et al. “Endosomal Trafficking and DNA Damage Checkpoint Kinases Dictate Survival to Replication Stress by Regulating Amino Acid Uptake and Protein Synthesis.” Developmental Cell 56, no. 18 (September 27, 2021): 2607-2622.e6. https://doi.org/10.1016/j.devcel.2021.08.019.
Waters, Maxine, Mare Vlok, Elouise E. Kroon, Maritha J. Kotze, Kelebogile E. Moremi, Sunday O. Oladejo, Kanshukan Rajartnam, et al. “Proteomic Signatures of Post-Vaccination/Post-Infection Syndrome (PV/PIS): Insights into Immune Dysregulation and Coagulopathy,” May 5, 2025. https://doi.org/10.21203/rs.3.rs-6521005/v1.
Dambal, Shweta, Mahmoud Alfaqih, Sergio Sanders, Erick Maravilla, Adela Ramirez-Torres, Gloria C. Galvan, Mariana Reis-Sobreiro, et al. “27-Hydroxycholesterol Impairs Plasma Membrane Lipid Raft Signaling as Evidenced by Inhibition of IL6-JAK-STAT3 Signaling in Prostate Cancer Cells.” Molecular Cancer Research: MCR 18, no. 5 (May 2020): 671–84. https://doi.org/10.1158/1541-7786.MCR-19-0974.
Miao, Hui, Timothy E. Vanderleest, Rashmi Budhathoki, Dinah Loerke, and J. Todd Blankenship. “A PtdIns(3,4,5)P3 Dispersal Switch Engages Cell Ratcheting at Specific Cell Surfaces.” Developmental Cell 56, no. 18 (September 27, 2021): 2579-2591.e4. https://doi.org/10.1016/j.devcel.2021.08.016.
Tse, Carmen, Ashleigh Warner, Rufaik Farook, and James G Cronin. “Phytochemical Targeting of STAT3 Orchestrated Lipid Metabolism in Therapy-Resistant Cancers.” Biomolecules 10, no. 8 (July 28, 2020): 1118. https://doi.org/10.3390/biom10081118.
Winston, Linda A, and Tony Hunter. “Intracellular Signalling: Putting JAKs on the Kinase MAP.” Current Biology 6, no. 6 (June 1, 1996): 668–71. https://doi.org/10.1016/S0960-9822(09)00445-X.
Mukherjee, Amartya, Chaithra Anoop, and Upendra Nongthomba. “What a Tangled Web We Weave: Crosstalk between JAK – STAT and Other Signalling Pathways during Development in Drosophila.” The FEBS Journal, January 16, 2025, febs.17391. https://doi.org/10.1111/febs.17391.
Wu, Qinghua, Xu Wang, Dan Wan, Juan Li, and Zonghui Yuan. “Crosstalk of JNK1-STAT3 Is Critical for RAW264.7 Cell Survival.” Cellular Signalling 26, no. 12 (December 1, 2014): 2951–60. https://doi.org/10.1016/j.cellsig.2014.09.013.
Gupta, Sahil, Arpit Jain, Shahzad Nawaz Syed, Ryan G. Snodgrass, Beatrice Pflüger-Müller, Matthias S. Leisegang, Andreas Weigert, et al. “IL-6 Augments IL-4-Induced Polarization of Primary Human Macrophages through Synergy of STAT3, STAT6 and BATF Transcription Factors.” OncoImmunology 7, no. 10 (October 3, 2018): e1494110. https://doi.org/10.1080/2162402X.2018.1494110.
Kastner, Lauren, David Dwyer, and F Xiao-Feng Qin. “Synergistic Effect of IL-6 and IL-4 in Driving Fate Revision of Natural Foxp3+ Regulatory T Cells.” The Journal of Immunology 185, no. 10 (November 15, 2010): 5778–86. https://doi.org/10.4049/jimmunol.0901948.
Francis, Vanessa I., and Steven L. Porter. “Multikinase Networks: Two-Component Signaling Networks Integrating Multiple Stimuli.” Annual Review of Microbiology 73 (September 8, 2019): 199–223. https://doi.org/10.1146/annurev-micro-020518-115846.
Sabaawy, Hatem E, Bríd M Ryan, Hossein Khiabanian, and Sharon R Pine. “JAK/STAT of All Trades: Linking Inflammation with Cancer Development, Tumor Progression and Therapy Resistance.” Carcinogenesis 42, no. 12 (August 20, 2021): 1411–19. https://doi.org/10.1093/carcin/bgab075.
Wang, Yuxin, Anette H. H. Van Boxel-Dezaire, HyeonJoo Cheon, Jinbo Yang, and George R. Stark. “STAT3 Activation in Response to IL-6 Is Prolonged by the Binding of IL-6 Receptor to EGF Receptor.” Proceedings of the National Academy of Sciences 110, no. 42 (October 15, 2013): 16975–80. https://doi.org/10.1073/pnas.1315862110.
Liu, Fusheng, Hou Wu, Xiaoyu Yang, Yuqin Dong, Guoyou Huang, Guy M. Genin, Tian Jian Lu, and Feng Xu. “A New Model of Myofibroblast-Cardiomyocyte Interactions and Their Differences across Species.” Biophysical Journal 120, no. 17 (September 7, 2021): 3764–75. https://doi.org/10.1016/j.bpj.2021.06.040.
Hendrayani, Siti-Fauziah, Bothaina Al-Harbi, Mysoon M. Al-Ansari, Gabriela Silva, and Abdelilah Aboussekhra. “The Inflammatory/Cancer-Related IL-6/STAT3/NF-κB Positive Feedback Loop Includes AUF1 and Maintains the Active State of Breast Myofibroblasts.” Oncotarget 7, no. 27 (May 26, 2016): 41974–85. https://doi.org/10.18632/oncotarget.9633.
Hu, Zhengyang, Qihai Sui, Xing Jin, Guangyao Shan, Yiwei Huang, Yanjun Yi, Dejun Zeng, et al. “IL6-STAT3-C/EBPβ-IL6 Positive Feedback Loop in Tumor-Associated Macrophages Promotes the EMT and Metastasis of Lung Adenocarcinoma.” Journal of Experimental & Clinical Cancer Research : CR 43 (February 29, 2024): 63. https://doi.org/10.1186/s13046-024-02989-x.
Libermann, T. A., and D. Baltimore. “Activation of Interleukin-6 Gene Expression through the NF-Kappa B Transcription Factor.” Molecular and Cellular Biology 10, no. 5 (May 1990): 2327–34. https://doi.org/10.1128/mcb.10.5.2327-2334.1990.
McLoughlin, Rachel M., Brendan J. Jenkins, Dianne Grail, Anwen S. Williams, Ceri A. Fielding, Clare R. Parker, Matthias Ernst, Nicholas Topley, and Simon A. Jones. “IL-6 Trans-Signaling via STAT3 Directs T Cell Infiltration in Acute Inflammation.” Proceedings of the National Academy of Sciences 102, no. 27 (July 5, 2005): 9589–94. https://doi.org/10.1073/pnas.0501794102.
Klemm, Carolin, Christin Bruchhagen, Andre van Krüchten, Silke Niemann, Bettina Löffler, Georg Peters, Stephan Ludwig, and Christina Ehrhardt. “Mitogen-Activated Protein Kinases (MAPKs) Regulate IL-6 over-Production during Concomitant Influenza Virus and Staphylococcus Aureus Infection.” Scientific Reports 7, no. 1 (February 14, 2017): 42473. https://doi.org/10.1038/srep42473.
Costa-Pereira, Ana P. “Regulation of IL-6-Type Cytokine Responses by MAPKs.” Biochemical Society Transactions 42, no. 1 (February 2014): 59–62. https://doi.org/10.1042/BST20130267.
Nyati, Kishan K., Kazuya Masuda, Mohammad Mahabub-Uz Zaman, Praveen K. Dubey, David Millrine, Jaya P. Chalise, Mitsuru Higa, et al. “TLR4-Induced NF-κB and MAPK Signaling Regulate the IL-6 mRNA Stabilizing Protein Arid5a.” Nucleic Acids Research 45, no. 5 (March 17, 2017): 2687–2703. https://doi.org/10.1093/nar/gkx064.
Bian, Wenjie, Yingni Li, Feng Sun, Xiaolin Sun, Ru Li, Changsheng Xia, Jiangnan Fu, Yuxin Zhang, Shuang Chen, and Yanying Liu. “Immune Phenotype Changes in IgG4-Related Disease: CD161 + Treg and Foxp3 + Treg.” Clinical Rheumatology 42, no. 4 (April 2023): 1113–24. https://doi.org/10.1007/s10067-022-06445-z.
Deimel, Lachlan P., Zheyi Li, Sreeja Roy, and Charani Ranasinghe. “STAT3 Determines IL-4 Signalling Outcomes in Naïve T Cells.” Scientific Reports 11, no. 1 (May 18, 2021): 10495. https://doi.org/10.1038/s41598-021-89860-7.
Liu, Jiachen, Wei Yin, Lisa S. Westerberg, Pamela Lee, Quan Gong, Yan Chen, Lingli Dong, and Chaohong Liu. “Immune Dysregulation in IgG4-Related Disease.” Frontiers in Immunology 12 (September 1, 2021). https://doi.org/10.3389/fimmu.2021.738540.
Camporeale, Annalisa, and Valeria Poli. “IL-6, IL-17 and STAT3: A Holy Trinity in Auto-Immunity?” Frontiers in Bioscience-Landmark 17, no. 6 (June 1, 2012): 2306–26. https://doi.org/10.2741/4054.
Zongfei, Ji, Chen Rongyi, Cui Xiaomeng, Ma Lili, Ma Lingying, Kong Xiufang, Dai Xiaomin, et al. “In Vitro IL-6/IL-6R Trans-Signaling in Fibroblasts Releases Cytokines That May Be Linked to the Pathogenesis of IgG4-Related Disease.” Frontiers in Immunology 11 (July 8, 2020). https://doi.org/10.3389/fimmu.2020.01272.
Aochi, Satsuki, Masaaki Uehara, and Motohisa Yamamoto. “IgG4-Related Disease Emerging after COVID-19 mRNA Vaccination.” Internal Medicine 62, no. 10 (2023): 1547–51. https://doi.org/10.2169/internalmedicine.1125-22.
Buhre, Jana Sophia, Tamas Pongracz, Inga Künsting, Anne S. Lixenfeld, Wenjun Wang, Jan Nouta, Selina Lehrian, et al. “mRNA Vaccines against SARS-CoV-2 Induce Comparably Low Long-Term IgG Fc Galactosylation and Sialylation Levels but Increasing Long-Term IgG4 Responses Compared to an Adenovirus-Based Vaccine.” Frontiers in Immunology 13 (January 12, 2023). https://doi.org/10.3389/fimmu.2022.1020844.
Hartley, Gemma E., Holly A. Fryer, Paul A. Gill, Irene Boo, Scott J. Bornheimer, P. Mark Hogarth, Heidi E. Drummer, Robyn E. O’Hehir, Emily S. J. Edwards, and Menno C. van Zelm. “Third Dose COVID-19 mRNA Vaccine Enhances IgG4 Isotype Switching and Recognition of Omicron Subvariants by Memory B Cells after mRNA but Not Adenovirus Priming.” bioRxiv, September 19, 2023. https://doi.org/10.1101/2023.09.15.557929.
Irrgang, Pascal, Juliane Gerling, Katharina Kocher, Dennis Lapuente, Philipp Steininger, Katharina Habenicht, Monika Wytopil, et al. “Class Switch toward Noninflammatory, Spike-Specific IgG4 Antibodies after Repeated SARS-CoV-2 mRNA Vaccination.” Science Immunology 8, no. 79 (January 27, 2023): eade2798. https://doi.org/10.1126/sciimmunol.ade2798.
Kiszel, Petra, Pál Sík, János Miklós, Erika Kajdácsi, György Sinkovits, László Cervenak, and Zoltán Prohászka. “Class Switch towards Spike Protein-Specific IgG4 Antibodies after SARS-CoV-2 mRNA Vaccination Depends on Prior Infection History.” Scientific Reports 13, no. 1 (August 13, 2023): 13166. https://doi.org/10.1038/s41598-023-40103-x.
Sheehan, Jared, Caleb M. Ardizzone, Mayank Khanna, Amber J. Trauth, Michael E. Hagensee, and Alistair J. Ramsay. “Dynamics of Serum-Neutralizing Antibody Responses in Vaccinees through Multiple Doses of the BNT162b2 Vaccine.” Vaccines 11, no. 11 (November 15, 2023): 1720. https://doi.org/10.3390/vaccines11111720.
Sheehan, Jared, Caleb M. Ardizzone, Mayank Khanna, Amber J. Trauth, Michael E. Hagensee, and Alistair J. Ramsay. “Dynamics of Serum-Neutralizing Antibody Responses in Vaccinees through Multiple Doses of the BNT162b2 Vaccine.” Vaccines 11, no. 11 (November 15, 2023): 1720. https://doi.org/10.3390/vaccines11111720.
Uversky, Vladimir, Elrashdy Redwan, William Makis, and Alberto Rubio-Casillas. “IgG4 Antibodies Induced by Repeated Vaccination May Generate Immune Tolerance to the SARS-CoV-2 Spike Protein.” Vaccines 11, no. 5 (May 17, 2023): 991. https://doi.org/10.3390/vaccines11050991.
Gelderloos, Anne T., Marije K. Verheul, Irene Middelhof, Mary-Lène de Zeeuw-Brouwer, Robert S. van Binnendijk, Anne-Marie Buisman, and Puck B. van Kasteren. “Repeated COVID-19 mRNA Vaccination Results in IgG4 Class Switching and Decreased NK Cell Activation by S1-Specific Antibodies in Older Adults.” Immunity & Ageing 21, no. 1 (September 14, 2024): 63. https://doi.org/10.1186/s12979-024-00466-9.
Kalkeri, Raj, Mingzhu Zhu, Shane Cloney-Clark, Joyce S. Plested, Anand Parekh, Drew Gorinson, Rongman Cai, et al. “Altered IgG4 Antibody Response to Repeated mRNA versus Recombinant Protein SARS-CoV-2 Vaccines.” Journal of Infection 88, no. 3 (March 2024): 106119. https://doi.org/10.1016/j.jinf.2024.106119.
Kobbe, Robin, Cornelius Rau, Ulf Schulze-Sturm, Felix Stahl, Luis Fonseca-Brito, Anke Diemert, Marc Lütgehetmann, Marylyn M. Addo, Petra Arck, and Leonie M. Weskamm. “Delayed Induction of Noninflammatory SARS-CoV-2 Spike-Specific IgG4 Antibodies Detected 1 Year After BNT162b2 Vaccination in Children.” The Pediatric Infectious Disease Journal 43, no. 12 (July 30, 2024): 1200–1203. https://doi.org/10.1097/INF.0000000000004488.
Mizuno, Tomohito, Yoko Endo, Atsushi Suzuki, Masashi Suzuki, Tomohito Mizuno, Yoko Endo, Atsushi Suzuki, and Masashi Suzuki. “IgG4-Related Membranous Nephropathy After COVID-19 Vaccination: A Case Report.” Cureus 16, no. 3 (March 12, 2024). https://doi.org/10.7759/cureus.56028.
Tam, Jerry C. H., Abbie C. Sibayan, Jeffrey Seow, Carl Graham, Ashwini Kurshan, Blair Merrick, Richard J. Stanton, and Katie J. Doores. “Spike-Specific IgG4 Generated Post BNT162b2 mRNA Vaccination Is Inhibitory When Directly Competing with Functional IgG Subclasses.” bioRxiv, February 13, 2025. https://doi.org/10.1101/2025.02.12.637095.
Ayres, Janelle S., and David S. Schneider. “Two Ways to Survive an Infection: What Resistance and Tolerance Can Teach Us about Treatments for Infectious Diseases.” Nature Reviews. Immunology 8, no. 11 (November 2008): 889–95. https://doi.org/10.1038/nri2432.
Babayev, Huseyn, Ali Sahin, Sena Ardicli, Mubeccel Akdis, and Cezmi A. Akdis. “Tracing the Evolutionary Pathway of IgG4 : Implications for Immune Tolerance and Regulation.” Allergy 80, no. 1 (January 2025): 346–48. https://doi.org/10.1111/all.16383.
Han, Lei, Tianxiang Wu, Qin Zhang, Anning Qi, and Xiaohui Zhou. “Immune Tolerance Regulation Is Critical to Immune Homeostasis.” Edited by Arif Siddiqui. Journal of Immunology Research 2025, no. 1 (January 2025): 5006201. https://doi.org/10.1155/jimr/5006201.
Schmeling, Max, Vibeke Manniche, and Peter Riis Hansen. “Batch‐dependent Safety of the BNT162b2 mRNA COVID‐19 Vaccine.” European Journal of Clinical Investigation 53, no. 8 (August 2023): e13998. https://doi.org/10.1111/eci.13998.
Manniche, Vibeke, Max Schmeling, Jonathan D. Gilthorpe, and Peter Riis Hansen. “Reports of Batch-Dependent Suspected Adverse Events of the BNT162b2 mRNA COVID-19 Vaccine: Comparison of Results from Denmark and Sweden.” Medicina 60, no. 8 (August 19, 2024): 1343. https://doi.org/10.3390/medicina60081343.
Jablonowski, Karl, and Brian Hooker. “Batch-Dependent Safety of the BNT162b2 mRNA COVID-19 Vaccine in the United States - Science, Public Health Policy and the Law,” September 26, 2024. https://publichealthpolicyjournal.com/batch-dependent-safety-of-the-bnt162b2-mrna-covid-19-vaccine-in-the-united-states/.
Hulscher, Nicolas, and Peter McCullough. “Delayed Fatal Pulmonary Hemorrhage Following Covid-19 Vaccination: Case Report, Batch Analysis, And Proposed Autopsy Checklist.” International Journal of Innovative Research in Medical Science 10, no. 02 (February 8, 2025): 37–42. https://doi.org/10.23958/ijirms/vol10-i02/2035.
Guetzkow, Josh A, and Retsef Levi. “Effect of mRNA Vaccine Manufacturing Processes on Efficacy and Safety Still an Open Question.” BMJ 2022;378:O1731, June 12, 2025. https://doi.org/10.1136/bmj.o1731.
McKernan, Kevin, Yvonne Helbert, Liam T. Kane, and Stephen McLaughlin. “Sequencing of Bivalent Moderna and Pfizer mRNA Vaccines Reveals Nanogram to Microgram Quantities of Expression Vector dsDNA per Dose,” April 10, 2023. https://doi.org/10.31219/osf.io/b9t7m.
Speicher, David J, Jessica Rose, L. Maria Gutschi, David M Wiseman, and Kevin McKernan. “DNA Fragments Detected in Monovalent and Bivalent Pfizer/BioNTech and Moderna modRNA COVID-19 Vaccines from Ontario, Canada: Exploratory Dose Response Relationship with Serious Adverse Events.,” October 19, 2023. https://doi.org/10.31219/osf.io/mjc97.
König, Brigitte, and Jürgen O. Kirchner. “Methodological Considerations Regarding the Quantification of DNA Impurities in the COVID-19 mRNA Vaccine Comirnaty®.” Methods and Protocols 7, no. 3 (May 8, 2024): 41. https://doi.org/10.3390/mps7030041.
Wang, Tyler J., Alex Kim, and Kevin Kim. “A Rapid Detection Method of Replication-Competent Plasmid DNA from COVID-19 mRNA Vaccines for Quality Control.” Journal of High School Science 8, no. 4 (December 29, 2024): 427–39.
Kämmerer, Ulrike, Schulz, Verena, Steger. Klaus. “BioNTech RNA-Based COVID-19 Injections Contain Large Amounts Of Residual DNA Including An SV40 Promoter/Enhancer Sequence - Science, Public Health Policy and the Law,” December 3, 2024. https://publichealthpolicyjournal.com/biontech-rna-based-covid-19-injections-contain-large-amounts-of-residual-dna-including-an-sv40-promoter-enhancer-sequence/.
Kaiser, Stefanie, Steffen Kaiser, Jenny Reis, and Rolf Marschalek. “Quantification of Objective Concentrations of DNA Impurities in mRNA Vaccines.” Vaccine 55 (May 10, 2025): 127022. https://doi.org/10.1016/j.vaccine.2025.127022.
Kurinomaru, Takaaki, Naoshi Kojima, and Ryoji Kurita. “An Alkylating Immobilization Linker for Immunochemical Epigenetic Assessment.” Chemical Communications 53, no. 59 (July 20, 2017): 8308–11. https://doi.org/10.1039/C7CC02883K.
Ammann, Gregor, Maximilian Berg, Jan Felix Dalwigk, and Stefanie M. Kaiser. “Pitfalls in RNA Modification Quantification Using Nucleoside Mass Spectrometry.” Accounts of Chemical Research 56, no. 22 (November 9, 2023): 3121–31. https://doi.org/10.1021/acs.accounts.3c00402.
Ban, Eunmi, and Aeri Kim. “PicoGreen Assay for Nucleic Acid Quantification - Applications, Challenges, and Solutions.” Analytical Biochemistry 692 (September 1, 2024): 115577. https://doi.org/10.1016/j.ab.2024.115577.
Schober, Gretchen B., Sandra Story, and Dev P. Arya. “A Careful Look at Lipid Nanoparticle Characterization: Analysis of Benchmark Formulations for Encapsulation of RNA Cargo Size Gradient.” Scientific Reports 14, no. 1 (January 29, 2024): 2403. https://doi.org/10.1038/s41598-024-52685-1.
Wei, Xing, Yingxiang Liu, Weijie Hao, Peiwen Feng, Lei Zhang, Hongni Xue, Qunli Zhou, and Zekun Guo. “PGC7 Regulates Genome-Wide DNA Methylation by Regulating ERK-Mediated Subcellular Localization of DNMT1.” International Journal of Molecular Sciences 24, no. 4 (February 4, 2023): 3093. https://doi.org/10.3390/ijms24043093.
Cohen-Armon, Malka, Leonid Visochek, Dana Rozensal, Adi Kalal, Ilona Geistrikh, Rodika Klein, Sarit Bendetz-Nezer, Zhong Yao, and Rony Seger. “DNA-Independent PARP-1 Activation by Phosphorylated ERK2 Increases Elk1 Activity: A Link to Histone Acetylation.” Molecular Cell 25, no. 2 (January 26, 2007): 297–308. https://doi.org/10.1016/j.molcel.2006.12.012.
Navandar, Mohit, Angela Garding, Sanjeeb Kumar Sahu, Abhijeet Pataskar, Sandra Schick, and Vijay K. Tiwari. “ERK Signalling Modulates Epigenome to Drive Epithelial to Mesenchymal Transition.” Oncotarget 8, no. 17 (April 25, 2017): 29269–81. https://doi.org/10.18632/oncotarget.16493.
Li, Jialun, Ruiping Wang, Xueli Hu, Yingying Gao, Zhen Wang, Jiwen Li, and Jiemin Wong. “Activated MEK/ERK Pathway Drives Widespread and Coordinated Overexpression of UHRF1 and DNMT1 in Cancer Cells.” Scientific Reports 9, no. 1 (January 29, 2019): 907. https://doi.org/10.1038/s41598-018-37258-3.
Pradhan, Nibedita, Sabnam Parbin, Swayamsiddha Kar, Laxmidhar Das, R. Kirtana, G. Suma Seshadri, Dipta Sengupta, Moonmoon Deb, Chahat Kausar, and Samir Kumar Patra. “Epigenetic Silencing of Genes Enhanced by Collective Role of Reactive Oxygen Species and MAPK Signaling Downstream ERK/Snail Axis: Ectopic Application of Hydrogen Peroxide Repress CDH1 Gene by Enhanced DNA Methyltransferase Activity in Human Breast Cancer.” Biochimica et Biophysica Acta (BBA) - Molecular Basis of Disease 1865, no. 6 (June 1, 2019): 1651–65. https://doi.org/10.1016/j.bbadis.2019.04.002.
Sobhiafshar, Ulduz, Betül Çakici, Erdem Yilmaz, Nalan Yildiz Ayhan, Laila Hedaya, Mustafa Can Ayhan, Cansu Yerinde, et al. “Interferon Regulatory Factor 4 Modulates Epigenetic Silencing and Cancer‐critical Pathways in Melanoma Cells.” Molecular Oncology 18, no. 10 (June 16, 2024): 2423–48. https://doi.org/10.1002/1878-0261.13672.
Mishra, Gyan Prakash, Atimukta Jha, Abdul Ahad, Kaushik Sen, Aishwarya Sen, Sreeparna Podder, Subhasish Prusty, Viplov Kumar Biswas, Bhawna Gupta, and Sunil Kumar Raghav. “Epigenomics of Conventional Type-I Dendritic Cells Depicted Preferential Control of TLR9 versus TLR3 Response by NCoR1 through Differential IRF3 Activation.” Cellular and Molecular Life Sciences: CMLS 79, no. 8 (July 18, 2022): 429. https://doi.org/10.1007/s00018-022-04424-w.
Hu, Ye, Xin Wang, Jiaying Song, Jiacheng Wu, Jia Xu, Yangyang Chai, Yuanyuan Ding, et al. “Chromatin Remodeler ARID1A Binds IRF3 to Selectively Induce Antiviral Interferon Production in Macrophages.” Cell Death & Disease 12, no. 8 (July 27, 2021): 1–11. https://doi.org/10.1038/s41419-021-04032-9.
Carpenter, Susan, Paulina Wochal, Aisling Dunne, and Luke A. J. O’Neill. “Toll-like Receptor 3 (TLR3) Signaling Requires TLR4 Interactor with Leucine-Rich Repeats (TRIL).” The Journal of Biological Chemistry 286, no. 44 (November 4, 2011): 38795–804. https://doi.org/10.1074/jbc.M111.255893.
Ding, Xibing, Shuqing Jin, Yao Tong, Xi Jiang, Zhixia Chen, Shuya Mei, Liming Zhang, Timothy R. Billiar, and Quan Li. “TLR4 Signaling Induces TLR3 Up-Regulation in Alveolar Macrophages during Acute Lung Injury.” Scientific Reports 7, no. 1 (February 15, 2017): 34278. https://doi.org/10.1038/srep34278.
Fitzpatrick, John-Mark, Eleanor Minogue, Lucy Curham, Harry Tyrrell, Philip Gavigan, William Hind, and Eric J. Downer. “MyD88-Dependent and -Independent Signalling via TLR3 and TLR4 Are Differentially Modulated by Δ9-Tetrahydrocannabinol and Cannabidiol in Human Macrophages.” Journal of Neuroimmunology 343 (June 15, 2020): 577217. https://doi.org/10.1016/j.jneuroim.2020.577217.
Palazzo, M., S. Gariboldi, L. Zanobbio, G. F. Dusio, S. Selleri, M. Bedoni, A. Balsari, and C. Rumio. “Cross-Talk among Toll-like Receptors and Their Ligands.” International Immunology 20, no. 5 (May 1, 2008): 709–18. https://doi.org/10.1093/intimm/dxn027.
Suh, Hyeon-Sook, Meng-Liang Zhao, Namjong Choi, Thomas J. Belbin, Celia F. Brosnan, and Sunhee C. Lee. “TLR3 and TLR4 Are Innate Antiviral Immune Receptors in Human Microglia: Role of IRF3 in Modulating Antiviral and Inflammatory Response in the CNS.” Virology 392, no. 2 (September 30, 2009): 246–59. https://doi.org/10.1016/j.virol.2009.07.001.
Tan, Rebecca S. T., Bow Ho, Bernard P. Leung, and Jeak L. Ding. “TLR Cross-Talk Confers Specificity to Innate Immunity.” International Reviews of Immunology 33, no. 6 (November 2014): 443–53. https://doi.org/10.3109/08830185.2014.921164.
Ghosh, Tarun K., Dan J. Mickelson, Jonathan C. Solberg, Kenneth E. Lipson, Jon R. Inglefield, and Sefik S. Alkan. “TLR–TLR Cross Talk in Human PBMC Resulting in Synergistic and Antagonistic Regulation of Type-1 and 2 Interferons, IL-12 and TNF-α.” International Immunopharmacology 7, no. 8 (August 1, 2007): 1111–21. https://doi.org/10.1016/j.intimp.2007.04.006.
Nouri-Shirazi, Mahyar, Saba Tamjidi, Erika Nourishirazi, and Elisabeth Guinet. “TLR8 Combined withTLR3 or TLR4 Agonists Enhances DC-NK Driven Effector Tc1 Cells.” Immunology Letters 193 (January 1, 2018): 58–66. https://doi.org/10.1016/j.imlet.2017.10.015.
Assier, Eric, Viviana Marin-Esteban, Alain Haziot, Enrico Maggi, Dominique Charron, and Nuala Mooney. “TLR7/8 Agonists Impair Monocyte-Derived Dendritic Cell Differentiation and Maturation.” Journal of Leukocyte Biology 81, no. 1 (January 1, 2007): 221–28. https://doi.org/10.1189/jlb.0705385.
Hu, Cexun, Keyu Zhang, Feng Jiang, Hui Wang, and Qixiang Shao. “Epigenetic Modifications in Thymic Epithelial Cells: An Evolutionary Perspective for Thymus Atrophy.” Clinical Epigenetics 13, no. 1 (November 24, 2021): 210. https://doi.org/10.1186/s13148-021-01197-0.
Université Paris-Saclay. “The Unknown Inner Workings That Control Our Genes,” February 23, 2023. http://www.universite-paris-saclay.fr/en/news/unknown-inner-workings-control-our-genes.
Breton, Carrie V., Remy Landon, Linda G. Kahn, Michelle Bosquet Enlow, Alicia K. Peterson, Theresa Bastain, Joseph Braun, et al. “Exploring the Evidence for Epigenetic Regulation of Environmental Influences on Child Health across Generations.” Communications Biology 4, no. 1 (June 22, 2021): 1–15. https://doi.org/10.1038/s42003-021-02316-6.
Langille, Ellen, Khalid N. Al-Zahrani, Zhibo Ma, Minggao Liang, Liis Uuskula-Reimand, Roderic Espin, Katie Teng, et al. “Loss of Epigenetic Regulation Disrupts Lineage Integrity, Induces Aberrant Alveogenesis, and Promotes Breast Cancer.” Cancer Discovery 12, no. 12 (December 2, 2022): 2930–53. https://doi.org/10.1158/2159-8290.CD-21-0865.
León, Beatriz, María López-Bravo, and Carlos Ardavín. “Monocyte-Derived Dendritic Cells.” Seminars in Immunology, Dendritic Cell Heterogenity, 17, no. 4 (August 1, 2005): 313–18. https://doi.org/10.1016/j.smim.2005.05.013.
Italiani, Paola, and Diana Boraschi. “From Monocytes to M1/M2 Macrophages: Phenotypical vs. Functional Differentiation.” Frontiers in Immunology 5 (October 17, 2014). https://doi.org/10.3389/fimmu.2014.00514.
Musolino, Elettra, Christina Pagiatakis, Simone Serio, Marina Borgese, Federica Gamberoni, Rosalba Gornati, Giovanni Bernardini, and Roberto Papait. “The Yin and Yang of Epigenetics in the Field of Nanoparticles.” Nanoscale Advances 4, no. 4 (n.d.): 979–94. https://doi.org/10.1039/d1na00682g.
Connors, Jennifer, David Joyner, Nathan J. Mege, Gina M. Cusimano, Matthew R. Bell, Jennifer Marcy, Bhavani Taramangalam, et al. “Lipid Nanoparticles (LNP) Induce Activation and Maturation of Antigen Presenting Cells in Young and Aged Individuals.” Communications Biology 6, no. 1 (February 17, 2023): 1–13. https://doi.org/10.1038/s42003-023-04555-1.
Zhang, Yingxue, Eid Alshammari, Nouran Yonis, and Zhe Yang. “Adverse Epigenetic Effects of Environmental Engineered Nanoparticles as Drug Carriers.” In Impact of Engineered Nanomaterials in Genomics and Epigenomics, edited by Saura C. Sahu, 1st ed., 251–81. Wiley, 2023. https://doi.org/10.1002/9781119896258.ch10.
sanei, Maryam, Bagher Amirheidari, and Naghmeh Satarzadeh. “Mutuality of Epigenetic and Nanoparticles: Two Sides of a Coin.” Heliyon 10, no. 1 (January 15, 2024): e23679. https://doi.org/10.1016/j.heliyon.2023.e23679.
Pongma, Chitsuda, Pornlapat Keawvilai, Atsadang Boonmee, Benjawan Wongprom, Thitiporn Pattarakankul, Chutamath Sittplangkoon, Mohamad-Gabriel Alameh, et al. “Effect of mRNA Formulated with Lipid Nanoparticles on the Transcriptomic and Epigenetic Profiles of F4/80+ Liver-Associated Macrophages.” Scientific Reports 15, no. 1 (January 7, 2025): 1146. https://doi.org/10.1038/s41598-025-85234-5.
Mokhtari, Tahere, Mohammad N. Taheri, Sarah Akhlaghi, Armin Aryannejad, Yuda Xiang, Vineet Mahajan, Kamyar Keshavarz, et al. “Enhanced Epigenetic Modulation via mRNA-Encapsulated Lipid Nanoparticles Enables Targeted Anti-Inflammatory Control.” bioRxiv, February 28, 2025, 2025.02.24.639996. https://doi.org/10.1101/2025.02.24.639996.
Qin, Zhen, Aurélie Bouteau, Christopher Herbst, and Botond Z. Igyártó. “Pre-Exposure to mRNA-LNP Inhibits Adaptive Immune Responses and Alters Innate Immune Fitness in an Inheritable Fashion.” PLOS Pathogens 18, no. 9 (February 9, 2022): e1010830. https://doi.org/10.1371/journal.ppat.1010830.
Hellgren, Fredrika, Anja Rosdahl, Rodrigo Arcoverde Cerveira, Klara Lenart, Sebastian Ols, Yong-Dae Gwon, Seta Kurt, et al. “Modulation of Innate Immune Response to mRNA Vaccination after SARS-CoV-2 Infection or Sequential Vaccination in Humans.” JCI Insight 9, no. 9 (n.d.): e175401. https://doi.org/10.1172/jci.insight.175401.
Yamaguchi, Yuta, Yasuhiro Kato, Ryuya Edahiro, Jonas N. Søndergaard, Teruaki Murakami, Saori Amiya, Shinichiro Nameki, et al. “Consecutive BNT162b2 mRNA Vaccination Induces Short-Term Epigenetic Memory in Innate Immune Cells.” JCI Insight 7, no. 22 (November 22, 2022): e163347. https://doi.org/10.1172/jci.insight.163347.
Yona, Simon, Ki-Wook Kim, Yochai Wolf, Alexander Mildner, Diana Varol, Michal Breker, Dalit Strauss-Ayali, et al. “Fate Mapping Reveals Origins and Dynamics of Monocytes and Tissue Macrophages under Homeostasis.” Immunity 38, no. 1 (January 24, 2013): 79–91. https://doi.org/10.1016/j.immuni.2012.12.001.
Patel, Amit A., Yan Zhang, James N. Fullerton, Lies Boelen, Anthony Rongvaux, Alexander A. Maini, Venetia Bigley, et al. “The Fate and Lifespan of Human Monocyte Subsets in Steady State and Systemic Inflammation.” The Journal of Experimental Medicine 214, no. 7 (July 3, 2017): 1913–23. https://doi.org/10.1084/jem.20170355.
Gifford, Casey A., Michael J. Ziller, Hongcang Gu, Cole Trapnell, Julie Donaghey, Alexander Tsankov, Alex K. Shalek, et al. “Transcriptional and Epigenetic Dynamics during Specification of Human Embryonic Stem Cells.” Cell 153, no. 5 (May 23, 2013): 1149–63. https://doi.org/10.1016/j.cell.2013.04.037.
Nicholas, Dequina, Hui Tang, Qiongyi Zhang, Jai Rudra, Feng Xu, William Langridge, and Kangling Zhang. “Quantitative Proteomics Reveals a Role for Epigenetic Reprogramming During Human Monocyte Differentiation.” Molecular & Cellular Proteomics : MCP 14, no. 1 (January 2015): 15–29. https://doi.org/10.1074/mcp.M113.035089.
Wallner, Stefan, Christopher Schröder, Elsa Leitão, Tea Berulava, Claudia Haak, Daniela Beißer, Sven Rahmann, et al. “Epigenetic Dynamics of Monocyte-to-Macrophage Differentiation.” Epigenetics & Chromatin 9 (2016): 33. https://doi.org/10.1186/s13072-016-0079-z.
Davis, Frank M., and Katherine A. Gallagher. “Epigenetic Mechanisms in Monocytes/Macrophages Regulate Inflammation in Cardiometabolic and Vascular Disease.” Arteriosclerosis, Thrombosis, and Vascular Biology 39, no. 4 (April 2019): 623–34. https://doi.org/10.1161/ATVBAHA.118.312135.
Naler, Lynette B., Yuan-Pang Hsieh, Shuo Geng, Zirui Zhou, Liwu Li, and Chang Lu. “Epigenomic and Transcriptomic Analyses Reveal Differences between Low-Grade Inflammation and Severe Exhaustion in LPS-Challenged Murine Monocytes.” Communications Biology 5, no. 1 (January 28, 2022): 1–17. https://doi.org/10.1038/s42003-022-03035-2.
Simonis, Alexander, Sebastian J Theobald, Anna E Koch, Ram Mummadavarapu, Julie M Mudler, Andromachi Pouikli, Ulrike Göbel, et al. “Persistent Epigenetic Memory of SARS-CoV-2 mRNA Vaccination in Monocyte-Derived Macrophages.” Molecular Systems Biology 21, no. 4 (March 25, 2025): 341–60. https://doi.org/10.1038/s44320-025-00093-6.
Yuan, Zhi-Chao, Wang-Dong Xu, Xiao-Yan Liu, Xing-You Liu, An-Fang Huang, and Lin-Chong Su. “Biology of IL-36 Signaling and Its Role in Systemic Inflammatory Diseases.” Frontiers in Immunology 10 (October 31, 2019). https://doi.org/10.3389/fimmu.2019.02532.
Turner, Mark D., Belinda Nedjai, Tara Hurst, and Daniel J. Pennington. “Cytokines and Chemokines: At the Crossroads of Cell Signalling and Inflammatory Disease.” Biochimica et Biophysica Acta (BBA) - Molecular Cell Research 1843, no. 11 (November 1, 2014): 2563–82. https://doi.org/10.1016/j.bbamcr.2014.05.014.
Alaaeddine, Nada, John Antoniou, Mayssam Moussa, George Hilal, Gaby Kreichaty, Ismat Ghanem, Wissam Abouchedid, Elie Saghbini, and John A. Di Battista. “The Chemokine CCL20 Induces Proinflammatory and Matrix Degradative Responses in Cartilage.” Inflammation Research: Official Journal of the European Histamine Research Society ... [et Al.] 64, no. 9 (September 2015): 721–31. https://doi.org/10.1007/s00011-015-0854-5.
Korbecki, Jan, Magdalena Gąssowska-Dobrowolska, Jerzy Wójcik, Iwona Szatkowska, Katarzyna Barczak, Mikołaj Chlubek, and Irena Baranowska-Bosiacka. “The Importance of CXCL1 in Physiology and Noncancerous Diseases of Bone, Bone Marrow, Muscle and the Nervous System.” International Journal of Molecular Sciences 23, no. 8 (April 11, 2022): 4205. https://doi.org/10.3390/ijms23084205.
Amati, Anca-Laura, Anna Zakrzewicz, Robin Siebers, Sigrid Wilker, Sarah Heldmann, Dariusz Zakrzewicz, Andreas Hecker, J. Michael McIntosh, Winfried Padberg, and Veronika Grau. “Chemokines (CCL3, CCL4, and CCL5) Inhibit ATP-Induced Release of IL-1β by Monocytic Cells.” Mediators of Inflammation 2017 (2017): 1434872. https://doi.org/10.1155/2017/1434872.
Kim, Young Hee, So Young Jang, Yoon Kyung Shin, Young Rae Jo, Byeol-A. Yoon, Soo Hyun Nam, Byung-Ok Choi, et al. “Serum CXCL13 Reflects Local B-Cell Mediated Inflammatory Demyelinating Peripheral Neuropathy.” Scientific Reports 9, no. 1 (November 11, 2019): 16535. https://doi.org/10.1038/s41598-019-52643-2.
Kowarik, Markus C., Sabine Cepok, Johann Sellner, Verena Grummel, Martin S. Weber, Thomas Korn, Achim Berthele, and Bernhard Hemmer. “CXCL13 Is the Major Determinant for B Cell Recruitment to the CSF during Neuroinflammation.” Journal of Neuroinflammation 9, no. 1 (May 16, 2012): 93. https://doi.org/10.1186/1742-2094-9-93.
Rupprecht, Tobias A, Andreas Plate, Michaela Adam, Manfed Wick, Stefan Kastenbauer, Caroline Schmidt, Matthias Klein, Hans-Walter Pfister, and Uwe Koedel. “The Chemokine CXCL13 Is a Key Regulator of B Cell Recruitment to the Cerebrospinal Fluid in Acute Lyme Neuroborreliosis.” Journal of Neuroinflammation 6 (December 30, 2009): 42. https://doi.org/10.1186/1742-2094-6-42.
Salomonsson, S., P. Larsson, P. Tengnér, E. Mellquist, P. Hjelmström, and M. Wahren‐Herlenius. “Expression of the B Cell‐Attracting Chemokine CXCL13 in the Target Organ and Autoantibody Production in Ectopic Lymphoid Tissue in the Chronic Inflammatory Disease Sjögren’s Syndrome.” Scandinavian Journal of Immunology 55, no. 4 (April 2002): 336–42. https://doi.org/10.1046/j.1365-3083.2002.01058.x.
Bardina, Susana V., Daniela Michlmayr, Kevin W. Hoffman, Christopher J. Obara, Janet Sum, Israel F. Charo, Wuyuan Lu, Alexander G. Pletnev, and Jean K. Lim. “Differential Roles of Chemokines CCL2 and CCL7 in Monocytosis and Leukocyte Migration during West Nile Virus Infection.” Journal of Immunology (Baltimore, Md. : 1950) 195, no. 9 (November 1, 2015): 4306–18. https://doi.org/10.4049/jimmunol.1500352.
Dash, Shiba Prasad, Saloni Gupta, and Pranita P. Sarangi. “Monocytes and Macrophages: Origin, Homing, Differentiation, and Functionality during Inflammation.” Heliyon 10, no. 8 (April 30, 2024): e29686. https://doi.org/10.1016/j.heliyon.2024.e29686.
Liu, Yangyang, Yadi Cai, Li Liu, Yudong Wu, and Xiangyang Xiong. “Crucial Biological Functions of CCL7 in Cancer.” PeerJ 6 (June 14, 2018): e4928. https://doi.org/10.7717/peerj.4928.
Allavena, P., G. Bianchi, D. Zhou, J. van Damme, P. Jílek, S. Sozzani, and A. Mantovani. “Induction of Natural Killer Cell Migration by Monocyte Chemotactic Protein-1, -2 and -3.” European Journal of Immunology 24, no. 12 (December 1994): 3233–36. https://doi.org/10.1002/eji.1830241249.
Pang, Alina P. S., Albert T. Higgins-Chen, Florence Comite, Ioana Raica, Christopher Arboleda, Hannah Went, Tavis Mendez, et al. “Longitudinal Study of DNA Methylation and Epigenetic Clocks Prior to and Following Test-Confirmed COVID-19 and mRNA Vaccination.” Frontiers in Genetics 13 (June 3, 2022). https://doi.org/10.3389/fgene.2022.819749.
Giannotta, Girolamo, Antonio Murrone, and Nicola Giannotta. “COVID-19 mRNA Vaccines: From Inflammation to Hyperinflammation,” January 26, 2023. https://doi.org/10.20944/preprints202301.0460.v1.
Hirota, Masato, Miho Tamai, Sachie Yukawa, Naoyuki Taira, Melissa M. Matthews, Takeshi Toma, Yu Seto, et al. “Human Immune and Gut Microbial Parameters Associated with Inter-Individual Variations in COVID-19 mRNA Vaccine-Induced Immunity.” Communications Biology 6, no. 1 (April 20, 2023): 1–14. https://doi.org/10.1038/s42003-023-04755-9.
Kim, Hyeonhui, Hyo-Suk Ahn, Nahee Hwang, Yune Huh, Seonghyeon Bu, Kyung Jin Seo, Se Hwan Kwon, et al. “Epigenomic Landscape Exhibits Interferon Signaling Suppression in the Patient of Myocarditis after BNT162b2 Vaccination.” Scientific Reports 13, no. 1 (June 1, 2023): 8926. https://doi.org/10.1038/s41598-023-36070-y.
Mele, Dalila, Sabrina Ottolini, Andrea Lombardi, Daniela Conteianni, Alessandra Bandera, Barbara Oliviero, Stefania Mantovani, et al. “Long‐term Dynamics of Natural Killer Cells in Response to SARS‐CoV‐2 Vaccination: Persistently Enhanced Activity Postvaccination.” Journal of Medical Virology 96, no. 4 (April 2024): e29585. https://doi.org/10.1002/jmv.29585.
Goel, Rishi R., Mark M. Painter, Sokratis A. Apostolidis, Divij Mathew, Wenzhao Meng, Aaron M. Rosenfeld, Kendall A. Lundgreen, et al. “mRNA Vaccines Induce Durable Immune Memory to SARS-CoV-2 and Variants of Concern.” Science 374, no. 6572 (December 3, 2021): abm0829. https://doi.org/10.1126/science.abm0829.
Ivanova, Daria L., Ryan Krempels, Stephen L. Denton, Kevin D. Fettel, Giandor M. Saltz, David Rach, Rida Fatima, et al. “NK Cells Negatively Regulate CD8 T Cells to Promote Immune Exhaustion and Chronic Toxoplasma Gondii Infection.” Frontiers in Cellular and Infection Microbiology 10 (July 8, 2020). https://doi.org/10.3389/fcimb.2020.00313.
Erdogan, Mumin Alper, Orkun Gurbuz, Mehmet Fatih Bozkurt, and Oytun Erbas. “Prenatal Exposure to COVID-19 mRNA Vaccine BNT162b2 Induces Autism-Like Behaviors in Male Neonatal Rats: Insights into WNT and BDNF Signaling Perturbations.” Neurochemical Research 49, no. 4 (April 2024): 1034–48. https://doi.org/10.1007/s11064-023-04089-2.
Kim, D., O. Rath, W. Kolch, and K.-H. Cho. “A Hidden Oncogenic Positive Feedback Loop Caused by Crosstalk between Wnt and ERK Pathways.” Oncogene 26, no. 31 (July 2007): 4571–79. https://doi.org/10.1038/sj.onc.1210230.
Zhang, Ying, Tyler Pizzute, and Ming Pei. “A Review of Crosstalk between MAPK and Wnt Signals and Its Impact on Cartilage Regeneration.” Cell and Tissue Research 358, no. 3 (December 2014): 633–49. https://doi.org/10.1007/s00441-014-2010-x.
Jeong, Woo-Jeong, Eun Ji Ro, and Kang-Yell Choi. “Interaction between Wnt/β-Catenin and RAS-ERK Pathways and an Anti-Cancer Strategy via Degradations of β-Catenin and RAS by Targeting the Wnt/β-Catenin Pathway.” Npj Precision Oncology 2, no. 1 (February 20, 2018): 1–10. https://doi.org/10.1038/s41698-018-0049-y.
Daisy Precilla, S., Indrani Biswas, Shreyas S. Kuduvalli, and T. S. Anitha. “Crosstalk between PI3K/AKT/mTOR and WNT/β-Catenin Signaling in GBM - Could Combination Therapy Checkmate the Collusion?” Cellular Signalling 95 (July 2022): 110350. https://doi.org/10.1016/j.cellsig.2022.110350.
Khan, Meraj, and Lokesh Verma. “Crosstalk between Signaling Pathways (Rho/ROCK, TGF-β and Wnt/β-Catenin Pathways/ PI3K-AKT-mTOR) in Cataract: A Mechanistic Exploration and Therapeutic Strategy.” Gene 947 (May 2025): 149338. https://doi.org/10.1016/j.gene.2025.149338.
Jin, Yun-Yun, Ya-Ping Liang, Jia-Qi Pan, Wen-Hao Huang, Yan-Meng Feng, Wei-Jia Sui, Han Yu, Xiao-Dan Tang, Lin Zhu, and Jian-Huan Chen. “RNA Editing in Response to COVID-19 Vaccines: Unveiling Dynamic Epigenetic Regulation of Host Immunity.” Frontiers in Immunology 15 (September 6, 2024). https://doi.org/10.3389/fimmu.2024.1413704.
Föhse, Konstantin, Büsra Geckin, Martijn Zoodsma, Gizem Kilic, Zhaoli Liu, Rutger J. Röring, Gijs J. Overheul, et al. “The Impact of BNT162b2 mRNA Vaccine on Adaptive and Innate Immune Responses.” Clinical Immunology 255 (October 1, 2023): 109762. https://doi.org/10.1016/j.clim.2023.109762.
Noé, Andrés, Thanh D. Dang, Christine Axelrad, Emma Burrell, Susie Germano, Sonja Elia, David Burgner, Kirsten P. Perrett, Nigel Curtis, and Nicole L. Messina. “BNT162b2 COVID-19 Vaccination in Children Alters Cytokine Responses to Heterologous Pathogens and Toll-like Receptor Agonists.” Frontiers in Immunology 14 (August 25, 2023): 1242380. https://doi.org/10.3389/fimmu.2023.1242380.
Estep, Benjamin K., Charles J. Kuhlmann, Satoru Osuka, Gajendra W. Suryavanshi, Yoshiko Nagaoka-Kamata, Ciearria N. Samuel, Madison T. Blucas, Chloe E. Jepson, Paul A. Goepfert, and Masakazu Kamata. “Skewed Fate and Hematopoiesis of CD34+ HSPCs in Umbilical Cord Blood amid the COVID-19 Pandemic.” iScience 25, no. 12 (December 22, 2022): 105544. https://doi.org/10.1016/j.isci.2022.105544.
Gur, Hilit, Rita Krauthgamer, Esther Bachar-Lustig, Helena Katchman, Rinat Arbel-Goren, Alain Berrebi, Tirza Klein, et al. “Immune Regulatory Activity of CD34+ Progenitor Cells: Evidence for a Deletion-Based Mechanism Mediated by TNF-α.” Blood 105, no. 6 (March 15, 2005): 2585–93. https://doi.org/10.1182/blood-2002-11-3463.
Refaeli, Yosef, Luk Van Parijs, Stephen I. Alexander, and Abul K. Abbas. “Interferon γ Is Required for Activation-Induced Death of T Lymphocytes.” The Journal of Experimental Medicine 196, no. 7 (October 7, 2002): 999–1005. https://doi.org/10.1084/jem.20020666.
Zeng, Weihua, Akira Miyazato, Guibin Chen, Sachiko Kajigaya, Neal S. Young, and Jaroslaw P. Maciejewski. “Interferon-γ-Induced Gene Expression in CD34 Cells: Identification of Pathologic Cytokine-Specific Signature Profiles.” Blood 107, no. 1 (January 1, 2006): 167–75. https://doi.org/10.1182/blood-2005-05-1884.
Morales-Mantilla, Daniel E., and Katherine Y. King. “The Role of Interferon-Gamma in Hematopoietic Stem Cell Development, Homeostasis, and Disease.” Current Stem Cell Reports 4, no. 3 (September 1, 2018): 264–71. https://doi.org/10.1007/s40778-018-0139-3.
Munje, Chinmay, Robert K. Hills, Anthony Whetton, Alan K. Burnett, Richard L. Darley, and Alex Tonks. “Cord Blood-Derived Quiescent CD34+ Cells Are More Transcriptionally Matched to AML Blasts Than Cytokine-Induced Normal Human Hematopoietic CD34+ Cells.” Gene Expression 16, no. 4 (December 9, 2015): 169–75. https://doi.org/10.3727/105221615X14399878166159.
Andrews, Jared M., and Jacqueline E. Payton. “Epigenetic Dynamics in Normal and Malignant B Cells: Die a Hero or Live to Become a Villain.” Current Opinion in Immunology 57 (April 2019): 15–22. https://doi.org/10.1016/j.coi.2018.09.020.
Bennett, Hunter, Ty D. Troutman, Mashito Sakai, and Christopher K. Glass. “Epigenetic Regulation of Kupffer Cell Function in Health and Disease.” Frontiers in Immunology 11 (January 26, 2021). https://doi.org/10.3389/fimmu.2020.609618.
Duliban, M., P. Pawlicki, A. Kamińska, B. Yurdakok-Dikmen, K. Tekin, and M. Kotula-Balak. “Status of Estrogen Receptor Expression and Epigenetic Methylation in Leydig Cells after Exposure to Metalloestrogen - Selenium.” Reproductive Toxicology 118 (June 1, 2023): 108389. https://doi.org/10.1016/j.reprotox.2023.108389.
Hudson, Jaylen, and Laszlo Farkas. “Epigenetic Regulation of Endothelial Dysfunction and Inflammation in Pulmonary Arterial Hypertension.” International Journal of Molecular Sciences 22, no. 22 (November 9, 2021): 12098. https://doi.org/10.3390/ijms222212098.
Sullivan, Adrienne E. “Epigenetic Control of Cell Potency and Fate Determination during Mammalian Gastrulation.” Genes 14, no. 6 (May 25, 2023): 1143. https://doi.org/10.3390/genes14061143.
Tsang, Tsz Hong, Meike Wiese, Martin Helmstädter, Thomas Stehle, Janine Seyfferth, Maria Shvedunova, Herbert Holz, Gerd Walz, and Asifa Akhtar. “Transcriptional Regulation by the NSL Complex Enables Diversification of IFT Functions in Ciliated versus Nonciliated Cells.” Science Advances 9, no. 34 (August 25, 2023): eadh5598. https://doi.org/10.1126/sciadv.adh5598.
Arzate‐Mejía, Rodrigo G., David Valle‐García, and Félix Recillas‐Targa. “Signaling Epigenetics: Novel Insights on Cell Signaling and Epigenetic Regulation.” IUBMB Life 63, no. 10 (October 2011): 881–95. https://doi.org/10.1002/iub.557.
Wu, Yan-Lin, Zheng-Jun Lin, Chang-Chun Li, Xiao Lin, Su-Kang Shan, Bei Guo, Ming-Hui Zheng, Fuxingzi Li, Ling-Qing Yuan, and Zhi-hong Li. “Epigenetic Regulation in Metabolic Diseases: Mechanisms and Advances in Clinical Study.” Signal Transduction and Targeted Therapy 8 (March 2, 2023): 98. https://doi.org/10.1038/s41392-023-01333-7.
Yang, Zhihong, and Li Wang. “Regulation of microRNA Expression and Function by Nuclear Receptor Signaling.” Cell & Bioscience 1, no. 1 (September 21, 2011): 31. https://doi.org/10.1186/2045-3701-1-31.
Saj, Abil, and Eric C. Lai. “Control of microRNA Biogenesis and Transcription by Cell Signaling Pathways.” Current Opinion in Genetics & Development 21, no. 4 (August 2011): 504–10. https://doi.org/10.1016/j.gde.2011.04.010.
Bortoletto, Angelina S., and Ronald J. Parchem. “KRAS Hijacks the miRNA Regulatory Pathway in Cancer.” Cancer Research 83, no. 10 (May 15, 2023): 1563–72. https://doi.org/10.1158/0008-5472.CAN-23-0296.
Inui, Masafumi, Graziano Martello, and Stefano Piccolo. “MicroRNA Control of Signal Transduction.” Nature Reviews Molecular Cell Biology 11, no. 4 (April 2010): 252–63. https://doi.org/10.1038/nrm2868.
Paroo, Zain, Xuecheng Ye, She Chen, and Qinghua Liu. “Phosphorylation of the Human microRNA-Generating Complex Mediates MAPK/Erk Signaling.” Cell 139, no. 1 (October 2, 2009): 112–22. https://doi.org/10.1016/j.cell.2009.06.044.
Copeland, Jacqueline, Kayla Wilson, and Marcos Simoes-Costa. “Micro-Managing Pattern Formation: miRNA Regulation of Signaling Systems in Vertebrate Development.” The FEBS Journal 289, no. 17 (September 2022): 5166–75. https://doi.org/10.1111/febs.16139.
Plaisier, Christopher L., Min Pan, and Nitin S. Baliga. “A miRNA-Regulatory Network Explains How Dysregulated miRNAs Perturb Oncogenic Processes across Diverse Cancers.” Genome Research 22, no. 11 (November 2012): 2302–14. https://doi.org/10.1101/gr.133991.111.
Hill, Meredith, and Nham Tran. “Global miRNA to miRNA Interactions: Impacts for miR-21.” Trends in Cell Biology 31, no. 1 (January 2021): 3–5. https://doi.org/10.1016/j.tcb.2020.10.005.
Jin, Daeyong, and Hyunju Lee. “A Computational Approach to Identifying Gene-microRNA Modules in Cancer.” PLOS Computational Biology 11, no. 1 (January 22, 2015): e1004042. https://doi.org/10.1371/journal.pcbi.1004042.
Gao, Fan, Fang Wang, Yue Chen, Bolin Deng, Fujian Yang, Huifen Cao, Junjie Chen, Huiling Chen, Fei Qi, and Philipp Kapranov. “The Human Genome Encodes a Multitude of Novel miRNAs.” Nucleic Acids Research 53, no. 4 (February 8, 2025): gkaf070. https://doi.org/10.1093/nar/gkaf070.
Yr, Fujii. “Quantum microRNA Assessment of COVID-19 RNA Vaccine: Hidden Potency of BNT162b2 SASR-CoV-2 Spike RNA as MicroRNA Vaccine.” Advancements in Case Studies 3, no. 1 (April 16, 2021): 1–12.
Miyashita, Yusuke, Takanobu Yoshida, Yuriko Takagi, Hirotake Tsukamoto, Ken Takashima, Takahisa Kouwaki, Katsunari Makino, Satoshi Fukushima, Kimitoshi Nakamura, and Hiroyuki Oshiumi. “Circulating Extracellular Vesicle microRNAs Associated with Adverse Reactions, Proinflammatory Cytokine, and Antibody Production after COVID-19 Vaccination.” Npj Vaccines 7, no. 1 (February 8, 2022): 1–11. https://doi.org/10.1038/s41541-022-00439-3.
Zhang, Duo, Heedoo Lee, Ziwen Zhu, Jasleen K. Minhas, and Yang Jin. “Enrichment of Selective miRNAs in Exosomes and Delivery of Exosomal miRNAs in Vitro and in Vivo.” American Journal of Physiology - Lung Cellular and Molecular Physiology 312, no. 1 (January 1, 2017): L110–21. https://doi.org/10.1152/ajplung.00423.2016.
Asgarpour, Kasra, Zahra Shojaei, Fatemeh Amiri, Jafar Ai, Maryam Mahjoubin-Tehran, Faezeh Ghasemi, Reza ArefNezhad, Michael R. Hamblin, and Hamed Mirzaei. “Exosomal microRNAs Derived from Mesenchymal Stem Cells: Cell-to-Cell Messages.” Cell Communication and Signaling 18, no. 1 (September 11, 2020): 149. https://doi.org/10.1186/s12964-020-00650-6.
Chetta, Massimiliano, Marina Tarsitano, Maria Oro, Maria Rivieccio, and Nenad Bukvic. “An in Silico Pipeline Approach Uncovers a Potentially Intricate Network Involving Spike SARS-CoV-2 RNA, RNA Vaccines, Host RNA-Binding Proteins (RBPs), and Host miRNAs at the Cellular Level.” Journal of Genetic Engineering and Biotechnology 20, no. 1 (September 6, 2022): 129. https://doi.org/10.1186/s43141-022-00413-5.
Lin, Yen-Pin, Yi-Shan Hsieh, Mei-Hsiu Cheng, Ching-Fen Shen, Ching-Ju Shen, and Chao-Min Cheng. “Using MicroRNA Arrays as a Tool to Evaluate COVID-19 Vaccine Efficacy.” Vaccines 10, no. 10 (October 8, 2022): 1681. https://doi.org/10.3390/vaccines10101681.
Légaré, Cécilia, Andrée-Anne Clément, Véronique Desgagné, Kathrine Thibeault, Frédérique White, Simon-Pierre Guay, Benoit J. Arsenault, et al. “Human Plasma Pregnancy-Associated miRNAs and Their Temporal Variation within the First Trimester of Pregnancy.” Reproductive Biology and Endocrinology : RB&E 20 (January 14, 2022): 14. https://doi.org/10.1186/s12958-021-00883-1.
Bernhardt, Grisilda Vidya, Pooja Shivappa, Kavitha Bernhardt, Sujatha Bhat, Janita R.T. Pinto, Malay Jhancy, and Suresh Kumar. “Markers of Inflammation in Obese Pregnant Women: Adenosine Deaminase and High Sensitive C – Reactive Protein.” European Journal of Obstetrics & Gynecology and Reproductive Biology: X 16 (October 14, 2022): 100167. https://doi.org/10.1016/j.eurox.2022.100167.
Carone, Benjamin R., Lucas Fauquier, Naomi Habib, Jeremy M. Shea, Caroline E. Hart, Ruowang Li, Christoph Bock, et al. “Paternally Induced Transgenerational Environmental Reprogramming of Metabolic Gene Expression in Mammals.” Cell 143, no. 7 (December 2010): 1084–96. https://doi.org/10.1016/j.cell.2010.12.008.
Mørkve Knudsen, Toril, Faisal I. Rezwan, Yu Jiang, Wilfried Karmaus, Cecilie Svanes, and John W. Holloway. “Transgenerational and Intergenerational Epigenetic Inheritance in Allergic Diseases.” Journal of Allergy and Clinical Immunology 142, no. 3 (September 1, 2018): 765–72. https://doi.org/10.1016/j.jaci.2018.07.007.
Stati, Gianmarco, Paolo Amerio, Mario Nubile, Silvia Sancilio, Francesco Rossi, and Roberta Di Pietro. “Concern about the Effectiveness of mRNA Vaccination Technology and Its Long-Term Safety: Potential Interference on miRNA Machinery.” International Journal of Molecular Sciences 24, no. 2 (January 11, 2023): 1404. https://doi.org/10.3390/ijms24021404.
Brook, Byron, Morgan Goetz, Valerie Duval, Romain Micol, and David J. Dowling. “mRNA Vaccines: miRNA-Based Controlled Biodistribution and Directed Adjuvantation.” Trends in Immunology 46, no. 5 (May 1, 2025): 357–60. https://doi.org/10.1016/j.it.2025.03.004.
Erhard, Florian, Jürgen Haas, Diana Lieber, Georg Malterer, Lukasz Jaskiewicz, Mihaela Zavolan, Lars Dölken, and Ralf Zimmer. “Widespread Context Dependency of microRNA-Mediated Regulation.” Genome Research 24, no. 6 (June 2014): 906–19. https://doi.org/10.1101/gr.166702.113.
Denzler, Rémy, Vikram Agarwal, Joanna Stefano, David P Bartel, and Markus Stoffel. “Assessing the ceRNA Hypothesis with Quantitative Measurements of miRNA and Target Abundance.” Molecular Cell 54, no. 5 (June 5, 2014): 766–76. https://doi.org/10.1016/j.molcel.2014.03.045.
Boniolo, Fabio, Markus Hoffmann, Norman Roggendorf, Bahar Tercan, Jan Baumbach, Mauro A A Castro, A Gordon Robertson, Dieter Saur, and Markus List. “spongEffects: ceRNA Modules Offer Patient-Specific Insights into the miRNA Regulatory Landscape.” Edited by Anthony Mathelier. Bioinformatics 39, no. 5 (May 4, 2023): btad276. https://doi.org/10.1093/bioinformatics/btad276.
Ayoub, Shymaa E., Olfat G. Shaker, Mohamed Masoud, Essam A. Hassan, Eman M. Ezzat, Mona I. Ahmed, Randa I. Ahmed, et al. “Altered Expression of Serum lncRNA CASC2 and miRNA-21-5p in COVID-19 Patients.” Human Genomics 18, no. 1 (February 12, 2024): 18. https://doi.org/10.1186/s40246-024-00578-9.
Karagkouni, Dimitra, Maria D Paraskevopoulou, Spyros Tastsoglou, Giorgos Skoufos, Anna Karavangeli, Vasilis Pierros, Elissavet Zacharopoulou, and Artemis G Hatzigeorgiou. “DIANA-LncBase v3: Indexing Experimentally Supported miRNA Targets on Non-Coding Transcripts.” Nucleic Acids Research, November 16, 2019, gkz1036. https://doi.org/10.1093/nar/gkz1036.
Ma, Benchi, Shihao Wang, Wenzheng Wu, Pufan Shan, Yufan Chen, Jiaqi Meng, Liping Xing, et al. “Mechanisms of circRNA/lncRNA-miRNA Interactions and Applications in Disease and Drug Research.” Biomedicine & Pharmacotherapy 162 (June 1, 2023): 114672. https://doi.org/10.1016/j.biopha.2023.114672.
Chen, Qing, Yidong Wang, Huimin Dang, and Xiaoling Wu. “MicroRNA‑148a‑3p Inhibits the Proliferation of Cervical Cancer Cells by Regulating the Expression Levels of DNMT1 and UTF1.” Oncology Letters 22, no. 2 (August 1, 2021): 1–9. https://doi.org/10.3892/ol.2021.12878.
Friedrich, Michael, Katharina Pracht, Mir‐Farzin Mashreghi, Hans‐Martin Jäck, Andreas Radbruch, and Barbara Seliger. “The Role of the miR‐148/‐152 Family in Physiology and Disease.” European Journal of Immunology 47, no. 12 (December 2017): 2026–38. https://doi.org/10.1002/eji.201747132.
Drillis, Georgios, Maria Goulielmaki, Demetrios A. Spandidos, Sofia Aggelaki, and Vassilios Zoumpourlis. “Non‑coding RNAs (miRNAs and lncRNAs) and Their Roles in Lymphogenesis in All Types of Lymphomas and Lymphoid Malignancies (Review).” Oncology Letters 21, no. 5 (May 1, 2021): 1–17. https://doi.org/10.3892/ol.2021.12654.
Dionysiou, Margarita, Yiouli P. Ktena, Han-Hsuan Fu, Azeb Haile, Demetrios Garbis, Sarah Watt, Shawn Lupold, and Kenneth R. Cooke. “Role of Mir-21 in Allogeneic T-Cell Responses Post-HCT.” Transplantation and Cellular Therapy 30, no. 2 (February 2024): S255–56. https://doi.org/10.1016/j.jtct.2023.12.340.
Brieño-Enríquez, Miguel A., Jesús García-López, David B. Cárdenas, Sylvain Guibert, Elouan Cleroux, Lukas Děd, Juan de Dios Hourcade, Jana Pěknicová, Michael Weber, and Jesús del Mazo. “Exposure to Endocrine Disruptor Induces Transgenerational Epigenetic Deregulation of MicroRNAs in Primordial Germ Cells.” PLoS ONE 10, no. 4 (April 21, 2015): e0124296. https://doi.org/10.1371/journal.pone.0124296.
Maccani, Matthew A., James F. Padbury, and Carmen J. Marsit. “miR-16 and miR-21 Expression in the Placenta Is Associated with Fetal Growth.” PLoS ONE 6, no. 6 (June 15, 2011): e21210. https://doi.org/10.1371/journal.pone.0021210.
Fujisawa, Akinori. “Increased Age-Adjusted Cancer Mortality after the 3rd mRNA-Lipid Nanoparticle Vaccine Dose during the COVID-19 Pandemic in Japan,” January 1, 2023. https://doi.org/10.5281/zenodo.8352450.
Bilger, Andrea, and Paul F. Lambert. “Rapid-Onset Cancer.” Tumour Virus Research 19 (June 1, 2025): 200312. https://doi.org/10.1016/j.tvr.2024.200312.
You, Mengshu, Zhuolin Xie, Nan Zhang, Yixuan Zhang, Desheng Xiao, Shuang Liu, Wei Zhuang, Lili Li, and Yongguang Tao. “Signaling Pathways in Cancer Metabolism: Mechanisms and Therapeutic Targets.” Signal Transduction and Targeted Therapy 8, no. 1 (May 10, 2023): 1–27. https://doi.org/10.1038/s41392-023-01442-3.
Tartaglia, Marco, and Bruce D. Gelb. “Disorders of Dysregulated Signal Traffic through the RAS-MAPK Pathway: Phenotypic Spectrum and Molecular Mechanisms.” Annals of the New York Academy of Sciences 1214 (December 2010): 99–121. https://doi.org/10.1111/j.1749-6632.2010.05790.x.
Bahar, Md Entaz, Hyun Joon Kim, and Deok Ryong Kim. “Targeting the RAS/RAF/MAPK Pathway for Cancer Therapy: From Mechanism to Clinical Studies.” Signal Transduction and Targeted Therapy 8, no. 1 (December 18, 2023): 1–38. https://doi.org/10.1038/s41392-023-01705-z.
Seger, Rony. “Special Issue: MAPK Signaling Cascades in Human Health and Diseases.” International Journal of Molecular Sciences 25, no. 20 (October 18, 2024): 11226. https://doi.org/10.3390/ijms252011226.
Lambert, Samuel A., Arttu Jolma, Laura F. Campitelli, Pratyush K. Das, Yimeng Yin, Mihai Albu, Xiaoting Chen, Jussi Taipale, Timothy R. Hughes, and Matthew T. Weirauch. “The Human Transcription Factors.” Cell 172, no. 4 (February 2018): 650–65. https://doi.org/10.1016/j.cell.2018.01.029.
Liu, Ting, Lingyun Zhang, Donghyun Joo, and Shao-Cong Sun. “NF-κB Signaling in Inflammation.” Signal Transduction and Targeted Therapy 2, no. 1 (July 14, 2017): 1–9. https://doi.org/10.1038/sigtrans.2017.23.
Esnault, Cyril, Francesco Gualdrini, Stuart Horswell, Gavin Kelly, Aengus Stewart, Phil East, Nik Matthews, and Richard Treisman. “ERK-Induced Activation of TCF Family of SRF Cofactors Initiates a Chromatin Modification Cascade Associated with Transcription.” Molecular Cell 65, no. 6 (March 16, 2017): 1081-1095.e5. https://doi.org/10.1016/j.molcel.2017.02.005.
Luu, Winnie, Laura J. Sharpe, Julian Stevenson, and Andrew J. Brown. “Akt Acutely Activates the Cholesterogenic Transcription Factor SREBP-2.” Biochimica Et Biophysica Acta 1823, no. 2 (February 2012): 458–64. https://doi.org/10.1016/j.bbamcr.2011.09.017.
Li, Jing, Fan Yang, Feng Wei, and Xiubao Ren. “The Role of Toll-like Receptor 4 in Tumor Microenvironment.” Oncotarget 8, no. 39 (July 8, 2017): 66656–67. https://doi.org/10.18632/oncotarget.19105.
Zheng, Xin, Song Li, and Hui Yang. “Roles of Toll-Like Receptor 3 in Human Tumors.” Frontiers in Immunology 12 (April 27, 2021): 667454. https://doi.org/10.3389/fimmu.2021.667454.
Yuan, Ziyi, Ruyu Yan, Zuyi Fu, Tao Wu, and Chaoxiu Ren. “Impact of Physicochemical Properties on Biological Effects of Lipid Nanoparticles: Are They Completely Safe.” Science of The Total Environment 927 (June 1, 2024): 172240. https://doi.org/10.1016/j.scitotenv.2024.172240.
Feng, Yawen, Hongying Fu, Xing Zhang, Suqin Liu, and Xiaoran Wei. “Lysosome Toxicities Induced by Nanoparticle Exposure and Related Mechanisms.” Ecotoxicology and Environmental Safety 286 (November 1, 2024): 117215. https://doi.org/10.1016/j.ecoenv.2024.117215.
Cui, Shaohui, Yueying Wang, Yan Gong, Xiao Lin, Yinan Zhao, Defu Zhi, Quan Zhou, and Shubiao Zhang. “Correlation of the Cytotoxic Effects of Cationic Lipids with Their Headgroups.” Toxicology Research 7, no. 3 (March 22, 2018): 473–79. https://doi.org/10.1039/c8tx00005k.
Azarnezhad, Asaad, Hadi Samadian, Mehdi Jaymand, Mahsa Sobhani, and Amirhossein Ahmadi. “Toxicological Profile of Lipid-Based Nanostructures: Are They Considered as Completely Safe Nanocarriers?” Critical Reviews in Toxicology 50, no. 2 (February 2020): 148–76. https://doi.org/10.1080/10408444.2020.1719974.
Sun, Da, and Zheng-Rong Lu. “Structure and Function of Cationic and Ionizable Lipids for Nucleic Acid Delivery.” Pharmaceutical Research 40, no. 1 (2023): 27–46. https://doi.org/10.1007/s11095-022-03460-2.
Zamfir, Maria-Alexandra, Liliana Moraru, Camelia Dobrea, Andreea-Elena Scheau, Simona Iacob, Cosmin Moldovan, Cristian Scheau, Constantin Caruntu, and Ana Caruntu. “Hematologic Malignancies Diagnosed in the Context of the mRNA COVID-19 Vaccination Campaign: A Report of Two Cases.” Medicina (Kaunas, Lithuania) 58, no. 7 (June 30, 2022): 874. https://doi.org/10.3390/medicina58070874.
Tachita, Takuto, Takenori Takahata, Satoru Yamashita, Toru Ebina, Kosuke Kamata, Kazufumi Yamagata, Yoshiko Tamai, and Hirotake Sakuraba. “Newly Diagnosed Extranodal NK/T-Cell Lymphoma, Nasal Type, at the Injected Left Arm after BNT162b2 mRNA COVID-19 Vaccination.” International Journal of Hematology 118, no. 4 (October 1, 2023): 503–7. https://doi.org/10.1007/s12185-023-03607-w.
Sogabe, Nobuhiro, Masatomo Kuno, Yu Nakagama, Yosuke Makuuchi, Naonori Harada, Teruhito Takakuwa, Hiroshi Okamura, et al. “[Warm autoimmune hemolytic anemia and IgM-monoclonal gammopathy following BNT162b2 COVID-19 vaccine in a patient with splenic marginal zone lymphoma].” [Rinsho Ketsueki] The Japanese Journal of Clinical Hematology 63, no. 10 (2022): 1379–85. https://doi.org/10.11406/rinketsu.63.1379.
Sekizawa, Akinori, Kenichi Hashimoto, Shinichi Kobayashi, Sawako Kozono, Takahiro Kobayashi, Yusuke Kawamura, Motohiro Kimata, et al. “Rapid Progression of Marginal Zone B-Cell Lymphoma after COVID-19 Vaccination (BNT162b2): A Case Report.” Frontiers in Medicine 9 (August 1, 2022). https://doi.org/10.3389/fmed.2022.963393.
O´Sullivan, C., F. Zach, T. Moser, G. Pilz, A. Harrer, E. Trinka, C. Enzinger, J. A. R. Pfaff, and P. Wipfler. “Misinterpretation of Glioblastoma as ADEM: Potentially Harmful Consequences of over-Diagnosis of COVID-19 Vaccine-Associated Adverse Events.” Journal of Neurology 269, no. 2 (2022): 616–18. https://doi.org/10.1007/s00415-021-10707-2.
Mizutani, M., H. Mitsui, T. Amano, Y. Ogawa, N. Deguchi, S. Shimada, A. Miwa, T. Kawamura, and Y. Ogido. “Two Cases of Axillary Lymphadenopathy Diagnosed as Diffuse Large B-Cell Lymphoma Developed Shortly after BNT162b2 COVID-19 Vaccination.” Journal of the European Academy of Dermatology and Venereology: JEADV 36, no. 8 (August 2022): e613–15. https://doi.org/10.1111/jdv.18136.
Kyriakopoulos, Anthony M., Greg Nigh, Peter A. McCullough, Maria D. Olivier, and Stephanie Seneff. “Bell’s Palsy or an Aggressive Infiltrating Basaloid Carcinoma Post-mRNA Vaccination for COVID-19? : A Case Report and Review of the Literature.” EXCLI Journal 22 (September 15, 2023): 992–1011. https://doi.org/10.17179/excli2023-6145.
Kim, Soo Ryang, Soo Ki Kim, Takako Fujii, Hisato Kobayashi, Toyokazu Okuda, Takanobu Hayakumo, Atsushi Nakai, et al. “Drug-Induced Sarcoidosis-like Reaction Three Months after BNT162b2 mRNA COVID-19 Vaccination: A Case Report and Review of Literature.” World Journal of Clinical Cases 11, no. 1 (January 6, 2023): 177–86. https://doi.org/10.12998/wjcc.v11.i1.177.
Kaimori, Ryo, Haruto Nishida, Tomohisa Uchida, Mari Tamura, Kohji Kuroki, Kumi Murata, Kinta Hatakeyama, et al. “Histopathologically TMA-like Distribution of Multiple Organ Thromboses Following the Initial Dose of the BNT162b2 mRNA Vaccine (Comirnaty, Pfizer/BioNTech): An Autopsy Case Report.” Thrombosis Journal 20, no. 1 (October 6, 2022): 61. https://doi.org/10.1186/s12959-022-00418-7.
Govind Babu, K, and Gita R Bhat. “Cancer-Associated Thrombotic Microangiopathy.” Ecancermedicalscience 10 (June 28, 2016): 649. https://doi.org/10.3332/ecancer.2016.649.
Goldman, Serge, Dominique Bron, Thomas Tousseyn, Irina Vierasu, Laurent Dewispelaere, Pierre Heimann, Elie Cogan, and Michel Goldman. “Rapid Progression of Angioimmunoblastic T Cell Lymphoma Following BNT162b2 mRNA Vaccine Booster Shot: A Case Report.” Frontiers in Medicine 8 (November 25, 2021). https://doi.org/10.3389/fmed.2021.798095.
Cohen, Dan, Shir Hazut Krauthammer, Yael C. Cohen, Chava Perry, Irit Avivi, Yair Herishanu, and Einat Even-Sapir. “Correlation between BNT162b2 mRNA Covid-19 Vaccine-Associated Hypermetabolic Lymphadenopathy and Humoral Immunity in Patients with Hematologic Malignancy.” European Journal of Nuclear Medicine and Molecular Imaging 48, no. 11 (October 1, 2021): 3540–49. https://doi.org/10.1007/s00259-021-05389-x.
Cavanna, Luigi, Sergio Ottavio Grassi, Livia Ruffini, Emanuele Michieletti, Egidio Carella, Dante Palli, Adriano Zangrandi, et al. “Non-Hodgkin Lymphoma Developed Shortly after mRNA COVID-19 Vaccination: Report of a Case and Review of the Literature.” Medicina 59, no. 1 (January 12, 2023): 157. https://doi.org/10.3390/medicina59010157.
Ahmed, Fahad, Nashwah Memon, and Adel Haque. “Multiple Keratoacanthomas Following Moderna Messenger RNA-1273 COVID-19 Vaccination Resolved With 5-Fluorouracil Treatment: Case Report.” JMIR Dermatology 5, no. 4 (December 1, 2022): e41739. https://doi.org/10.2196/41739.
Usmani, Shariq M., and Thorsten R. Mempel. “Cancer Cells Relax and Resist Cytotoxic Attack.” Immunity 54, no. 5 (May 11, 2021): 853–55. https://doi.org/10.1016/j.immuni.2021.04.017.
Arasanz, Hugo, Miren Zuazo, Ana Bocanegra, Luisa Chocarro, Ester Blanco, Maite Martínez, Idoia Morilla, et al. “Hyperprogressive Disease: Main Features and Key Controversies.” International Journal of Molecular Sciences 22, no. 7 (April 3, 2021): 3736. https://doi.org/10.3390/ijms22073736.
Kraaij, Marina D., Nigel D. L. Savage, Sandra W. Van Der Kooij, Karin Koekkoek, Jun Wang, J. Merlijn Van Den Berg, Tom H. M. Ottenhoff, et al. “Induction of Regulatory T Cells by Macrophages Is Dependent on Production of Reactive Oxygen Species.” Proceedings of the National Academy of Sciences 107, no. 41 (October 12, 2010): 17686–91. https://doi.org/10.1073/pnas.1012016107.
Ma, Xueqian, Yu Gao, Yanlei Chen, Jinhao Liu, Chunyu Yang, Cuifen Bao, Yanping Wang, Yang Feng, Xiaoyu Song, and Shifeng Qiao. “M2-Type Macrophages Induce Tregs Generation by Activating the TGF-β/Smad Signalling Pathway to Promote Colorectal Cancer Development.” OncoTargets and Therapy Volume 14 (December 2021): 5391–5402. https://doi.org/10.2147/OTT.S336548.
Zhong, Hui, and Karina Yazdanbakhsh. “Differential Control of Helios+/− Treg Development by Monocyte Subsets through Disparate Inflammatory Cytokines.” Blood 121, no. 13 (March 28, 2013): 2494–2502. https://doi.org/10.1182/blood-2012-11-469122.
Loacker, Lorin, Janine Kimpel, Zoltán Bánki, Christoph Q. Schmidt, Andrea Griesmacher, and Markus Anliker. “Increased PD-L1 Surface Expression on Peripheral Blood Granulocytes and Monocytes after Vaccination with SARS-CoV2 mRNA or Vector Vaccine.” Clinical Chemistry and Laboratory Medicine (CCLM) 61, no. 1 (January 1, 2023): e17–19. https://doi.org/10.1515/cclm-2022-0787.
Barmada, Anis, Jon Klein, Anjali Ramaswamy, Nina N. Brodsky, Jillian R. Jaycox, Hassan Sheikha, Kate M. Jones, et al. “Cytokinopathy with Aberrant Cytotoxic Lymphocytes and Profibrotic Myeloid Response in SARS-CoV-2 mRNA Vaccine–Associated Myocarditis.” Science Immunology 8, no. 83 (May 26, 2023): eadh3455. https://doi.org/10.1126/sciimmunol.adh3455.
Cuccaro, Annarosa, Silvia Bellesi, Eugenio Galli, Ilaria Zangrilli, Francesco Corrente, Elisa Cupelli, Federica Fatone, et al. “PD-L1 Expression in Peripheral Blood Granulocytes at Diagnosis as Prognostic Factor in Classical Hodgkin Lymphoma.” Journal of Leukocyte Biology 112, no. 3 (August 24, 2022): 539–45. https://doi.org/10.1002/JLB.5AB0121-041R.
Guydosh, Nicholas R., and Rachel Green. “Translation of Poly(A) Tails Leads to Precise mRNA Cleavage.” RNA 23, no. 5 (May 2017): 749–61. https://doi.org/10.1261/rna.060418.116.
Fu, Min, Jianmei Gu, Maoye Wang, Jiahui Zhang, Yanke Chen, Pengcheng Jiang, Taofeng Zhu, and Xu Zhang. “Emerging Roles of tRNA-Derived Fragments in Cancer.” Molecular Cancer 22, no. 1 (February 13, 2023): 30. https://doi.org/10.1186/s12943-023-01739-5.
Nishimura, Kazuki, Kiyoshi Takahara, Kazumasa Komura, Mitsuaki Ishida, Kensuke Hirosuna, Ryoichi Maenosono, Masahiko Ajiro, et al. “Mechanistic Insights into Lethal Hyper Progressive Disease Induced by PD-L1 Inhibitor in Metastatic Urothelial Carcinoma.” Npj Precision Oncology 8, no. 1 (September 17, 2024): 1–8. https://doi.org/10.1038/s41698-024-00707-6.
Soltani, Mojdeh, Mohammad Abbaszadeh, Hamed Fouladseresht, Mark J. M. Sullman, and Nahid Eskandari. “PD-L1 Importance in Malignancies Comprehensive Insights into the Role of PD-L1 in Malignancies: From Molecular Mechanisms to Therapeutic Opportunities.” Clinical and Experimental Medicine 25, no. 1 (April 3, 2025): 106. https://doi.org/10.1007/s10238-025-01641-y.
Yu, Wendan, Yijun Hua, Huijuan Qiu, Jiaojiao Hao, Kun Zou, Zongjuan Li, Sheng Hu, et al. “PD-L1 Promotes Tumor Growth and Progression by Activating WIP and β-Catenin Signaling Pathways and Predicts Poor Prognosis in Lung Cancer.” Cell Death & Disease 11, no. 7 (July 6, 2020): 1–16. https://doi.org/10.1038/s41419-020-2701-z.
Cheon, HyeonJoo, Elise G. Holvey-Bates, Daniel J. McGrail, and George R. Stark. “PD-L1 Sustains Chronic, Cancer Cell–Intrinsic Responses to Type I Interferon, Enhancing Resistance to DNA Damage.” Proceedings of the National Academy of Sciences of the United States of America 118, no. 47 (November 23, 2021): e2112258118. https://doi.org/10.1073/pnas.2112258118.
Roy, Shalini, Anand K Gupta, Madhurima Banerjee, Pijush K. Das, and Anindita Ukil. “PD-1 Negatively Tunes Macrophage Immune Activation by Turning off JNK and STAT1 Signaling: Exploited by Leishmania for Its Intra-Macrophage Survival.” Cellular Immunology 391–392 (September 1, 2023): 104758. https://doi.org/10.1016/j.cellimm.2023.104758.
Lin, Xin, Kuan Kang, Pan Chen, Zhaoyang Zeng, Guiyuan Li, Wei Xiong, Mei Yi, and Bo Xiang. “Regulatory Mechanisms of PD-1/PD-L1 in Cancers.” Molecular Cancer 23 (May 18, 2024): 108. https://doi.org/10.1186/s12943-024-02023-w.
Stutvoet, Thijs S, Arjan Kol, Elisabeth GE de Vries, Marco de Bruyn, Rudolf SN Fehrmann, Anton GT Terwisscha van Scheltinga, and Steven de Jong. “MAPK Pathway Activity Plays a Key Role in PD‐L1 Expression of Lung Adenocarcinoma Cells.” The Journal of Pathology 249, no. 1 (September 2019): 52–64. https://doi.org/10.1002/path.5280.
Xu, Ruyue, Xinkuang Liu, Amin Li, Li Song, Jiaojiao Liang, Jiafeng Gao, and Xiaolong Tang. “C-Met up-Regulates the Expression of PD-L1 through MAPK/NF-κBp65 Pathway.” Journal of Molecular Medicine 100, no. 4 (April 1, 2022): 585–98. https://doi.org/10.1007/s00109-022-02179-2.
Liu, Zongcai, Fen Ning, Yanna Cai, Huiying Sheng, Ruidan Zheng, Xi Yin, Zhikun Lu, et al. “The EGFR‐P38 MAPK Axis Up‐regulates PD‐L1 through miR‐675‐5p and Down‐regulates HLA‐ABC via Hexokinase‐2 in Hepatocellular Carcinoma Cells.” Cancer Communications 41, no. 1 (January 2021): 62–78. https://doi.org/10.1002/cac2.12117.
Erdoğdu, Batuhan, Ozan Kaplan, Bilge Başak Fidan, Mustafa Çelebier, Ümit Yavuz Malkan, and Ibrahim C. Haznedaroglu. “Metabolomic Profiling of Leukemic Hematopoiesis: Effects of BNT162b2 mRNA COVID-19 Vaccine Administration.” Current Molecular Medicine, June 3, 2025. https://doi.org/10.2174/0115665240361878250601074746.
Tjong, Elysia, Manjari Dimri, and Shamim S. Mohiuddin. “Biochemistry, Tetrahydrofolate.” In StatPearls. Treasure Island (FL): StatPearls Publishing, 2025. http://www.ncbi.nlm.nih.gov/books/NBK539712/.
“Tetrahydrofolic Acid - an Overview | ScienceDirect Topics.” Accessed June 17, 2025. https://www.sciencedirect.com/topics/medicine-and-dentistry/tetrahydrofolic-acid.
Mullen, Nicholas J., and Pankaj K. Singh. “Nucleotide Metabolism: A Pan-Cancer Metabolic Dependency.” Nature Reviews Cancer 23, no. 5 (May 2023): 275–94. https://doi.org/10.1038/s41568-023-00557-7.
Watanabe, M., T. Tomita, and T. Yasuda. “Membrane-Damaging Action of Staphylococcal Alpha-Toxin on Phospholipid-Cholesterol Liposomes.” Biochimica Et Biophysica Acta 898, no. 3 (April 23, 1987): 257–65. https://doi.org/10.1016/0005-2736(87)90065-4.
Andrews, Norma W., Patricia E. Almeida, and Matthias Corrotte. “Damage Control: Cellular Mechanisms of Plasma Membrane Repair.” Trends in Cell Biology 24, no. 12 (December 2014): 734–42. https://doi.org/10.1016/j.tcb.2014.07.008.
Sønder, Stine Lauritzen, Swantje Christin Häger, Anne Sofie Busk Heitmann, Lisa B. Frankel, Catarina Dias, Adam Cohen Simonsen, and Jesper Nylandsted. “Restructuring of the Plasma Membrane upon Damage by LC3-Associated Macropinocytosis.” Science Advances 7, no. 27 (July 2, 2021): eabg1969. https://doi.org/10.1126/sciadv.abg1969.
Huang, Nai-Jia, Ying-Cing Lin, Chung-Yueh Lin, Novalia Pishesha, Caroline A. Lewis, Elizaveta Freinkman, Colin Farquharson, José Luis Millán, and Harvey Lodish. “Enhanced Phosphocholine Metabolism Is Essential for Terminal Erythropoiesis.” Blood 131, no. 26 (June 28, 2018): 2955–66. https://doi.org/10.1182/blood-2018-03-838516.
Vettore, Lisa, Rebecca L. Westbrook, and Daniel A. Tennant. “New Aspects of Amino Acid Metabolism in Cancer.” British Journal of Cancer 122, no. 2 (January 2020): 150–56. https://doi.org/10.1038/s41416-019-0620-5.
Rogers, Robert S., Rohit Sharma, Hardik B. Shah, Owen S. Skinner, Xiaoyan A. Guo, Apekshya Panda, Rahul Gupta, et al. “Circulating N-Lactoyl-Amino Acids and N-Formyl-Methionine Reflect Mitochondrial Dysfunction and Predict Mortality in Septic Shock.” Metabolomics 20, no. 2 (2024): 36. https://doi.org/10.1007/s11306-024-02089-z.
Han, Guanghui, Weizhe Zhen, Yuan Dai, Hongni Yu, Dongyue Li, and Tao Ma. “Dihuang-Yinzi Alleviates Cognition Deficits via Targeting Energy-Related Metabolism in an Alzheimer Mouse Model as Demonstrated by Integration of Metabolomics and Network Pharmacology.” Frontiers in Aging Neuroscience 14 (April 1, 2022). https://doi.org/10.3389/fnagi.2022.873929.
Van Meer, Gerrit, Dennis R. Voelker, and Gerald W. Feigenson. “Membrane Lipids: Where They Are and How They Behave.” Nature Reviews Molecular Cell Biology 9, no. 2 (February 2008): 112–24. https://doi.org/10.1038/nrm2330.
Hou, Xiaoting, Yu Chen, Noah D. Carrillo, Vincent L. Cryns, Richard A. Anderson, Jichao Sun, Songlin Wang, and Mo Chen. “Phosphoinositide Signaling at the Cytoskeleton in the Regulation of Cell Dynamics.” Cell Death & Disease 16, no. 1 (April 14, 2025): 1–12. https://doi.org/10.1038/s41419-025-07616-x.
Krajnik, Amanda, Joseph A. Brazzo, Kalyanaraman Vaidyanathan, Tuhin Das, Javier Redondo-Muñoz, and Yongho Bae. “Phosphoinositide Signaling and Mechanotransduction in Cardiovascular Biology and Disease.” Frontiers in Cell and Developmental Biology 8 (December 14, 2020). https://doi.org/10.3389/fcell.2020.595849.
Oh, Byung-Chul. “Phosphoinositides and Intracellular Calcium Signaling: Novel Insights into Phosphoinositides and Calcium Coupling as Negative Regulators of Cellular Signaling.” Experimental & Molecular Medicine 55, no. 8 (August 2023): 1702–12. https://doi.org/10.1038/s12276-023-01067-0.
Manna, Prasenjit, and Sushil K. Jain. “Phosphatidylinositol-3,4,5-Triphosphate and Cellular Signaling: Implications for Obesity and Diabetes.” Cellular Physiology and Biochemistry : International Journal of Experimental Cellular Physiology, Biochemistry, and Pharmacology 35, no. 4 (2015): 1253–75. https://doi.org/10.1159/000373949.
Tanaka, Manami, Tomoo Tanaka, Xiaolong Zhu, Fei Teng, Hong Lin, Zhu Luo, Ying Pan, et al. “Huaier Effects on Functional Compensation with Destructive Ribosomal RNA Structure after Anti-SARS-CoV-2 mRNA Vaccination.” Archives of Clinical and Biomedical Research 6, no. 3 (June 22, 2022): 553–74.
Lari, Arezou, Hamid Gholami Pourbadie, Ali Sharifi-Zarchi, Maryam Akhtari, Leila Nejatbakhsh Samimi, Ahmadreza Jamshidi, and Mahdi Mahmoudi. “Dysregulation of Ribosome-Related Genes in Ankylosing Spondylitis: A Systems Biology Approach and Experimental Method.” BMC Musculoskeletal Disorders 22 (September 14, 2021): 789. https://doi.org/10.1186/s12891-021-04662-2.
Jiao, Lijuan, Yuzhe Liu, Xi-Yong Yu, Xiangbin Pan, Yu Zhang, Junchu Tu, Yao-Hua Song, and Yangxin Li. “Ribosome Biogenesis in Disease: New Players and Therapeutic Targets.” Signal Transduction and Targeted Therapy 8 (January 9, 2023): 15. https://doi.org/10.1038/s41392-022-01285-4.
Moghimi, Seyed Moein, and Dmitri Simberg. “Pro-Inflammatory Concerns with Lipid Nanoparticles.” Molecular Therapy 30, no. 6 (June 1, 2022): 2109–10. https://doi.org/10.1016/j.ymthe.2022.04.011.
Thiaville, Michelle M., Yuan-Xiang Pan, Altin Gjymishka, Can Zhong, Randal J. Kaufman, and Michael S. Kilberg. “MEK Signaling Is Required for Phosphorylation of eIF2α Following Amino Acid Limitation of HepG2 Human Hepatoma Cells.” The Journal of Biological Chemistry 283, no. 16 (April 18, 2008): 10848–57. https://doi.org/10.1074/jbc.M708320200.
Quevedo, Celia, Matilde Salinas, and Alberto Alcázar. “Initiation Factor 2B Activity Is Regulated by Protein Phosphatase 1, Which Is Activated by the Mitogen-Activated Protein Kinase-Dependent Pathway in Insulin-like Growth Factor 1-Stimulated Neuronal Cells.” Journal of Biological Chemistry 278, no. 19 (May 2003): 16579–86. https://doi.org/10.1074/jbc.M212936200.
Parveen, Sana, Haripriya Parthasarathy, Dhiviya Vedagiri, Divya Gupta, Hitha Gopalan Nair, and Krishnan Harinivas Harshan. “Regulation of eIF2α Phosphorylation by MAPKs Influences Polysome Stability and Protein Translation,” August 30, 2021. https://doi.org/10.1101/2021.08.30.458160.
Zhang, Liguo, Alexsia Richards, M. Inmaculada Barrasa, Stephen H. Hughes, Richard A. Young, and Rudolf Jaenisch. “Reverse-Transcribed SARS-CoV-2 RNA Can Integrate into the Genome of Cultured Human Cells and Can Be Expressed in Patient-Derived Tissues.” Proceedings of the National Academy of Sciences 118, no. 21 (May 25, 2021): e2105968118. https://doi.org/10.1073/pnas.2105968118.
Zhang, Liguo, Punam Bisht, Anthony Flamier, M. Inmaculada Barrasa, Max Friesen, Alexsia Richards, Stephen H. Hughes, and Rudolf Jaenisch. “LINE1-Mediated Reverse Transcription and Genomic Integration of SARS-CoV-2 mRNA Detected in Virus-Infected but Not in Viral mRNA-Transfected Cells.” Viruses 15, no. 3 (February 25, 2023): 629. https://doi.org/10.3390/v15030629.
Sattar, Sarah, Juraj Kabat, Kailey Jerome, Friederike Feldmann, Kristina Bailey, and Masfique Mehedi. “Nuclear Translocation of Spike mRNA and Protein Is a Novel Feature of SARS-CoV-2.” Frontiers in Microbiology 14 (January 26, 2023). https://doi.org/10.3389/fmicb.2023.1073789.
Domazet-Lošo, Tomislav. “mRNA Vaccines: Why Is the Biology of Retroposition Ignored?” Genes 13, no. 5 (April 20, 2022): 719. https://doi.org/10.3390/genes13050719.
Aldén, Markus, Francisko Olofsson Falla, Daowei Yang, Mohammad Barghouth, Cheng Luan, Magnus Rasmussen, and Yang De Marinis. “Intracellular Reverse Transcription of Pfizer BioNTech COVID-19 mRNA Vaccine BNT162b2 In Vitro in Human Liver Cell Line.” Current Issues in Molecular Biology 44, no. 3 (February 25, 2022): 1115–26. https://doi.org/10.3390/cimb44030073.
Chandrasekharappa, S C, and K N Subramanian. “Effects of Position and Orientation of the 72-Base-Pair-Repeat Transcriptional Enhancer on Replication from the Simian Virus 40 Core Origin.” Journal of Virology 61, no. 10 (October 1987): 2973–80. https://doi.org/10.1128/jvi.61.10.2973-2980.1987.
Dean, David A., Brenda S. Dean, Susanne Muller, and Louis C. Smith. “Sequence Requirements for Plasmid Nuclear Import.” Experimental Cell Research 253, no. 2 (December 15, 1999): 713–22. https://doi.org/10.1006/excr.1999.4716.
Strayer, David S, Francisco Branco, Mark A Zern, Priscilla Yam, Sandra A Calarota, Carmen N Nichols, John A Zaia, et al. “Durability of Transgene Expression and Vector Integration: Recombinant SV40-Derived Gene Therapy Vectors.” Molecular Therapy 6, no. 2 (August 2002): 227–37. https://doi.org/10.1006/mthe.2002.0657.
Lim, Samuel, R. Rogers Yocum, Pamela A. Silver, and Jeffrey C. Way. “High Spontaneous Integration Rates of End-Modified Linear DNAs upon Mammalian Cell Transfection.” Scientific Reports 13, no. 1 (April 26, 2023): 6835. https://doi.org/10.1038/s41598-023-33862-0.
Šenigl, Filip, Anni I. Soikkeli, Salomé Prost, David G. Schatz, Martina Slavková, Jiří Hejnar, and Jukka Alinikula. “The SV40 Virus Enhancer Functions as a Somatic Hypermutation-Targeting Element with Potential Tumorigenic Activity.” Tumour Virus Research 18 (December 1, 2024): 200293. https://doi.org/10.1016/j.tvr.2024.200293.
Baker, Meghan A., David C. Kaelber, David S. Bar-Shain, Pedro L. Moro, Bob Zambarano, Megan Mazza, Crystal Garcia, Adam Henry, Richard Platt, and Michael Klompas. “Advanced Clinical Decision Support for Vaccine Adverse Event Detection and Reporting.” Clinical Infectious Diseases : An Official Publication of the Infectious Diseases Society of America 61, no. 6 (September 15, 2015): 864–70. https://doi.org/10.1093/cid/civ430.
Pethő, Ákos, Deján Dobi, Magdolna Kardos, and Karolina Schnabel. “Unexpected Renal Side Effects of mRNA COVID-19 Vaccines; a Single-Center Experience and Short Review.” The American Journal of the Medical Sciences 369, no. 6 (June 1, 2025): 739–44. https://doi.org/10.1016/j.amjms.2025.01.002.
Haj, Majd, Yann Frey, Amit Levon, Avishai Maliah, Tal Ben-Yishay, Rachel Slutsky, Riham Smoom, et al. “The cGAS–STING, P38 MAPK, and P53 Pathways Link Genome Instability to Accelerated Cellular Senescence in ATM-Deficient Murine Lung Fibroblasts.” Proceedings of the National Academy of Sciences 122, no. 2 (January 14, 2025): e2419196122. https://doi.org/10.1073/pnas.2419196122.
Zhou, Xiaogang, Jing Wang, Lu Yu, Gan Qiao, Dalian Qin, Betty Yuen-Kwan Law, Fang Ren, Jianming Wu, and Anguo Wu. “Mitophagy and cGAS–STING Crosstalk in Neuroinflammation.” Acta Pharmaceutica Sinica. B 14, no. 8 (August 2024): 3327–61. https://doi.org/10.1016/j.apsb.2024.05.012.
Small, Sara H., E. Jessica Tang, Ryan L. Ragland, Yaroslava Ruzankina, David W. Schoppy, Rahul S. Mandal, M. Rebecca Glineburg, et al. “Induction of IL19 Expression through JNK and cGAS-STING Modulates DNA Damage–Induced Cytokine Production.” Science Signaling 14, no. 714 (December 21, 2021): eaba2611. https://doi.org/10.1126/scisignal.aba2611.
Wu, Tinghuai, Junjie Xing, and Anna A. Birukova. “Cell-Type-Specific Crosstalk between P38 MAPK and Rho Signaling in Lung Micro- and Macrovascular Barrier Dysfunction Induced by Staphylococcus Aureus-Derived Pathogens.” Translational Research: The Journal of Laboratory and Clinical Medicine 162, no. 1 (July 2013): 45–55. https://doi.org/10.1016/j.trsl.2013.03.005.
Zhang, Qian, Hong Wang, Yue-Jin Yang, Qiu-Ting Dong, Tian-Jie Wang, Hai-Yan Qian, Na Li, Xi-Mei Wang, and Chen Jin. “Atorvastatin Treatment Improves the Effects of Mesenchymal Stem Cell Transplantation on Acute Myocardial Infarction: The Role of the RhoA/ROCK/ERK Pathway.” International Journal of Cardiology 176, no. 3 (October 20, 2014): 670–79. https://doi.org/10.1016/j.ijcard.2014.07.071.
Soriano, Olga, Marta Alcón-Pérez, Miguel Vicente-Manzanares, and Esther Castellano. “The Crossroads between RAS and RHO Signaling Pathways in Cellular Transformation, Motility and Contraction.” Genes 12, no. 6 (May 27, 2021): 819. https://doi.org/10.3390/genes12060819.
Naka, Takashi, Mariko Hatakeyama, Naoto Sakamoto, and Akihiko Konagaya. “Compensation Effect of the MAPK Cascade on Formation of Phospho-Protein Gradients.” Bio Systems 83, no. 2–3 (2006): 167–77. https://doi.org/10.1016/j.biosystems.2005.06.015.
Michmerhuizen, Nicole L., Elizabeth Leonard, Aditi Kulkarni, and J. Chad Brenner. “Differential Compensation Mechanisms Define Resistance to PI3K Inhibitors in PIK3CA Amplified HNSCC.” Otorhinolaryngology-Head and Neck Surgery 1, no. 2 (2016): 44–50. https://doi.org/10.15761/ohns.1000111.
Hishida, Tomoaki, Yutaka Nakachi, Yosuke Mizuno, Miyuki Katano, Yasushi Okazaki, Masatsugu Ema, Satoru Takahashi, et al. “Functional Compensation between Myc and PI3K Signaling Supports Self-Renewal of Embryonic Stem Cells.” Stem Cells (Dayton, Ohio) 33, no. 3 (March 2015): 713–25. https://doi.org/10.1002/stem.1893.
Mendoza, Michelle C., E. Emrah Er, and John Blenis. “The Ras-ERK and PI3K-mTOR Pathways: Cross-Talk and Compensation.” Trends in Biochemical Sciences 36, no. 6 (June 1, 2011): 320–28. https://doi.org/10.1016/j.tibs.2011.03.006.