Vorwort
Hier wird eine kritische Hypothese aufgestellt.
Diese Hypothese ist nicht dazu gedacht, zu gefallen. Sie erhebt keinerlei Anspruch auf Vollständigkeit und wird in zwei Teilen - aufgrund ihrer Dringlichkeit - publiziert werden, bevor sie in ein gesamtes Werk überführt wird.
Die Teilung in 2 Teilaspekte folgt einer intrinsischen Logik. Bereits in den pharmakokinetischen Aspekten, soll bewiesen werden, dass eine Technologie zum Scheitern verurteilt ist – die ihre Ursprünge in den 1960ern genoss – wenn ihre eigene Natur einem kontrollierbaren Einsatz widerspricht.
Dieser erste Teil ist kein Einstieg. Er ist eine Prüfung der pharmakokinetischen Realität.
Jeder, der den kommenden zweiten Teil kritisieren will, ist gesamtheitlich - aus einer prüfbaren Perspektive - verpflichtet, diesen Teil gelesen und verstanden zu haben.
Zur Stilistik:
Anders als gewohnt – wird meine Kritik nüchtern und möglichst ohne persönliche Wertung formuliert.
Die Fußnoten zu den erwähnten Studien finden sich am Satzende. Ich hätte sie lieber jeweils direkt hinter die Autoren gesetzt, aber dann gibt es Probleme mit Maschinenübersetzung. Und es macht den Lesefluss unmöglich. Die Autotranslation sollte funktionieren, wenn auch mit einigen Limitationen: Es ist einfach nicht möglich, präzise Deutsch zu Englisch in wissenschaftlichen Begriffen zu arbeiten. Deshalb kann es an einigen Stellen minimal holpern. Beispielsweise wird Kapitel als “Chapter” übersetzt, obwohl in diesem Fall “section”/Sektion korrekter im Englischen wäre, was wiederum in der deutschen Sprache unüblich wäre.
LNPs und modRNA – Eine unvorhersehbare Kombination
Abstrakt
Das Trägersystem Lipid Nanoparticles (LNPs) ist stark bioaktiv. LNPs sind eine Komplexformulierung aus unterschiedlichen lipidischen Nanopartikeln (NPs). Dabei darf die Formulierung nicht mit den Eigenschaften der einzelnen Lipid-Komponenten verwechselt werden. Im Falle der Impfstoffe BNT162b2 (Pfizer) und mRNA-1273 (Moderna) handelt es sich um eine Formulierung aus synthetischen Phospholipiden, Cholesterolen, PEGylierten Lipiden und ionisierbaren Lipiden (tertiäre Amine mit verzweigtem, ungesättigtem Fettsäureschwanz).1 2
Es gibt zahlreiche unbekannte Mechanismen, die zwar teilweise beobachtet und charakterisiert wurden, jedoch bislang weder systemisch verstanden sind, noch im Kontext komplexer biologischer Systeme ausreichend untersucht wurden. Zu den Unbekannten zählen Mechanismen wie die Lipid-Protein-Corona-Interaktionen der LNPs, mögliche oxidative Verunreinigungen, Mizellenbildung der Trägersysteme, kovalente Bindungen und LNP-Addukte, die Biodistribution, Biodegradation, Transfektionsprozesse, endosomale und/oder lysosomale Effekte, u.v.m. Einige dieser werden in dieser kritischen Hypothesenprüfung diskutiert.
1. Einleitung
Ziel dieser Hypothesenschrift ist es, bislang ungelöste Fragen zur weltweit in Milliarden Dosen eingesetzten LNP-modRNA-Impftechnologie zu beleuchten. Die Arbeit gliedert sich in drei Hauptbereiche – LNPs, modRNA und empirische Beobachtungen. Der Fokus dieser Arbeit mag auf den ersten Blick einseitig wirken.
Dies ist jedoch eine bewusste methodische Entscheidung: Epidemiologische Studien mit nur schwer prüfbaren Sterblichkeitsraten (IFR), Studien – die sich auf relative Risikoreduktion (RRR) berufen –, modellbasierte Infektionskurven und Studien mit Surrogatparametern (die beispielsweise Antikörpertiter als Endpunkte werten) werden ausgeklammert. Bei Modellierungen und epidemiologischen Daten sind die Auswertungen oftmals nicht zuverlässig. Sie haben mehrere strukturelle Schwierigkeiten, die von Melderegisterungenauigkeiten, annahmeabhängigen Modellen und externen Einflussgrößen zu systematischen Verzerrungen führen.3 4 5 6 7
Diese Überlegung basiert auf einer methodischen Prämisse a priori: In einer Hypothesenschrift können ausschließlich zelluläre in vitro- und in vivo-Modelle, Biophysik (beispielsweise Säurekonstante pKa), Transkriptom- und Proteomanalysen, RNA-Sequenzierungen, Massenspektrometrie und vergleichbare Methoden als Argumente herangezogen werden, da diese experimentell validierbar oder falsifizierbar sind.
Eine weitere Einschränkung liegt darin, dass die SARS-CoV2-Spike-Proteine (SP) und ihre Pathogenität außerhalb des Rahmens dieser Arbeit liegen. Zunächst müssen die Transfektion und der endosomale Escape (die Disruption des Endosoms) untersucht werden. Anschließend müssen die modRNA-Translation und -Verarbeitung in den Fokus rücken, auch wenn diese Vorgänge fast gleichzeitig ablaufen. Die Produktion von Spike-Proteinen kann als dritte Stufe der erzwungenen pathogenen Mechanismen betrachtet werden, die durch eine modRNA-Injektion ausgelöst werden. Das SP wurde bereits ausführlich untersucht.8 9 10 11
Die hier verfolgte Hypothese lautet:
Weder Biodistribution noch Transfektionsraten sind in vivo tatsächlich kontrollierbar.
LNPs sind durch ihre intrinsische Natur bioaktiv und verändern das gesamte Zellverhalten aufgrund tiefgreifender Eingriffe in das nichtlineare, zelltypabhängige Signaltransduktionsnetzwerk.
Bereits der Zellmembrankontakt mit LNPs löst die Signaltransduktionskaskaden rezeptorabhängig und -unabhängig in einer Art aus, die so unter physiologischen Bedingungen nicht zu beobachten ist.
Die daraus folgenden Konsequenzen sind weitreichend und unzureichend charakterisiert.
Darüber hinaus wird die modRNA weitere antagonistische und synergistische Prozesse forcieren.
In den folgenden Unterkapiteln wird zunächst eine kurze Einordnung, welche Aspekte für eine Technologie relevant sind, erörtert, da diese Technologie direkt in das zelluläre Geschehen eingreift. In den anschließenden zwei Kapiteln werden die Charakteristika der LNPs und modRNA dargestellt und die Eigenschaften untersucht, die sich aus der Literatur ableiten lassen. Im abschließenden 4. Kapitel wird eine mechanistische Plausibilitätsprüfung angestrebt. In der Diskussion wird überprüft, ob die hier aufgestellten Hypothesen zutreffen und plausibel bewiesen wurden, sowie Perspektiven diskutiert, welche langfristigen Konsequenzen zu erwarten sind. Des Weiteren wird geprüft, ob LNPs als Trägersystem eine reale Chance auf Basis derzeitig verfügbarer technischer Möglichkeiten und des grundlegenden, sich ständig erweiternden systembiologischen Verständnisses haben.
1.1 Diskrepanz zwischen Theorie und experimenteller Beobachtung
Wie in dem systematischen Review zur Historie von Cullis et al. und dem von Tenchov et al. zu den prinzipiellen, theoretischen Wirkmechanismen gezeigt, sind LNP keineswegs neu.12 13 Es wird bereits seit über 60 Jahren an lipiden Trägersystemen mit unterschiedlichsten Formulierungen geforscht. Es handelt sich um Partikel im Nanometerbereich, die entweder in festem (beispielsweise metallische NPs) oder im lipiden Zustand (LNPs) vorliegen. Sie sollen einen Wirkstoff oder ein Gen an ihrer Oberfläche binden oder absorbieren bzw. – im Falle der lipiden Partikel – dieses einkapseln. Die zugrundeliegende Annahme der Lipidpartikelträger ist, dass die Liposome und LNP (welche auf der Technologie der Liposome fußen) das gewünschte genetische Material oder einen Wirkstoff einkapseln. Anschließend schleusen sie es durch Fusion mit der Membran – als biokompatibel durch ihre lipide Natur getarnt – und/oder über Rezeptor-vermittelte Bindungen ein.
Die größten Unterschiede zwischen Liposomen und LNPs liegen in der Komplexität und Größe. Theoretisch soll dies eine zusätzliche Stabilität gewährleisten, die Bioverfügbarkeit steigern, die Biodistribution kontrollieren und die Transfektionseffizienz sowie Translationsausbeute verbessern. Während Liposome eine einfache Doppelschicht zur Einkapselung aufweisen, nutzen LNPs eine wesentlich komplexere Struktur und Mechanik.
Die meisten der Wirkmechanismen wie Lipid-Zellmembranfusion, Rezeptorbindung und endosomaler/lysosomaler Escape, gelten sowohl für unterschiedlich geladenen Liposome als auch für die meisten LNP-Formulierungen. Der Grad der Interaktionen und Biodistribution hängt jedoch stärker von den jeweiligen Formulierungen ab, als es bei den Liposomen der Fall wäre. In der Praxis – sofern belastbare Biodistributionsstudien verfügbar sind – stoßen diese theoretischen Annahmen an verschiedenen Hürden, die im Folgenden diskutiert werden sollen.14 Eine in vivo-Rückverfolgung gestaltet sich insbesondere aufgrund der inhärenten Eigenschaften der LNPs als schwierig und technisch nur bedingt umsetzbar.
Für eine realistische Risikobewertung der Transfektion sollte zunächst die Lipidchemie der Zellmembran in den Mittelpunkt gerückt werden. Zellmembranen interagieren mit Zytokinen, Ionen, Proteinen, LPS und vielen weiteren molekularen Stoffen. Die Prozesse dieser Interaktionen, die Aufnahme von Stoffen, sowie die daraus resultierenden Mechanismen, sind komplex und nicht ausreichend charakterisiert. Auch die Zell-Zell-Interaktionen sind noch nicht vollständig verstanden. Dies liegt an der unterschiedlichen Zusammensetzung des Lipidmosaiks der Zellmembran, die von Zelle zu Zelle und Zelltyp zu Zelltyp variiert.15 Die Membran ist untrennbar mit dem intrazellulären Signaltransduktionsnetzwerk verflochten. Diese werden durch endozytotische Prozesse und Rezeptorkonformationsänderungen angestoßen und reguliert. Die Summe dieser Prozesse wird im Folgenden als “Membran-Chemie” zusammengefasst.16 17
Bereits hier ist nach jüngsten Erkenntnissen eine hochkomplexe Interaktionsebene erreicht.
Dimerisierungsnetzwerke – wie beispielsweise für die Mitogen Activated Protein Kinase (MAPk) charakterisiert18 19 – sind kompetitiv auf die kontextabhängige Intensität des Eingangssignals reaktiv.20 Die Schwellenwerte der Eingangssignale sind nichtlineare und können schon bei kleinsten Veränderungen sensitiv und feedback- und feedforward Loops und Signalreaktionen bewirken, wie Thiemicke et al. berichten.21 Diese neuesten Erkenntnisse stellen auch die aktuell vorherrschende Lehrmeinung infrage, wonach von einer rein transienten und moderaten Natur der Signaltransduktion ausgegangen wird und diese transient-moderate Aktivierungen immer den gewünschten Effekt erzielen würden.22 23 24 2526 27 28 Dementsprechend werden LNPs zwingend die Membran-Chemie beeinflussen. LNPs müssen demnach einen direkten Einfluss auf alle nachgeschalteten Prozesse, die von der Zellmembran abhängig sind, haben. Da die Signaltransduktionsnetzwerke als Masterregulatoren29 30 31 für alle intrazellulären Prozesse verstanden sind, muss somit die Zellmembran als zwingender Ausgangspunkt für deren Regulation berücksichtigt werden.32 33 Doch genau hier fehlt es in der Breite und Tiefe. Es gibt kaum Untersuchungen, wie LNPs sich auf die Signaltransduktion auswirken und kaum bis keine in vivo Langzeitdaten.
1.2 praktische Limitationen
In der Theorie erscheinen Komplexe wie Mizellenbildung, endosomaler Escape und endosomale Disruption noch als sauber getrennte Unterpunkte.34 35 Es ist jedoch experimentell teilweise nur schwer bis nicht trennbar zu verfolgen. In Säugetierzellen herrschen permanente Fließgleichgewichte und Dynamiken, die auf ständigen äußeren Input, intrazellulär reagieren, ihn interpretieren und anschließend verarbeiten müssen.36 37 38 39 Säugetierzellen – als selbstorganisierende Einheiten, die oft in Verbänden verknüpft sind40 – sind auf reziproke Input-Output-Wechselwirkungen angewiesen.41 42 Diese Perspektive wurde und wird – insbesondere in der LNP/mRNA-Forschung – häufig vernachlässigt.
Folglich müssen – wie bereits in der Einleitung angesprochen – zuallererst die Fragen nach der Biodistribution, Biodegradation, den transfizierten Zielzellen und der Transfektionsrate (Häufigkeit) geklärt werden. Denn erst auf dieser Grundlage sind – durch die zelltypspezifische, kontextabhängige Natur von Signaltransduktionskaskaden – präzise Veränderungen auf transkriptomischer, proteomischer, genomischer und epigenomischer Ebene nachvollziehbar. Dadurch können potenzielle Risiken und dauerhafte systemische Schäden erst identifiziert werden.
2. Pharmakokinetische Aspekte
In diesem Kapitel werden grundsätzliche Begrifflichkeiten sowie Konzepte und bestehende Schwächen der LNP-Forschung diskutiert.
Der Begriff Transfektion muss deutlich von der Genexpression unterschieden werden.43 44 Die ionisierbaren Lipide zirkulieren mit dem Blutfluss für Tage bis Wochen, wie von Couture-Senécal et al. in einem zellfreien Modell gezeigt wurde.45 Die Autoren stellten für ALC-0315 eine Halbwertszeit von 6 - 8 Tagen fest. Dies entspricht – theoretisch berechnet – der zugrunde gelegten pharmakokinetischen Formel folgend
einem Zeitraum von 30.5 bis 40.5 Tagen für einen 97 %igen Abbau. Die applizierte Dosis betrug 1 mg/kg bei “Winstar-Han-Ratten” (Korrektur: Wistar-Han-Ratten). Direkte Vergleiche zu SM-102 sind problematisch, da hier 0.2 mg/kg in Sprague-Dawley-Ratten verabreicht wurden. Weder Tiermodell noch Dosis lassen sich direkt gegenüberstellen. LNPs verteilen sich in nahezu allen Geweben – wie Lunge, Leber, Milz, Knochenmark, Gehirn – und akkumulieren vorwiegend in der Leber.46 47 48 49 50 51 52 53 54 55 56
2.1 Biodistribution - Unterschiede zwischen Distribution und Transfektion
Die präzise Bestimmung der Biodistribution bleibt eine nicht ausreichend gelöste Herausforderung. Wie bereits von Hawthorne et al. (Hawthorne, G, (November 2019): 1993–2001 [Fußnote 14]) erwähnt, sollten die kationischen bzw. ionisierbaren Lipide – als Kernstück der LNPs – wegen ihrer nicht endogenen Natur als Marker für das gesamte LNP dienen. Dies gilt also auch für die ionisierbaren Lipide von BNT162b2 (Pfizer/BioNTech) und mRNA-1273 (Moderna). Die Autoren betonen, dass es “herausfordernd” ist, intakte LNPs aus in vivo entnommenen Gewebeproben zu detektieren, und dass die verwendeten Biomarker zur Verfolgung der modRNA keine zuverlässigen Indikatoren für die Gesamtverteilung der LNPs darstellen. Denn sie zeigen nur das genetische Material beim endosomalen Escape, nicht die Gesamtverteilung der LNPs.
Diese Problematik betrifft viele der Studien zu modRNA-LNP-Technologien. Trotz des Vorschlags von Hawthorne et al. , Flüssigchromatographie-Massenspektrometrie (LC-MS) zu verwenden, um eine valide Aussage über die Verteilung und Biodegradation treffen zu können, werden zumeist fluoreszenzbasierte Reporter für die Verfolgung der Genexpression genutzt. Oft werden die Expressionsprofile der modRNA mit der Transfektionsrate und Biodistribution fälschlicherweise gleichgesetzt. Ein Beispiel für eine solch vereinfachte Modellierung liefern Guéguen et al. in vivo. (Guéguen, C., 2024 [Fußnote 55]) Sie untersuchten jedoch für ihre Lipidformulierung nur die Lunge, Milz und Leber. Wie schon kritisiert, setzen sie Transfektion mit Genexpression gleich. Es stellt sich in ihrer Studie heraus, dass die Expression in allen untersuchten Organen, trotz der Intention, gezielt die Lunge zu transfizieren, beobachtet wurde. Daraus lässt sich indirekt auch eine unvorhersehbare Verteilung der LNPs ableiten – jedoch ohne eine präzise Quantifikation, da nicht jedes einzelne LNP unbedingt eine Zelle transfiziert. Zudem stellt sich die Frage, ob LNPs, die länger als einige Stunden in einem biologischen System zirkulieren, die Transfektionsfähigkeit verlieren würden oder weitere Transfektionsevents auch noch später geschehen könnten.
Abbildung übernommen aus: Guéguen et al., European Journal of Pharmaceutics and Biopharmaceutics, Bd. 195, 2024, Art.-Nr. 114077. Lizenz: CC BY 4.0.
Aus dieser Tatsache folgt außerdem, dass auch die Transfektionsrate nicht präzise steuerbar ist. Egal, welche Formulierung verwendet wird, kommt es dadurch zu Off-Target-Effekten. Bemerkenswert ist dabei auch das im Supplementary zu findende Zytokinprofil, das ein hochinflammatorisches und recht chaotisches (nicht kanonisches) Bild abliefert.
Auch Ci et al. demonstrierten die randomisierte Biodistribution und Transfektion. (Ci, L., 2023 [Fußnote 54]) Ihre Biodistributionsstudie ist eine der wenigen, bei der die Methodik eine robuste, nach Stand der jetzigen technischen Möglichkeiten, Differenzierung aufweist. Die Autoren nutzten in vivo eine Nanoradioaktivmarkierung, um klar zwischen Genexpression und LNP-Verteilung differenzieren zu können. Aus dieser umfassenden und methodisch anspruchsvollen Arbeit ergibt sich für das verwendete ionisierbare Lipid ein klares Bild:
Abbildung übernommen aus Biodistribution of Lipid 5, mRNA, and Its Translated Protein Following Intravenous Administration of mRNA-Encapsulated Lipid Nanoparticles in RatsCi, Lei et al., Drug Metabolism and Disposition, Volume 51, Issue 7, 813 - 823, Lizenz: CC BY 4.0
Die Autoren wiesen sowohl LNP-Konzentrationen als auch Genexpression in Skelettmuskel, Plasma, Herz, Magen, Jejunum, Leber, Nieren, Hoden, Gebärmutter, Thymus, Milz, Knochenmark, Auge, Gehirn und Lunge nach. Dies ist deckungsgleich mit einem Dokument, welches im Rahmen einer Informationsfreiheitsanfrage (FOI) von der Australischen Arzeneimittelbehörde veröffentlicht wurde. (TGA foi-2389-06.pdf, [Fußnote 46]) Die in diesem Fall genutzte “modRNA-Luciferase” zeigte eine ähnliche Verteilung und Konzentration, und anschließende Protein-Translation. Entgegen der Behauptung handelt es sich nicht um die Biodistribution der LNPs, sondern ausschließlich um die mRNA-Expression. Das Luciferase-Assay war selbst nach 9 Tagen noch aktiv detektierbar, da der ‚Total Flux‘ Schwellenwert mit 10⁵ relativ hoch angesetzt war.
Abbildung übernommen aus FOI; Nonclinical Evaluation of BNT162b2 [mRNA] COVID-19 vaccine (COMIRNATY) Submission No. PM-2020-05461-1-2; TGA foi-2389-06.pdf, [Fußnote 46]; Submission No: PM-2020-05461-1-2; (Unter Berufung auf Zitatrecht (§ 51 UrhG))
Reiser et al. machten eine ähnliche Beobachtung, auch wenn ihre Arbeit nicht direkt darauf abzielte57: Sie wollten ein nichtlineares mathematisches Modell zur Verfolgung von Bioluminiszenzen auf Single Cell-Basis entwickeln und überprüfen. Dass dies ebenfalls für die Transfektion gelten muss, lässt sich durch die methodische Schwäche – nämlich der Gleichsetzung von Transfektion mit Genexpression – ableiten. Bemerkenswert ist, dass die Transfektionsrate von der Protein-Dichte im Blut und deren Konzentration direkt abhängig zu sein scheint. Erwähnenswert in diesem Kontext ist, dass die Autoren einen Prozess beobachteten, welcher erst in den letzten Jahren – mit Einführung der modRNA-COVID-19-Impfstoffe – wieder ins Forschungsbewusstsein rückte. Dies wird im folgenden Abschnitt detaillierter diskutiert.
2.2 Nichtlineare Transfektion unter den Aspekten der Protein-Corona und mRNA-Formulierung als “pay-load” sowie der LNP-Größe
Bis hierhin lässt sich festhalten, dass – entgegen dem konventionellen Verständnis – die Begriffe “Transfektion” und “Genexpression” im Laufe der Forschungsarbeiten zunehmend unklarer und nicht einheitlich definiert wurden. Entsprechend bleibt auch das Verständnis der Biodistribution limitiert.
Cedervall et al. gehörten zu den ersten, die einen neuen Mechanismus bei NPs systematisch charakterisierten58: Die Protein-Corona-Interaktion beschreibt den Prozess, wie sich durch Van-der-Waals-Kräfte, hydrophobe Interaktionen, elektrostatische Wechselwirkungen und weitere biochemische sowie biophysikalische Prozesse aus einer homogenen LNP-Formulierung ein heterogener LNP-Pool entwickelt. Dieser LNP-Pool wird durch den initialen Kontakt mit Proteinen und Molekülen im Blutplasma strukturell umgeprägt und funktional verändert. Somit bleibt der Inhalt der LNPs stabil, jedoch bildet die Oberfläche eine individuelle, vom Proteinmilieu geprägte Struktur in vivo. Weitere Arbeiten, die diese Beobachtungen auch für lipide NP bestätigen, lieferten Caracciolo et al. und Palchetti et al.59 60
Diese Veröffentlichungen hinterfragen zusammengenommen die gesamte Idee einer homogenen LNP-Formulierung und einer kontrollierbaren Biodistribution und Transfektionsrate. Auch Foroozandeh & Aziz fassten diesen Mechanismus in einer Übersichtsarbeit über Nanomaterialien zusammen.61 In einem weiteren Review von 2021 charakterisieren Onischenko et al. die Relevanz der Protein-Corona und beschreiben die Auswirkungen, welche diese auf die Anwendung von LNPs nimmt, detailliert zusammen62: Die Autoren kommen zu dem Schluss, dass mehrere Proteine, die mit der Liposomoberfläche wechselwirksam sind, zusammen biologische Effekte auslösen, wie Destabilisierung und Internalisierung des Liposoms.
Dementsprechend verändert sich die gesamte Oberflächenstruktur und die Opsonisierung wird für Phagozyten – wie Makrophagen und Dendritische Zellen – erleichtert. Einer der spannendsten Punkte, der in diesem Review betont wird, ist, dass in einer Arbeit von Sebastiani et al. gezeigt wurde, dass – wenn die LNPs mit der Protein-Corona von Apolipoprotein E (ApoE) binden – das gesamte Biodistributionsmuster der ursprünglich erdachten Formulierung durch die Strukturänderungen umgestaltet wird.63 Die ApoE Bindung, als bedeutender Faktor für Biodistribution und die zelluläre Aufnahme und Translationseffizienz wurde auch von van Straten et al. in vitro bestätigt.64 Weitere Arbeiten betonen ebenfalls die Bedeutung der Proteincorona für die Biodistribution, die Transfektionseffizienz und die Translationsausbeute.65 66 67 68 69 Die Bindung an diese Moleküle wird vorrangig durch das Zetapotenzial (ζ) bestimmt.70
Daraus folgt die Frage, ob diese Mechanismen überhaupt angemessen berücksichtigt und ausreichend adressiert wurden und ob es in vivo erprobte Lösungen für diese Probleme gab. Wie bereits unter Kapitel 2.1 diskutiert, scheint dies nicht der Fall gewesen zu sein.
Unter dem Aspekt, dass die LNPs in einer Massenimpfkampagne zum Einsatz kamen, ohne ausreichende Charakterisierung und Sicherheitsüberprüfungen, ist es dringend geboten, eine Charakterisierung unter den bis hierher diskutierten und weiteren – in den folgenden Abschnitten diskutierten – Aspekten vorzunehmen.
Es existieren bislang nur sehr wenige Informationen zu den LNP-Formulierungen von BNT162b2 (BioNtech/Pfizer) und mRNA-1273 (Moderna). Eine Arbeit, die erste Aufschlüsse über die Gesamtformulierung liefert, ist die von Schoenmaker et al., die das Verhältnis in % für BNT162b2 und mRNA-1273 wie folgt angaben71 (siehe auch 72 73):
BNT162b2
((4-hydroxybutyl)azanediyl)bis(hexane-1,6-diyl)bis(2-hexyldecanoate) (ALC-0315 [Acuitas]) [46.3] : Distearoyl-sn-glycero-3-phosphocholin (DSPC) [9.4] : Cholesterol [42.7] : 2-[(polyethylene glycol)-2000]-N,N-ditetradecylacetamid (ALC-0159) [1.6] Moderna
Heptadecan-9-yl 8-((2-hydroxyethyl)(8-(nonyloxy)-8-oxooctyl)amino)octanoat (SM-102, Moderna) [50.0] : 1,2-Distearoyl-sn-glycero-3-phosphocholin (DSPC) [10.0] : Cholesterol [38.5] : 1,2-dimyristoyl-rac-glycero-3-methoxypolyethylenglykol-2000 (PEG2000-DMG) [1.5] Einen weiteren Hinweis zur generellen Struktur des zentralen LNP-Bestandteils – den kationischen, ionisierbaren Lipiden (tertiäre Amine) – liefern Petersen et al., indem sie feststellen, dass der verzweigte, ungesättigte Fettsäureschwanz der kationischen Lipide eine Rolle für die gesteigerte Endosomdisruption spielt.([Petersen, D., 2024 Fußnote 2]) Dabei ist die kegelförmige Geometrie ein entscheidender Faktor.
An dieser Stelle sollte auch die bisherige Definition der Protein-Corona, wie zuvor beschrieben, kritisch überprüft werden. Die Aussage “Somit bleibt der Inhalt der LNPs stabil,…” bedarf womöglich einer Korrektur oder zumindest einer differenzierteren Betrachtung. Bleiben beim verkapselten genetischen Material – hier im Fall von BNT162b2 und mRNA-1273 – wirklich die ursprünglichen Eigenschaften erhalten?74 Diese Frage ist insbesondere im Kontext der sekundären und tertiären Struktur der modRNA relevant.
Brader et al. schreiben:75
“Cryo-electron microscopy images identify the mRNA location, revealing that mRNA may exist within solvent-filled cavities or may be substantially lipid associated.”,womit sie indirekt zeigen, dass es derzeit keine zuverlässige Technik gibt, den Inhalt der LNPs tatsächlich zu charakterisieren und eher von einer “Blackbox” ausgegangen werden sollte.
Gleichwohl forscht Moderna intensiv daran, kovalente Bindungen und RNA-LNP-Adducte zu minimieren, wie es aus dem Patent “US 11,524,023 B2”76 ersichtlich wird. Packer et al. berichteten im Jahr 2021 erstmals von diesem beobachteten Prozess.77 Dies deckt sich auch mit Hashiba et al., die ebenfalls Guanosin-Lipid-Konjugate in Adductform für piperidinbasierte, ionisierbare LNPs nachweisen. 78
Lamoot et al. verweisen im Jahr 2020 auf das Problem von kovalenten Oberflächenbindungen und betonen, dass NPs mit der richtigen Formulierung dauerhaft an die Zelloberfläche binden können.79 Diese Bindungen entstehen durch zufällige Konjugation und zeichnen sich durch eine langsame Reaktionskinetik in der Glykokalyx aus. Dies ist in einer Linie mit den Beobachtungen von Bussin et al.
Daraus ergibt sich die Frage, wie dieser Mechanismus, der eine langanhaltende Persistenz für die LNPs bedeuten würde, in vivo tatsächlich ausgeschlossen werden kann?
Ein weiterer Aspekt betrifft aldehydische Verunreinigungen – bedingt durch die thermodynamische Instabilität der LNPs – sowie oxidative Verunreinigungen, aufgrund der hochkomplexen Aufbereitung.80 81
Zählt man all die hier diskutierten Größen zusammen und berücksichtigt diese, ergibt sich ein klares und plausibles Bild: Hermosilla et al. zeigen, dass auch die Partikelgröße variiert.82 Wenn bereits die Prozessierung und eine Verabreichung in einen lebenden Organismus so vielfältige Störfaktoren aufweisen, ist es folgerichtig, dass weder die Biodistribution noch die Transfektion linear verlaufen können und nicht vorhersagbare, von in vivo-Modell zu in vivo-Modell variierende, Abweichungen in den gemessenen Werten entstehen. Es erscheint plausibel, dass eine nichtlineare Distributionsroute und Transfektionsrate – abhängig von der Formulierung der LNPs und den jeweiligen Lipid-Komponenten – die logische Konsequenz ist. Zusätzlich zu diesen Faktoren ist auch die modRNA-Payload ein entscheidender Aspekt (Anzahl der Stränge, Struktur der modRNA und weitere Faktoren). Wie Liao et al. beispielsweise feststellen, entscheidet die randomisiert verpackte Anzahl der modRNA-Stränge mit über das LNP-Verhalten.83 84 85 86
2.3 Welche “Zielzellen”?
Die nächste und abschließende Frage zum Thema Biodistribution ist die nach den Zellen (in den offiziellen Dokumenten der FDA und EMA als “Hostzellen” oder “Zielzellen” bezeichnet), die die modRNA translatieren sollen:
”The nucleoside-modified messenger RNA in the vaccine is formulated in lipid nanoparticles, which enable delivery of the RNA into host cells to allow expression of the SARS-CoV-2 S antigen.” (TGA foi-2389-06.pdf, [Fußnote 46])
“Encapsulation into LNPs enables transfection of the RNA into host cells after intramuscular (IM) injection.”FDA87
“The mRNA delivery system is based on the principle and observation that cells in vivo can take up mRNA, translate it, and express viral protein antigen(s) in the desired conformation.” EMA [spikevax-previously-covid-19-vaccine-moderna-epar-public-assessment-report_en.pdf Fußnote 49]
“In mRNA-Impfstoffen ist die genetische Information für ein Erreger-Protein in Form von Ribonukleinsäure (ribonucleic acid, RNA) enthalten. RNA erfüllt in Körperzellen verschiedene Funktionen, unter anderem die der sogenannten Boten-RNA (messenger RNA, mRNA)”PEI88
“The mRNA-1273 vaccine is delivered via intramuscular injection, and mRNA is subsequently delivered into cells, primarily antigen presenting cells at the injection site and draining lymph nodes.”FDA89
Wie unter Abschnitt 2.1 erläutert, ist die Biodistribution der LNPs nach intravenöser Gabe systemweit. Eine Retention im Muskel nach der modRNA-LNP-Gabe ist unwahrscheinlich. Die LNPs erreichen – abhängig von ihren physikochemischen Eigenschaften – zahlreiche Organe und Gewebe. Es können Blut-Organ-Schranken durch Transzytose umgangen oder durch direkte Penetration überwunden werden. Zhang et al. liefern dazu eine umfassende Übersichtsarbeit, die eine Vielzahl an Zielzellen – wie Epithelzellen, basale Zellen, Endothelzellen u.a. – benennt und die Mechanismen erklärt, wie diese in Abhängigkeit von der LNP-Formulierung besonders wahrscheinlich transfiziert werden.90
Dazu muss auch geklärt werden, welche Rezeptoren und Liganden die wahrscheinlichsten Interaktionspartner der LNPs neben der rezeptorunabhängigen “endozytoseähnlichen” Aufnahme sind.
In ihrem ersten Review 2008 halten Lonez et al. fest, dass nicht abschließend geklärt wurde, ob eine rezeptor-abhängige oder rezeptor-unabhängige, “endozytoseähnliche” Aufnahme der Liposome in die Zellen vermittelt wird.91
In ihrem zweiten Review zu Liposomen 2012 stellten Lonez et al. fest92:
“Endocytosis or endocytosis-like mechanisms are the main pathways of internalization for lipoplexes (cationic liposomes/DNA complexes), even if the exact nature of endocytic vesicles involved is still a matter of debate [16–18].”Es werden diverse Rezeptoren – wie TLR4, CD14, CD11b und CD11c – diskutiert und festgestellt, dass je nach Formulierung die Liposome an diese binden können und die Zellen während des “endozytoseähnlichen” Prozesses ins Endosom als Rezeptor-LNP-Komplex internalisieren.
Es ist wahrscheinlich, dass sowohl rezeptorvermittelte und rezeptorunabhängige Zellaufnahmen, abhängig von der Proteincorona, bei ein und derselben LNP-Formulierung gleichzeitig stattfinden.
Zu einer ähnlichen Schlussfolgerung kamen Akinc et al., indem sie zeigen, dass “iLNPs” sowohl durch endogenes ApoE-vermitteltes Targeting als auch durch exogenes Ligand-gebundenes Targeting, etwa via GalNAc an den ASGPR-Rezeptor, effektiv an Zielzellen gebunden und internalisiert werden können.93 Wie unter 1.2 und 2.2 gezeigt, wird der Prozess, ob ein rezeptor- oder rezeptorunabhängig vermittelter “endozytoseähnlicher” Prozess stattfindet, stark von der Proteincorona und dem Zustand der Zelle, auf die das LNP stößt, abhängen.
Paunovska et al. zeigen, dass LNPs an Apolipoprotein E sowie Low-density Lipoprotein Rezeptoren binden können94, während Chaudhary et al. den TLR4 und CD1d für die Internalisation identifizieren.95 Eine weitere Übersichtsarbeit dazu liefern Baimanov et al.96
Daraus lässt sich ableiten, dass es unwahrscheinlich ist, dass nur ein spezifischer Zelltyp transfiziert wird.
Massaro et al. transfizierten erfolgreich Alveolarepithelzellen und Lungenfibroblasten in vivo.97 Es bleibt jedoch unklar ob Off-Target Gewebe und Zellen mittransfiziert wurden. Ein vergleichbares Ergebnis zeigen Liu et al. für Alveolarepithelzellen Typ II in vivo. 98 Khan et al. demonstrieren darüber hinaus, dass sie Endothelzellen der Lunge mit ihrer LNP-Formulierung transfizieren können.99
Während Cheng et al. berichten, dass ihre “SORT” LNP-Formulierung 39% der Epithelzellen, 66% der Endothelzellen und 21% der Immunzellen in der Lunge transfizieren konnte.100 Darüber hinaus wurden 93% der Hepatozyten in der Leber und 12 % B-Zellen, 10 % T-Zellen und 20 % Makrophagen in der Milz transfiziert. Ihre Analyse konzentriert sich auf die Lunge, Milz und Leber bei ihren Versuchen. Sie berichten, keine hepatischen oder renalen Toxizitätseffekte beobachtet zu haben. Es bleibt jedoch fraglich, ob diese nach zwei Tagen – dem Zeitpunkt an dem die drei Versuchstiere für jede Gruppe getötet wurden – bereits sichtbar gewesen wären. Hier ist ebenfalls die Limitation der reinen RNA-Expressionsverfolgung zu beachten, wie bereits ausführlich unter 2 und 2.1 diskutiert. Eine interessante Schlussfolgerung, zu der die Autoren kommen, ist, dass ihr Helferlipid “DOTAP” die Route maßgeblich beeinflusste. Wobei diese Schlussfolgerung aufgrund der Limitation, nicht die LNP-Formulierung als solche verfolgt zu haben, eher spekulativ erscheint. Es bleibt also fraglich, ob sich die Biodistribution, die Transfektionsrate oder die Escaperate – und damit die Genexpression – veränderte.
Diese Beobachtung deckt sich mit den Experimenten von Johnson et al.101 Darüber hinaus stellten die Autoren fest, dass Kupfferzellen transfiziert werden. Die Autoren berichten in ihrem Abstract:
“In contrast, the surface of 3A5-SC14 LNPs was enriched in albumin but depleted in ApoE, which likely led to Kupffer cell delivery and detargeting of hepatocytes.” Dies steht in einer Linie mit den bereits erwähnten Beobachtungen von Akinc et al. (Akinc, A., 2010 [Fußnote 93]) Auch Hosseini-Kharat et al. erklären, dass Kupfferzellen ein bevorzugtes Ziel von LNPs sind. (Hosseini-Kharat, M., 2025 [Fußnote 56])
Sakurai et al. zeigen, dass ihre LNP-Formulierung Hirnkapillar-Endothelzellen (hCMEC/D3) transfizieren kann.102
Es gibt viele weitere Beispiele, die zeigen, dass es nicht nur einen Zelltyp und ein Organ gibt, die exklusiv transfiziert werden. Es kommt immer zur Transfektion von diversen Zelltypen – je nach Formulierung – in unterschiedlichen Verhältnissen. Weitere Beispiele, die erwähnenswert sind: Herz- und Skelettmuskulatur, knochenmarksmaturierte dendritische Zellen und Makrophagen, hepatische ITO-Zellen (HSCs) und viele weitere Zelltypen und Gewebe.103 104 105 106 107 108 109
Wie gezeigt, lässt sich nur schwer bis unmöglich eine Aussage darüber treffen, welche Zellen tatsächlich in welcher Häufigkeit mit den LNPs von BNT162b2 und mRNA-1273 transfiziert wurden. [Vergleich TGA Fußnote 46].
Abbildung übernommen aus FOI; Nonclinical Evaluation of BNT162b2 [mRNA] COVID-19 vaccine (COMIRNATY) Submission No. PM-2020-05461-1-2; TGA foi-2389-06.pdf, [Fußnote 46]; Submission No: PM-2020-05461-1-2; (Unter Berufung auf Zitatrecht (§ 51 UrhG))
Aufgrund des Expressionsprofils der TGA-Daten erscheint es plausibel anzunehmen, dass Immunzellen, HSCs, Kupfferzellen, Thekazellen, Stromazellen, Fibroblasten, Endothel- und Epithelzellen (im gesamten Verteilungsweg über alle Gewebe hinweg, beispielsweise retinale Pigmentepithelzellen), Leydig-Zellen, Sertoli-Zellen, peritubuläre Myofibroblasten usw. transfiziert wurden bzw. direkten LNP-Kontakt hatten – im Sinne, dass die Zellen hätten transfiziert werden können. (Siehe auch Han, X., 2021 [Fußnote 108]) Die eigentliche Herausforderung liegt nicht darin, die Frage zu klären, ob eine Transfektion erfolgte, sondern in welchem Verhältnis und wie systembiologische Bedingungen dies beeinflussen. Es lässt sich nach aktuellem Kenntnisstand festhalten, dass vorrangig der pKa-Wert, nicht das ζ-Potenzial, entscheidend für die Biodistribution und Zellaufnahme ist, während das ζ-Potenzial primär die Proteincorona und damit die Eintrittswahrscheinlichkeit bestimmt.110 (Straten, D., 2024 [Fußnote 64])
2.4 Endosomaler Escape - die Herausforderung aus klassisch-pharmakokinetischer Perspektive
Es erscheint schwierig, ausgehend von Degradation und Wirkweise aus klassisch-pharmakokinetischer Perspektive beurteilen zu können, wie LNPs abgebaut werden, da sie nicht im traditionellen Sinn durch Organaufnahme metabolisiert werden, wenn sie eine Zelle transfizieren. Dementsprechend reichen die Prozesse des endosomalen Escape und der Degradation über die gesamte Bandbreite der Pharmakodynamik.111112 Das klassische ADME-Modell greift nicht bei liposomalen und Nanopartikelträgersystemen. Ein Beispiel zur reinen pharmakokinetischen Perspektive, wie man dieses Problem überwinden kann, liefern Müller et al. auf dem Prinzip einer Single Cell-Analyse.113 Die wichtigsten Beobachtungen der Autoren sind, dass eine heterogene Aufnahme in den Zellen stattfand und ein zeitlich verzögerter endosomaler Escape, der unter den einzelnen Zellen variierte. Die Proteinausbeute richtete sich dabei nach der Menge der erfolgreich ins Zytosol entlassenen RNA.
Die Transfektion geschieht – wie in Kapitel 2 und Kapitel 2.2 diskutiert – rezeptorabhängig und/oder rezeptorunabhängig. Ermilova und Swenson kommen zu einer äußerst bemerkenswerten Beobachtung: Sie halten fest, dass sowohl ALC-0315 als auch SM-102 die Zellmembran penetrieren. Bei ihrer Kalkulation zur freien Energie schreiben sie: 114
“It was discovered that ALC-0315 binds strongest to the membrane, while DLin-MC3-DMA is not able to reside in the bilayer center. The ability to penetrate the membrane POPC by SM-102 and ALC-0315 can be related to their saturation, comparing to DLin-MC3-DMA.”Dies deckt sich auch mit den Beobachtungen von Pilkington et al.115
Der endosomale Escape – oder auch als endosomale Disruption bezeichnet – beschreibt den Prozess, bei dem die Payload nach der Transfektion (siehe Kapitel 2) freigesetzt wird. Während der endosomalen Maturierung, teilweise auch lysosomalen Übergangsphase, initiiert der zunehmende Säuregradient eine Protonierung der LNPs. Dies ist durch die unterschiedlichen pKa-Verhältnisse, die bei den ionisierbaren Lipiden zwischen 6.09 für ALC-0315 – 6.6 für SM-102 variieren, bedingt. Durch die Protonierung bilden sich Lamellen in dem Endosom bzw. Lysosom, wodurch die Payload in das Zytosol freigesetzt wird(alternativ als Proton-Schwamm-Effekt erwähnt).116 117
Doch auch hier finden sich Limitationen und nicht ausreichend charakterisierte und verstandene Hürden. Liu et al. merken an:118
“Our findings show that mRNA formulated with the three ionisable lipids in the currently approved LNP products result in significantly different degrees of protein expression in Jurkat, HEK293 and DC1940 cell lines. However, our assay revealed that, unexpectedly, neither the inherent endosomal escape ability of the LNPs, nor the amount of nucleic acid delivered into the cytosol reliably predicted the level of protein expression.”Chatterjee et al. berichten darüber hinaus (Chatterjee, S., 2024 [Fußnote 116])
“Despite the developments in the LNP field, a commonly overlooked aspect regards the very limited release of the nucleic acid payloads in the cytoplasm (17–19).” Es bleibt also unklar, wie viele LNPs transfizieren, und wie viele davon überhaupt einen endosomalen Escape initiieren. Und bereits hier zeigt sich die angesprochene Schwierigkeit Pharmakokinetik und Pharmakodynamik zu trennen, da eigentlich bereits hier die Folgen mitdiskutiert werden müssten. Diese werden jedoch in den späteren Kapiteln noch adressiert.
Eine weitere offene Frage ist, was mit den ionisierbaren Lipiden geschieht, die ihre Ladung auch nach dem LNP-Zerfall im Endosom behalten werden. Werden diese zusammen mit der Payload in das Zytosol entlassen? Einen der wenigen Hinweise auf diese bisher kaum adressierte Möglichkeit liefern Jörgensen et al.: (Jörgensen, A. M., 2023 [Fußnote 35])
“The optimum pKa-value of the polar head provides an uncharged, bioinert surface at neutral pH on the one hand, and allows protonation and subsequent interaction with the inner leaflet of the endosomal membrane on the other hand, when the pH is decreasing.[83] However, as often exclusively amines, predominantly tertiary ones, were screened, this optimum may only apply to ionizable cationic lipids, which achieve endosomal escape via interaction with acidic lipids on the inner leaflet of endosomes and subsequent disruption of the endosomal membrane.[83, 86]” Damit räumen sie ausdrücklich die Möglichkeit ein, dass die protonierten ionisierbaren Lipide in das Zytosol entweichen können, da sie direkt die endosomale Membran disruptieren. Doch daraus ergeben sich weitere Fragen, wie etwa die Interaktion dieser ionisierten Lipide mit der Payload, mit Mitochondrien, Signalmolekülen und vielen weiteren kaum untersuchten Möglichkeiten, die sehr viel Raum für hypothetische Überlegungen lassen.
2.5 Biodegradation
Hier wird der letzte Aspekt dieser Arbeit aus der pharmakokinetischen Perspektive behandelt: die Biodegradation.
Zu ALC-0315 und SM-102 existieren keine klinischen Daten, bezüglich ihrer Verweildauer und Aktivitätsdauer. Jörgensen et al. stellen weiter fest, dass die tertiären Amine der aktuell in Anwendung genutzten Covid-19-modRNA-Injektionen nicht wie übliche biokompatible “Bausteine” abgebaut werden. Nach der Fettsäureschwanzspaltung werden sie zwar per Definition als “biologisch abbaubar” betrachtet, aber im eigentlichen Sinne verbessert sich ihre Abbaubarkeit nicht, da ionisierbare kationische Lipidbausteine residieren.
Die Schlussfolgerungen von Jörgensen et al. stimmen durch indirekte Rückschlüsse mit aktuellen Ergebnissen von Couture-Senécal et al.119 überein. Die Autoren verfolgten die hydrolytische Aktivität und Deesterisierung ihrer LNP-Formulierung, ohne die Aminkopfgruppe zu berücksichtigen. Folglich beobachteten sie ausschließlich die Aktivität bis zur Fettsäureschwanztrennung. Bei ihrer LNP-Formulierung gab es keine messbaren Ausscheidungen im Urin und Kot. Daraus ergeben sich Fragen zum Verbleib der markierten Fettsäureschwänze, wie sie abgebaut werden, ob sie noch nach der Trennung messbar sind und wo die ionisierbaren Amine verbleiben. Des Weiteren wird nicht ersichtlich, wo die LNPs akkumulierten. Wenn 97 – 99 % der δO3-Formulierung weder ausgeschieden noch aktiv verfolgbar sind, ist dies kein Beweis für eine Degradation. Zudem zeigt sich in “Figure 3”, dass die δO3-Konzentration in Muskelgewebe und in der Milz zum Messpunktende ansteigt und es werden keine unteren Nachweisgrenzen angegeben. Weitere Messpunkte werden trotz der Konzentrationszunahme nicht berichtet.
Han et al. schreiben zur Biodegradation 2021 (Han, X. 2021 [Fußnote 108]):
“However, top-performing ionizable lipids synthesized using this method usually contain stable backbones and multiple tertiary amines, leading to slow degradation and potential toxicity.”Zusammenfassend lässt sich als Kapitelabschluss ein weiterer bemerkenswerter blinder Fleck feststellen: Die Biodegradationsstudien gehen sämtlich von einer Serum gesteuerten Bereinigung der LNPs aus. Aufgrund der amphiphilen Natur von LNPs erscheint dieser Eliminationsweg jedoch nicht plausibel.120 Folglich muss die terminale Halbwertszeit ermittelt werden.121 Daraus ergibt sich, dass ein einfaches Kompartimentmodell nicht ausreicht und mindestens ein zweifaches, wenn nicht sogar ein multiples Modell zur Ermittlung benötigt wird. Es existiert nur ein offizielles Dokument der Epar zu BNT162b2 in dem die terminale Halbwertszeit angegeben wurde und diese betrug für ALC-0159 und ALC-0315 72.7h. Dies deckt sich mit der in Kapitel 2 eingangs zitierten Formel und Couture-Senécal et al.(Couture-Senécal, J.,October 29, 2024 [Fußnote 45]) Deshalb ist es wahrscheinlich, dass ein einfaches lineares Ein-Kompartiment-Modell unzureichend ist und sowohl ein nichtlineares als auch ein Mehr-Kompartiment-Modell erforderlich sind, um das “metabolische Schicksal”122 123 124 und die Elimination des ionisierbaren Lipids zu bestimmen.
Eine abschließende und bislang nicht adressierte Frage lautet, wie viele LNP-Komplexe überhaupt in einer Injektion enthalten sind und wie sichergestellt wurde, dass jede Gabe ungefähr die gleiche Anzahl beinhaltet, da dies nur sehr schwer umsetzbar ist. Die Annahme “30 µg modRNA = definierte Wirkung” unter Berücksichtigung der physikochemischen Eigenschaften der LNPs ist implausibel.
3. Die modRNA – Charakteristika und nicht adressierte Fragen
Dieses Kapitel behandelt systematisch Modifizierungen der modRNA sowie potenziell funktionale Implikationen.
Für die SC2-Protein-kodierende RNA wurden in der BNT162b2- und mRNA-1273-RNA-Sequenz diverse Codonmodifizierungen und ein vollständiger Austausch des Uracils durch N1-Methylpseudouridin (m1Ψ) vorgenommen.125 126 127 128 129 In diesem Kapitel wird die Natur dieser Modifikationen anhand der Beispiele des 3'-Endes der untranslatierten Region (3'-UTR) und der Uracilsubstitutionen untersucht. Des Weiteren werden nicht ausreichend charakterisierte Aspekte für spätere Kapitel adressiert. Dieses Kapitel folgt dem Aufbau von Kapitel 2 und dient vorrangig einer charakterisierenden, nicht bewertenden Beweisführung.
3.1 Die 3'-UTR
Die 3'-UTR ist im Kontext der Translation ein besonders wichtiger Sequenzabschnitt: Sie ist funktional – unter anderem durch microRNA (miRNA)-Regulierung – bei der Translation essentiell.130 131 132 133
Ihre Bedeutung für eukaryotische Translations-Initiationsfaktoren (eIF) muss ebenfalls betont werden134135 136: Es stellte sich beispielsweise heraus, dass eIF1 in Kombination mit eIF4G1 darüber entscheidet, ob ein 48s-Ribosom die 5'-UTR präzise erkennt oder durch Fehler in einen aberranten Leserahmen gerät (ein Phänomen, dass oft als “ribosomaler Frame Shift” betitelt wird). (Weiss, B., 2024, [Fußnote 134] Dies führt zu ribosomalen Resten, die nicht mehr aktiv sind und teilweise an der 3'-UTR gebunden bleiben. Aus dieser Beobachtung lässt sich ableiten, dass die 3'-UTR nicht nur ein passives Ende der RNA vor dem Poly-Adenyl (A)-Schwanz darstellt, sondern direkte strukturbeeinflussende Effekte auf die gesamte mRNA hat. Zudem ist die Frage berechtigt, ob es Rückkopplungseffekte gibt und diese somit direkten Einfluss auf die eIF-Regulierung haben können.
Einen weiteren Aspekt zu eIFs und ihrer direkten Verknüpfung zur 3'-UTR liefern Mestre-Fos et al. in vitro indem sie nachweisen, dass eIF3 direkt an die 3'-UTR-Termini bei hochfrequentierten RNA-Translatierungen bindet und deren Translationsaktivität – nicht aber Translationseffizienz – regulieren.137 Da dieser Mechanismus eine neue Entdeckung darstellt, ist die Frage nach systemischer Relevanz noch nicht abschließend geklärt.
Eine weitere bemerkenswerte Beobachtung lässt sich aus dem aktuellsten Investorenbericht von BioNTech festhalten. Darin heißt es: 138
“Our platform patent filings relevant to our COVID-19 vaccines, collectively, the “BNT162b2 Platform Filings”, include certain mRNA Structure Filings relating to features for increasing translation efficiency and/or stability of mRNA constructs (e.g., certain 3’ UTR structures containing a specific sequence element, interrupted polyA tails, and certain 5’ cap/cap proximal sequence combinations), including filings that are jointly owned by BioNTech and TRON; also relevant are certain mRNA Manufacturing Filings.” Dieses “spezielle 3'-UTR-Element” soll im Weiteren näher betrachtet werden.
Granados-Riveron et al. charakterisieren 2021 die Codonoptimierungen in einer umfangreichen Übersichtsarbeit.(Granados-Riveron, J., 2021 [Fußnote 124]) BNT162b2 verwendet zwei Stopcodons, während mRNA1273 drei nutzt. Dabei stellen sie für die 3'-UTR der mRNA-1273 von Moderna fest, dass diese eine 110 Nukleotide (nt) lange Insertion des menschlichen Betaglobin-Gens (HBA1) zwischen dem letzten Stopcodon und dem Poly-A-Schwanz enthält. BioNTech/Pfizer nutzte ein 136-nt-langes AES/TLE5-Gensegment 6 nt downstream vom zweiten Stopcodon. Darüber hinaus wurde eine 12S rRNA (mtRNR1) mit einer Länge von 139 nt downstream vom ersten Stopcodon insertiert. Daraus ergibt sich bereits eine nicht beantwortbare Frage, da es keine offizielle Dokumentation zur Begründung gibt, warum hinter Stopcodons zusätzliche Fragmente menschlicher Gensequenzen eingefügt wurden. Auch die erkennbare Intention, die miRNA-Bindungsstellen zu minimieren und damit die Translationsausbeute zu optimieren, liefert dafür keine plausible Erklärung.
Ein interessanter Punkt, der sich aus der Arbeit von Ugur Sahin als Projektleiter im Jahr 2019 ableiten lässt139: Wenn, wie in der Arbeit von Orlandini Von Niessen et al. beschrieben, bewusst nach 3'-UTR-Sequenzen geforscht wurde, um die Translationsausbeute zu steigern und die miRNA-Bindungsstellen zu minimieren, bedeutet dies, dass zellinterne miRNAs für die Translation genutzt werden. Welche miRNAs jedoch, bleibt eine offene Frage und wird von Zelltyp und Zelle zu Zelle (nach ihrer Kontextaktivität) variieren.140
Abbildung übernommen aus ModernaTX, Inc. mRNA-1273 3.2.S.1.2 Structure; assets.publishing.service.gov.uk/media/659e8576e96df5000df843c2/FOI_22-1004_PDF_attachment___2_.pdf (Unter Berufung auf Zitatrecht (§ 51 UrhG))
Im Kontext der 3'-UTR ist auch der Poly-A-Schwanz bemerkenswert, da er eine nicht ausreichend charakterisierte Modifikation beinhaltet.
Die mRNA-1273 beinhaltet einen gemischten, 100 nt langen Poly-A-Schwanz, welcher von einem m1ΨCm1ΨAG-Motiv terminiert wird.141 Seine funktionelle Bedeutung ist unzureichend charakterisiert. Krawczyk et al. hypothesieren, dass der Poly-A-Schwanz re-adenyliert werden könnte.142 Allerdings weist die Studie, die dies diskutiert, mehrere methodische Schwächen auf, welche eine tiefere Interpretation erschweren. Die Autoren gehen davon aus, dass das m1ΨCm1ΨAG kein Teil des Poly-A-Schwanzes ist und schreiben:
“Finally, the presence of a TCTAG (mΨCmΨAG in the vaccine mRNA) was revealed through rapid amplification of cDNA 3′ end (3′-RACE) Illumina sequencing. The mΨCmΨAG pentamer is likely to represent the residue of restriction enzyme cleavage of the DNA template.” Abgesehen von der inkorrekten Nutzung von m1Ψ für N1-Methylpseudouridin (in der Arbeit “mΨ” bezeichnet) deckt sich dies nicht mit den in einem FOI publizierten Daten von Moderna und stellt somit eine grundlegende Fehlannahme dar.
Abbildung übernommen aus Krawczyk, Paweł S., Michał Mazur, Wiktoria Orzeł, Olga Gewartowska, Sebastian Jeleń, Wiktor Antczak, Karolina Kasztelan, et al. “Re-Adenylation by TENT5A Enhances Efficacy of SARS-CoV-2 mRNA Vaccines.” Nature 641, no. 8064 (May 2025): 984–92. https://doi.org/10.1038/s41586-025-08842-1. (Figure 3d); Lizenz: CC BY 4.0.
Darüber hinaus enthält die Studie statistische Signifikanzangaben, deren Plausibilität nicht ganz nachvollziehbar scheint. So wird etwa ein Likelihood-Ratio-Omnibus-Test (LRT) für eine Genanalyse zur Bewertung über verschiedene Zeiträume genutzt. Die Grafiken entsprechen teilweise nicht den Schlussfolgerungen der Autoren. Es ergeben sich offensichtliche Diskrepanzen zwischen den angegebenen p-Werten und der Heatmap. Die statistischen Ausreißer scheinen zu einer Überinterpretation der p-Werte zu führen. Eine mögliche Erklärung könnte beispielsweise die FDR-Korrektur (nach Benjamini-Hochberg) sein, da sie dazu führen kann, dass nur das am stärksten exprimierte Gen eine klare Signifikanzbewertung erreicht, weil zu strenge Cutoffs gesetzt werden.143 Es ist darüber hinaus möglich, dass weitere Störfaktoren nicht berücksichtigt werden – wie zu wenige biologische Replikate oder eine zu breite Streuung innerhalb der Gruppen.
Die Tent5-Familie katalysiert ausschließlich A bei der Poly-A-Schwanz-Adenylierung, während gemischte A/G-Motive eher mit der TENT4-Familie assoziiert werden.144 145 146 Zusammengenommen ergibt sich eine biologische Implausibilität der diskutierten Schlussfolgerungen.
Abbildung übernommen aus Nance, Kellie D., and Jordan L. Meier. “Modifications in an Emergency: The Role of N1-Methylpseudouridine in COVID-19 Vaccines.” ACS Central Science 7, no. 5 (May 26, 2021): 748–56. https://doi.org/10.1021/acscentsci.1c00197.; Lizenz: CC BY 4.0.
Bei BNT162b2 ist GACm1Ψ hinter 30 A downstream integriert und danach folgen weitere 70 A. (Nance, K.D., 2021 [Fußnote 126])
3.2 N1-Methylpseudouridin (m1Ψ) - Einleitung
Von Uridin zu Pseudouridin:
Die Uracil-Substitutionen sind einer der grundlegendsten und signifikantesten Unterschiede zur viralen SC2-Spike-mRNA. (Nance, K.D., 2021 [Fußnote 126]; 147; Morais, P, 2021 [Fußnote 127]) Im Folgenden werden ausschließlich die funktionalen Fragen der mRNA-Struktur, möglichen Temperaturunterschieden, der ursprünglichen Intention für die Substitution und der Genauigkeit der Translation diskutiert.
Pseudouridine Ψ sindeine Uridin-Modifikation, die zwar in menschlichen Zellen beobachtet wurden, aber eher unübliche Modifikationen darstellen. Uridin wiederum entsteht durch glykosidische Bindungen zwischen Uracil und Zucker. Diese Modifikation wurde 1951 erstmals von Waldo E. Cohn & Elliot Volkin entdeckt.148 Stand 2021 wurden 172 spezifische Nukleoside identifiziert.149
ψ ist eine posttranskriptionale und masseneutrale Modifikation. Dadurch ist eine Identifikation mittels MS nur schwer umsetzbar, ohne es vorher in ein Derivat umzuwandeln.150
Abbildung übernommen aus Mengel-Jorgensen, J. “Detection of Pseudouridine and Other Modifications in tRNA by Cyanoethylation and MALDI Mass Spectrometry.” Nucleic Acids Research 30, no. 23 (December 1, 2002): 135e–1135. https://doi.org/10.1093/nar/gnf135,; Lizenz: CC BY 4.0.
Das Basengerüst von Pseudouridin ist Pseudouracil, ein Pyrimidinring, der jedoch eine bemerkenswert abweichende Zuckerbindung aufweist. Die N1-glykosidische Bindung entfällt, und es bildet sich eine glykosidische C5-C-Bindung. Daraus folgt, dass die methylierte Variante (m1Ψ ) von Ψ das Methyl an der N1-Position gebunden hat.
Die methylierte Modifikation wurde erstmals in Streptomyces platensis identifiziert.151 m1Ψ wurde in tRNA und rRNA identifiziert.152 153 154 Pang et al. lieferten dazu eine Pionierarbeit.155
Die chemischen Eigenschaften von m1Ψ und Ψ sind vergleichbar. Die strukturellen und biochemischen Unterschiede beeinflussen jedoch direkt die Translation und Translationstemperatur.156 157158
Abbildung übernommen aus [Fußnote 156] Chen, Tien-Hao, Vladimir Potapov, Nan Dai, Jennifer L. Ong, and Bijoyita Roy. “N1-Methyl-Pseudouridine Is Incorporated with Higher Fidelity than Pseudouridine in Synthetic RNAs.” Scientific Reports 12, no. 1 (July 29, 2022): 13017. https://doi.org/10.1038/s41598-022-17249-1.; Lizenz: CC BY 4.0.
3.3 - Warum m1Ψ?
3.3.1 Die Stabilität
Zunächst lag der Fokus auf der Stabilität der mRNA. Ohne Modifikationen wird diese zu schnell abgebaut.159 Die Projekte von K. Karikó, D. Weissman, U. Sahin und Ö. Türeci fokussieren sich kontinuierlich auf Toll-Like-Rezeptoren (TLR)-Aktivitäten und RNA-Aktivität im Kontext des Immunsystems sowie auf Modifizierungen von mRNA zur therapeutischen Anwendung.160 161 162 163 164 165 166 Man kann argumentieren, dass Karikó, Weißmann, Sahin und Türeci ihre eigentliche Inspiration hauptsächlich aus real beobachteten Prozessen erhielten und ein bereits bekanntes Konzept auf die Idee der Modifikation von mRNA adaptierten.167 168 Es ist wichtig festzuhalten, dass nach aktuellem Kenntnisstand keine natürlich vorkommende humane mRNA-Spezies m1Ψ beinhaltet und lediglich RNA-Spezies mit Ψ an exakt lokalisierten Bindungsstellen der RNA identifiziert wurden.169 Es wurde bewusst eine Modifikation gewählt, die in der Natur ausschließlich in transkriptionalen und ribosomalen RNA-Molekülen anzufinden wäre. Eines der Ziele war es, die mRNA-Stabilität zu gewährleisten. Spenkuch et al.170 schreiben dazu in ihrer differenzierten Übersichtsarbeit:
“Indeed 2 recent studies reported various inducible Ψs in yeast mRNA.24,25 Further investigation identified the enzyme Pus7p to be mainly responsible for heat shock induced pseudouridylation in yeast: A change in localization of the enzyme from mainly nuclear to also cytosolic seems to allow mRNA pseudouridylation that presumably contributes to mRNA stability.25” Mauger et al. fassen darüber hinaus zusammen, dass die strukturellen Eigenschaften der mRNA die Halbwertszeit direkt betreffen. Sie stellen fest, dass insbesondere die sekundäre RNA-Struktur als dominanter Faktor wirkt.171
3.3.2 War die Halbwertszeit der einzige Grund?
Zur Beantwortung dieser Frage, sind zunächst folgende Arbeiten von Karikó et al. zu diskutieren:172
“It was reported previously that HEK293 cells transfected with TLR4 produced IL-8 in response to specific ligand (22). Therefore, we analyzed supernatants from TLR3-expressing HEK293 cells for IL-8 production after stimulation with RNA. Interestingly, in the absence of Lipofectin, these cells exhibited a high base-line secretion of IL-8 (typically 100–400 pg/ml) with a significant increase in IL-8 after poly(I)·poly(C) or mRNA stimulation.” In dem zitierten Paper wurde pcDNA3.0 genutzt.
Ihre Beobachtung, die Karikó et al. hier zeigen , dass auch die Stimulation von TLR3 eine Interleukin-8-Stimulation (IL-8) bewirkt, ist bahnbrechend. Die Erkenntnisse aufgeschlüsselt lauten wie folgt:173 174 175 176 177 178 179
Nicht nur der TLR4 – welcher gerade bei LPS die entscheidende Rolle über das Zellschicksal spielt – initiiert die Sezernierung von IL-8. IL-8 muss als hochinflammatorisches Zytokin, als eine Art “letzte Bastion”, betrachtet werden.
Neben dem TLR4 der Zellmembran sind auch die anderen zytosolischen TLRs beteiligt(TLR3, 7, 8, 9)
Es war bekannt, dass IL-8 eines der höchstinflammatorischen Zytokine ist, das nur bei wirklich lebensbedrohlichen oder chronisch entzündlichen Situationen stark hochreguliert wird.
Es ist bereits während dieser Experimente bekannt, dass LPS über den TLR4 IL-8 auslöst.
Die eigentliche neue Erkenntnis von Karikó et al. ist, dass TLR3 im Anschluss diese Prozesse überwacht und die IL-8-Antwort erneut aktiviert, um eine inflammatorische Reaktion zu verstärken. Daraus ergeben sich wichtige Schlussfolgerungen: Wenn genetisches Material in eine Zelle eingeschleust werden soll, ohne sie direkt durch Interleukine als gefährlich zu markieren, müssen sowohl TLR4 als auch TLR3 überwunden werden.
Abbildung übernommen aus Karikó, Katalin, Michael Buckstein, Houping Ni, and Drew Weissman. “Suppression of RNA Recognition by Toll-like Receptors: The Impact of Nucleoside Modification and the Evolutionary Origin of RNA.” Immunity 23, no. 2 (August 2005): 165–75. https://doi.org/10.1016/j.immuni.2005.06.008.; Lizenz: CC BY 4.0.
Dies führt zu der wohl bemerkenswertesten Arbeit von Karikó et al. und Ψ. (Karikó, K., 2005 [Fußnote 160]) Die Autoren schreiben, dass bereits eine Modifikation von 14–29 Nukleosiden mit m6A, Ψ oder m5C ausreichte, um eine halbierte TNF-α-Exprimierung in den dendritischen Zelltypen, die sie in vitro transfiziert hatten, zu erzwingen. Wie aus “Figure 2” erkennbar wird, supprimiert Ψ als einzige Modifikation alle zytosolischen TLRs (3, 7, 8, 9) gleichermaßen unter sämtlichen Bedingungen. Dies erklärt die rationale Entscheidung der Autoren, diese Modifikation weiterzuverfolgen.
Warum die Wahl letztlich auf m1Ψ fiel, lässt sich aus Arbeiten, wie jener von Svitkin et al. erklären:180 Die Autoren zeigen dass m1Ψ die Translationseffizienz eIF2α-abhängig und -unabhängig steigert. Sie demonstrieren, dass die immun/eIF2α-Phosphorylierungsabhängigkeit durch diese Modifikation abgeschaltet wurde und die Translationsrate drastisch stieg. Dies geschieht durch eine gesteigerte Ribosomenpause und -dichte.
Daraus ergibt sich bereits - nüchtern betrachtet - eine paradoxe Situation: Auf der einen Seite müssen Immuncheckpunkte überwunden werden, um eine effiziente Translationsausbeute zu erzielen. Auf der anderen Seite soll aber zugleich eine Immunantwort auf die pathogene modRNA erfolgen, welche jedoch durch die Modifikationen die Ursache der Immunsupprimierung ist.
3.4 Nicht ausreichend adressierte Probleme mit m1Ψ
3.4.1 ribsomales Frameshift und Nonsenscodons
Ψ ist schon seit Langem dafür bekannt, zu ribosomalen Frameshifts zu führen. Dabei kommt es durch Leseflussverzögerung beim Ablesen der RNA zu ribosomalen Stalling, was wiederum dazu führen kann, dass das Ribosom um ein – X Nukleotide verrutscht. Wie unter 3.2 ausgeführt, wurde es vorrangig in rRNA-Spezies und tRNA-Spezies nachgewiesen. Liao et al. halten dazu 2006 fest181:
“The absence of modified nucleoside pseudouridine (Ψ) at position 38–40 in tRNATrpCCA could also be a reason for less-efficient binding of this tRNA to the E-site. It is suggested that deficiency of modified nucleosides may change tRNA structure, resulting in different ribosome:tRNA interactions (29).”Eine wirklich bemerkenswerte Beobachtung machten Karijolich et al. 2011, indem sie eine Umwandlung von Nonsenscodons, welche die Translation terminieren, in Sensecodons nach Pseudouridylierung beobachteten182:
“Here, we demonstrate that pseudouridylation (conversion of uridine into pseudouridine (Ψ), ref. 4) of nonsense codons suppresses translation termination both in vitro and in vivo.”Auch Atkins et al. halten im Jahr 2016 fest183:
“Significant frameshifting generally occurs at a discrete site, though when occurring at multiple repeat codons as in the decoding of huntingtin, multiple sites are involved (376). Site-specific mRNA pseudouridylation occurs and is regulated by environmental conditions such as nutrient status (386) with ΨAG decoding being distinctive (387).”Daraus lässt sich ableiten, dass diese Frameshifts und Sense/Nonsensecodon-Rekodierung gleich oder vergleichbar für m1Ψ auftreten werden. Chen et al. vermerken dazu (Chen, Tien-Hao 2022 [Fußnote 156]):
”The uridine-depleted RNA sequences transcribed with T7 RNAP with equal molar unmodified rNTPs had a total combined error rate 6.0 ± 1.1 × 10−5 errors/base, while the error rates of ψ- and m1ψ-incorporating transcripts were observed to be 1.9 ± 0.4 × 10−4 and 9.9 ± 3.0 × 10−5 errors/base, respectively (Fig. 4a and Supplementary Table 7).”Eine kaum beachtete Arbeit, die bereits im Jahr 2021 die Möglichkeit der Frameshifts diskutierte, wurde von Xuhua Xia verfasst. Darin schrieb der Autor:184
“However, UGAU is known to cause a +1 frameshifting. It is reasonable to infer that ΨGAΨ may be the same. I have mentioned before that mammalian AZ1 gene with a stop codon context UGAU is prone to polyamine-induced +1 frameshifting [85]. Such a +1 frameshifting defeats the purpose of having multiple stop codons as a fail-safe mechanism.”Die Möglichkeit des Frameshiftings wurde experimentell von Mulroney et al. bestätigt.185 Wobei folgende Feststellung der Autoren verfrüht erscheint:
“Although there is no evidence that frameshifted products in humans generated from BNT162b2 vaccination are associated with adverse outcomes, for future use of mRNA technology it is important that mRNA sequence design is modified to reduce ribosome frameshifting events, as this may limit its future use for applications that require higher doses or more frequent dosing, such as the in vivo production of hormones.”Diese Schlussfolgerung erscheint unzulässig. Es existieren keine Langzeituntersuchungen. Das “off-target Protein” wurde nicht mittels Röntgenkristallografie (X-Ray), Kryo-EM oder vergleichbarer Methodik dreidimensional kartographiert und charakterisiert. Dies liegt daran, dass die Möglichkeit nicht in Betracht gezogen und untersucht wurde, obwohl die Hinweise, wie ausgeführt, existierten. In diesem Kontext ist anzumerken, dass – soweit bekannt – auch keine 3d-Strukturanalyse zum eigentlichen “Impfspike” gemacht wurde. Es kann also nicht von “keine Evidenz existiert” geredet werden, wenn die Untersuchungen nicht gemacht wurden. Denn die Abwesenheit von Evidenz ist weder mit Evidenzexistenz noch mit Nichtexistenz gleichzusetzen.
Die Möglichkeit des “Codon Readthrough” ist eng mit dem Thema Frameshifting verwandt. Interessanterweise zitierten auch Cernickis et al.186 in ihrer Diskussion über diese Möglichkeit die bereits erwähnte Arbeit von Karijolich et al. und halten fest:
”Although its context-dependent regulation is only beginning to be understood, Ψ is involved in multiple steps of mRNA processing (Figure 1) and has been mostly studied in protein translation. Karijolich and Yu demonstrated that Ψ-modified termination codons (ΨAA, ΨAG, and ΨGA) suppressed translation termination in yeast, leading to stop codon readthrough in premature termination codon (PTC)-bearing mRNAs [24].”Und auch hier stellt die Abwesenheit der Untersuchungen keine Evidenz dafür dar, dass dies nicht geschieht. Es sollte eher vom Gegenteil ausgegangen werden, bis diese Möglichkeit endgültig falsifiziert wurde. Adachi et al. zeigen im Jahr 2020, dass Codon Readthroughs in S. cerevisiae kontext- und sequenzunabhängig geschehen, wenn Stopcodons Ψ beinhalten.187
Einen weiteren, fundamentalen Hinweis auf die Möglichkeit eines Codon Readthroughs liefert eine kaum beachtete Arbeit von Veenstra et al., die bis heute nur als Preprint existiert. Sie stellen fest, dass das 141 kDa Impfspike von mRNA-1273 um 39 kDa varierte und eine Masse 180 kDa aufwies. 188
Abbildung übernommen aus Veenstra, Timothy D., Brad Pauley, Elisha Injeti, and Rocco J. Rotello. “In Vitro Characterization of SARS-CoV-2 Protein Translated from the Moderna mRNA-1273 Vaccine.” Allergy and Immunology, March 2, 2022. https://doi.org/10.1101/2022.03.01.22271618.; Lizenz: CC BY 4.0.
Jedoch erscheint die Schlussfolgerung der Autoren, dass sich 39 kDa mit Glykosylierung erklären lassen implausibel. Eine Überlegung dazu: 39 kDa würde eine Glykosylierungseffizienz von 60 % bedeuten. Das ist – alleine aus zytoplasmatischer Logik abgeleitet – unwahrscheinlich. Wie schon vorher diskutiert sind Ψ-substituierte Stopcodons anfälliger für Codon Readthroughs, und Modernas Sequenz nutzte gleich 3 Stopcodons in der Reihenfolge “UGA UAA UAG” oder um noch präziser zu werden “m1ΨGA m1ΨAA m1ΨAG”.(FOI_22-1004_PDF_attachment___2_.pdf [Fußnote 140]) Bemerkenswert ist auch, dass ein weiteres Band bei um die 250 kDa gezeigt wurde, welches die Autoren nicht weiter diskutieren.
Abbildung übernommen aus ModernaTX, Inc. mRNA-1273 3.2.S.1.2 Structure; assets.publishing.service.gov.uk/media/659e8576e96df5000df843c2/FOI_22-1004_PDF_attachment___2_.pdf (Unter Berufung auf Zitatrecht (§ 51 UrhG))
Dementsprechend sollte folgende Plausibilitätsprüfung angestellt werden: “Wenn Stop-Codons durchgelesen werden, würden c.a. 25,91 kDa ohne den Poly-A-Schwanz inbegriffen entstehen, und der Poly-A-Schwanz fügt weitere ca. 4,62 kDa hinzu. Insgesamt kommen also 30,53 kDa dazu. Die Glykosylierung könnte zwischen 4 und 13 kDa hinzufügen. 39 - 30,53 = 8,46.”*
Lin et al. merken an:189
“Moreover, when the uridine is replaced with a Ψ in a stop codon, it results in nonsense suppression at the translational level [[63]] and this Ψ-mediated read-through is independent of the sequence context [[67]].” Die Problemstellung bleibt insgesamt unbeantwortet und kontrovers diskutiert, da die Möglichkeit nie falsifiziert wurde und die biologischen, zugrundeliegenden Mechanismen plausibel und evident sind.
3.4.2 Biodegradation
Da es kaum verfügbaren Daten gibt, die sich explizit mit der Biodegradation von m1Ψ beschäftigt haben, ist dieses Kapitel vorrangig durch indirekte Hinweise und verwandte Mechanismen adressiert.
Die einzige Studie, die mit einem einzigen Nukleosid modifiziert für 21 Tage biolumineszierend in vivo m1Ψ verfolgte, ist aus dem Jahr 2015. Andries et al. beobachteten mit einer einzigen m1Ψ-Substitution, dass das Signal auch nach 21 Tagen noch, von allen Modifikationen, die sie testeten, am aktivsten war.190
Ho et al. merken dazu folgende wichtige und mit früheren Beobachtungen konsistente Feststellung an (siehe auch 3.3.2):191
“TLR7 and TLR8 also recognize RNA degradation products and require free guanosines and uridines, respectively, for maximum activation. Ψ and N1-methyl-Ψ could optimise mRNA performance by reduced immunogenicity and effective protein translation. Reduced binding to TLRs due to Ψ.”Morais et al. halten in ihrer Diskussion zu m1Ψ in der modRNA-LNP-Technologie im Jahr 2021 Folgendes fest (Morais, P, 2021 [Fußnote 127]) :
"Also, because pseudouridylation appears to be irreversible, Ψ is usually excreted from the body. Thus, this RNA modification has drawn attention as a potential biomarker for Alzheimer’s disease and certain types of cancer (Morais et al., 2021)."Stockert et al. stellen in ihrer Arbeit zu der Bedeutung von Ψ in Prostatakrebs 2019 folgendes fest:192
"Since pseudouridine cannot be salvaged and has to be generated de novo, it presumably gets excreted in body fluids [14]. Thus, it has been hypothesized that high levels of excreted modified nucleosides such as pseudouridine could serve as biomarkers of tumor growth [7]."Die vorhandene Lektüre legt nahe, dass Ψ eine posttranskriptionale Modifikation ist, die, wenn überhaupt, nur schwer enzymatisch abgebaut werden kann und normalerweise durch Exkretion aus dem biologischen System ihr “metabolisches Schicksal” erfüllt. Es gibt lediglich Hinweise auf eine gestörte Elimination, die im Folgenden nur am Rande als Plausibilitätsprüfung dienen sollen, da es um die mechanistischen Aspekte geht.
Hanna et al. wiesen mindestens 5 Tage die Persistenz von modRNA in vivo nach. Da ein zu hoch angesetztes Detektionslimit in dieser Arbeit vorliegt, fand dies hier Berücksichtigung und die Zeitverschiebung wird konservativ auf 5 Tage geschätzt.193
“In rats, up to 3 days following intramuscular administration, low vaccine mRNA levels were detected in the heart, lung, testis, and brain tissues, indicating tissue biodistribution.4”Fertig et al. zeigten in ihrer Untersuchung 15 Tage persistierende modRNA.194
“A recent ultrasensitive single-molecule assay was however able to detect the S-protein in the plasma of some mRNA-1273 COVID-19 vaccinees at 15 days following injection [12], while in another study, both mRNA and S-protein could be found in axillary LNs after 60 days [13].”Castruita et al. wiesen bis zu 28 Tage gemessene modRNA-Sequenzen nach.195
“In this paper, we describe the unexpected finding of SARS-CoV-2 vaccine mRNA sequences in plasma from 10 HCV patient samples up to 28 days after COVID-19 vaccination. These patients had recently received SARS-CoV-2 mRNA vaccinations according to the Danish roll-out vaccination plan.”Dies deckt sich mit den Ergebnissen von Krauson et al.196
“Using highly sensitive single-molecule array assays, spike protein derived from mRNA-1273 was detected in the plasma of patients up to 28 days from most recent vaccination20.”Magen et al. detektierten bis zu mindestens einem Monat.197
“However, in our patient, we observed the expression of vaccine mRNA in muscle tissues as late as one month after vaccination, which may indicate that the exogenously expressed mRNA was stable enough to persist over a long period of time.”Röltgen et al. wiesen 8 Wochen nach.198
“(…) mRNA vaccination stimulates robust GCs containing vaccine mRNA and spike antigen up to 8 weeks postvaccination in some cases.”Abschließend lässt sich hier also festhalten, dass die modRNA nicht die zu erwartenden metabolischen Phasen einer endogenen mRNA durchläuft. Viele der hier erwähnten Studien differenzierten methodisch zwischen Spikeprotein und modRNA, wodurch die Persistenz evident nachgewiesen ist.
3.4.3 miRNA
Wie bereits unter 3.1 erwähnt, werden miRNAs auch eine entscheidende Rolle bei der Translation spielen. Leider ist auch dieser Bereich nach aktuellem Wissensstand nicht ausreichend charakterisiert und eine der wenigen erwähnenswerten Arbeiten lieferten Lockhart et al. im Jahr 2018:199
“The increased silencing of GFP-4x126TS switches compared to GFP-1x126TS was seen at all levels of Ψ substitution, but it only reached statistical significance in the 100% Ψ-modified miRNA switches (p < 0.05).”Sie stellen fest, dass eine hundert prozentige Substitution durch Ψ die miRNA Regulation verschiebt und andere Bindungsregeln auslöst, was an der strukturellen Alteration der modifizierten RNA liegt (dazu im folgenden Kapitel detailliert). Die Autoren fahren fort:
”Recently, it has been reported that mRNAs containing m1Ψ modification outperformed the Ψ- and Ψ/m5C-modified mRNA platforms.20, 21 Therefore, we compared the silencing of GFP-4x126TS containing 100% m1Ψ substitutions to unmodified, 100% Ψ-modified, and 100% Ψ/m5C-modified miRNA switches in HEK293 cells. In the presence of miR-126-3p mimics, m1Ψ-modified GFP-4x126TS exhibited 31.8% relative GFP expression (p < 0.05) and was significantly different than unmodified GFP-4x126TS that showed only 6.8% GFP expression relative to vehicle controls (Figures 6A and 6B).”Aus der Arbeit lässt sich ableiten, dass der “globale miRNA-Haushalt” (die Bioverfügbarkeit der zellinternen miRNA) gestört wird, wenn miRNA-Sponging die Verfügbarkeit der gebundenen miRNA reduziert, indem es das Silencing behindert und die miRNA übermäßig lang gebunden bleibt.200 201 Das bedeutet im Detail, dass die zellinternen miRNA vermutlich zusätzlich rekrutiert werden oder ihre regulatorischen Rollen angepasst werden müssen.
3.4.4 Die sekundäre und tertiäre Struktur
Die Aspekte der sekundären und tertiären Struktur werden hier exemplarisch an zwei Beispielen skizziert, um die Komplexität und Wechselwirkungen zu veranschaulichen.
Für eine korrekte Translation einer RNA ist die sekundäre und tertiäre Struktur entscheidend.202 Eines der wohl wichtigsten Merkmale der sekundären Struktur sind G-Quadruplexe. Sie entstehen durch G-reiche Sequenzen, in denen Guanine in Anwesenheit von Kationen “G-Quartette” bilden. Diese wiederum können G-quadruplexe Strukturen durch Stapelung bilden. Eine rationale Frage, die sich daraus ableitet, lautet, wie diese Möglichkeit – bei den LNPs nach der Protonierung – und somit der kationisch, ionisierbaren Lipide im Endosom für die modRNA ausgeschlossen wurde. Wie unter 3.3.1 schon angeschnitten, zeigten Mauger et al. 2019 darüber hinaus, dass die sekundäre Struktur der mRNA direkt die Translationsausbeute beeinflusst. (Mauger,D. M., 2019 [Fußnote 169])
Die Tertiäre RNA-Struktur ist beispielsweise für die microRNA Maskierung oder Exponierung entscheidend. Wie von Gan und Gunsalus anhand von benachbarten RISC-Komplexen beschrieben, können mehr als eine miRNA gleichzeitig an räumlich nahe Bindungsstellen binden.203 Die tertiäre Struktur der mRNA entscheidet darüber, ob solche Mehrfachbindungen räumlich möglich sind. Und auch ob sie stabil bleiben.
Aus der Arbeit “mRNA structure determines modification by pseudouridine synthase 1” von Carlile et al. lässt sich plausibel ableiten, dass, wenn Pus1 und andere Enzyme strukturelle Präferenzen besitzen, umgekehrt auch die Thermodynamik und Faltung von mRNA durch Ψ verändert werden.204 Daraus wiederum ergibt sich, dass ähnliche Eigenschaften auch für m1Ψ gelten müssen.
Declaration of Competing Interest
The author declares no competing interests.Author contributions
F. Seger wrote the manuscriptAcknowledgments
This work was supported by Maria Gutschi, David Cowley, and a group of people eager to understand the technology. Special thanks to Dr. Hélène Banoun, Dr. Annelise Bocquet, David Cowley, Dr. Claire Craig, Dr. med. Johanna Deinert, Dr. Lynn Fynn, Maria Gutschi, Nicole Lorenzen, Dr. Jacobus Passer, Dr. Jens Pohl, Dr. Adriana Radler-Pohl, Pinsolle Typhaine, Dr. Richard Urso, the whistleblower “jikkyleaks”, and many others who made this work possible through their feedback and constructive discussions.
*And a very special thank you to "LeBiochimiste" for doing the maths.Wilson, Barnabas, and Kannoth Mukundan Geetha. “Lipid Nanoparticles in the Development of mRNA Vaccines for COVID-19.” Journal of Drug Delivery Science and Technology 74 (August 2022): 103553. https://doi.org/10.1016/j.jddst.2022.103553.
Petersen, Daria M. Strelkova, Ryan M. Weiss, Khalid A. Hajj, Saigopalakrishna S. Yerneni, Namit Chaudhary, Alexandra N. Newby, Mariah L. Arral, and Kathryn A. Whitehead. “Branched‐Tail Lipid Nanoparticles for Intravenous mRNA Delivery to Lung Immune, Endothelial, and Alveolar Cells in Mice.” Advanced Healthcare Materials 13, no. 22 (September 2024): 2400225. https://doi.org/10.1002/adhm.202400225.
Polack, Fernando P., Stephen J. Thomas, Nicholas Kitchin, Judith Absalon, Alejandra Gurtman, Stephen Lockhart, John L. Perez, et al. “Safety and Efficacy of the BNT162b2 mRNA Covid-19 Vaccine.” New England Journal of Medicine 383, no. 27 (December 31, 2020): 2603–15. https://doi.org/10.1056/NEJMoa2034577.
Olliaro, Piero, Els Torreele, and Michel Vaillant. “COVID-19 Vaccine Efficacy and Effectiveness—the Elephant (Not) in the Room.” The Lancet Microbe 2, no. 7 (July 2021): e279–80. https://doi.org/10.1016/S2666-5247(21)00069-0.
Donzelli, Alberto, Marco Alessandria, and Luca Orlando. “Comparison of Hospitalizations and Deaths from COVID-19 2021 versus 2020 in Italy: Surprises and Implications.” F1000Research, September 24, 2021. https://doi.org/10.12688/f1000research.73132.1.
Stang, Andreas, Johannes Robers, Birte Schonert, Karl-Heinz Jöckel, Angela Spelsberg, Ulrich Keil, and Paul Cullen. “The Performance of the SARS-CoV-2 RT-PCR Test as a Tool for Detecting SARS-CoV-2 Infection in the Population.” Journal of Infection 83, no. 2 (August 2021): 237–79. https://doi.org/10.1016/j.jinf.2021.05.022.
Buitrago-Garcia, Diana, Aziz Mert Ipekci, Leonie Heron, Hira Imeri, Lucia Araujo-Chaveron, Ingrid Arevalo-Rodriguez, Agustín Ciapponi, et al. “Occurrence and Transmission Potential of Asymptomatic and Presymptomatic SARS-CoV-2 Infections: Update of a Living Systematic Review and Meta-Analysis.” PLOS Medicine 19, no. 5 (May 26, 2022): e1003987. https://doi.org/10.1371/journal.pmed.1003987.
Trougakos, Ioannis P., Evangelos Terpos, Harry Alexopoulos, Marianna Politou, Dimitrios Paraskevis, Andreas Scorilas, Efstathios Kastritis, Evangelos Andreakos, and Meletios A. Dimopoulos. “Adverse Effects of COVID-19 mRNA Vaccines: The Spike Hypothesis.” Trends in Molecular Medicine 28, no. 7 (July 2022): 542–54. https://doi.org/10.1016/j.molmed.2022.04.007.
Parry, Peter I., Astrid Lefringhausen, Conny Turni, Christopher J. Neil, Robyn Cosford, Nicholas J. Hudson, and Julian Gillespie. “‘Spikeopathy’: COVID-19 Spike Protein Is Pathogenic, from Both Virus and Vaccine mRNA.” Biomedicines 11, no. 8 (August 17, 2023): 2287. https://doi.org/10.3390/biomedicines11082287.
Yonker, Lael M., Zoe Swank, Yannic C. Bartsch, Madeleine D. Burns, Abigail Kane, Brittany P. Boribong, Jameson P. Davis, et al. “Circulating Spike Protein Detected in Post–COVID-19 mRNA Vaccine Myocarditis.” Circulation 147, no. 11 (March 14, 2023): 867–76. https://doi.org/10.1161/CIRCULATIONAHA.122.061025.
Ota, Nakao, Masahiko Itani, Tomohiro Aoki, Aki Sakurai, Takashi Fujisawa, Yasuaki Okada, Kosumo Noda, Yoshiki Arakawa, Sadahisa Tokuda, and Rokuya Tanikawa. “Expression of SARS-CoV-2 Spike Protein in Cerebral Arteries: Implications for Hemorrhagic Stroke Post-mRNA Vaccination.” Journal of Clinical Neuroscience 136 (June 1, 2025): 111223. https://doi.org/10.1016/j.jocn.2025.111223.
Cullis, P. R., and P. L. Felgner. „The 60-Year Evolution of Lipid Nanoparticles for Nucleic Acid Delivery“. Nature Reviews. Drug Discovery 23, Nr. 9 (September 2024): 709–22. https://doi.org/10.1038/s41573-024-00977-6.
Tenchov, Rumiana, Robert Bird, Allison E. Curtze, and Qiongqiong Zhou. „Lipid Nanoparticles─From Liposomes to mRNA Vaccine Delivery, a Landscape of Research Diversity and Advancement“. ACS Nano 15, Nr. 11 (23. November 2021): 16982–15. https://doi.org/10.1021/acsnano.1c04996.
Hawthorne, Glen, Neil Henderson, Mikko Hölttä, Sima Khan, Johnny Lindqvist, and Amanda Wilson. “Overcoming Analytical Challenges to Generate Data Critical to Understanding Lipid Nanoparticle-Delivered Modified mRNA Biodistribution.” Bioanalysis 11, no. 21 (November 2019): 1993–2001. https://doi.org/10.4155/bio-2019-0138.
Nicolson, Garth L., and Gonzalo Ferreira de Mattos. “The Fluid–Mosaic Model of Cell Membranes: A Brief Introduction, Historical Features, Some General Principles, and Its Adaptation to Current Information.” Biochimica et Biophysica Acta (BBA) - Biomembranes 1865, no. 4 (April 1, 2023): 184135. https://doi.org/10.1016/j.bbamem.2023.184135.
Pelley, John W. “Membranes and Intracellular Signal Transduction.” In Elsevier’s Integrated Biochemistry, 37–46. Elsevier, 2007. https://doi.org/10.1016/B978-0-323-03410-4.50011-0.
Grecco, Hernán E., Malte Schmick, and Philippe I.H. Bastiaens. “Signaling from the Living Plasma Membrane.” Cell 144, no. 6 (March 2011): 897–909. https://doi.org/10.1016/j.cell.2011.01.029.
Cobb, M. H., and E. J. Goldsmith. „Dimerization in MAP-Kinase Signaling“. Trends in Biochemical Sciences 25, Nr. 1 (Januar 2000): 7–9. https://doi.org/10.1016/s0968-0004(99)01508-x.
Rauch, Nora, Oleksii S Rukhlenko, Walter Kolch, and Boris N Kholodenko. “MAPK Kinase Signalling Dynamics Regulate Cell Fate Decisions and Drug Resistance.” Current Opinion in Structural Biology, Multi-protein assemblies in signaling • Catalysis and regulation, 41 (December 1, 2016): 151–58. https://doi.org/10.1016/j.sbi.2016.07.019.
Parres-Gold, Jacob, Matthew Levine, Benjamin Emert, Andrew Stuart, and Michael B. Elowitz. “Contextual Computation by Competitive Protein Dimerization Networks.” Cell 188, no. 7 (April 2025): 1984-2002.e17. https://doi.org/10.1016/j.cell.2025.01.036.
Thiemicke, Alexander, and Gregor Neuert. „Rate Thresholds in Cell Signaling Have Functional and Phenotypic Consequences in Non-Linear Time-Dependent Environments“. Frontiers in Cell and Developmental Biology 11 (21. März 2023). https://doi.org/10.3389/fcell.2023.1124874.
Jiang, Yanfei, Zohreh AkhavanAghdam, Yutian Li, Brian M. Zid, and Nan Hao. “A Protein Kinase A–Regulated Network Encodes Short- and Long-Lived Cellular Memories.” Science Signaling 13, no. 632 (May 19, 2020): eaay3585. https://doi.org/10.1126/scisignal.aay3585.
Ochoa, David, David Bradley, and Pedro Beltrao. “Evolution, Dynamics and Dysregulation of Kinase Signalling.” Current Opinion in Structural Biology, Folding and binding in silico, in vitro and in cellula • Proteins: An Evolutionary Perspective, 48 (February 1, 2018): 133–40. https://doi.org/10.1016/j.sbi.2017.12.008.
Vijay, Siddharth, and Taranjit S. Gujral. “Non-Linear Deep Neural Network for Rapid and Accurate Prediction of Phenotypic Responses to Kinase Inhibitors.” iScience 23, no. 5 (May 22, 2020): 101129. https://doi.org/10.1016/j.isci.2020.101129.
Ghomlaghi, Milad, Anthony Hart, Nhan Hoang, Sungyoung Shin, and Lan K. Nguyen. “Feedback, Crosstalk and Competition: Ingredients for Emergent Non-Linear Behaviour in the PI3K/mTOR Signalling Network.” International Journal of Molecular Sciences 22, no. 13 (June 28, 2021): 6944. https://doi.org/10.3390/ijms22136944.
Landry, Benjamin D., David C. Clarke, and Michael J. Lee. “Studying Cellular Signal Transduction with OMIC Technologies.” Journal of Molecular Biology 427, no. 21 (October 23, 2015): 3416–40. https://doi.org/10.1016/j.jmb.2015.07.021.
Szischik, Candela L., Juliana Reves Szemere, Rocío Balderrama, Constanza Sánchez de la Vega, and Alejandra C. Ventura. “Transient Frequency Preference Responses in Cell Signaling Systems.” Npj Systems Biology and Applications 10, no. 1 (August 11, 2024): 1–18. https://doi.org/10.1038/s41540-024-00413-w.
Tsuruyama, Tatsuaki. „Nonlinear thermodynamics of biological signal transduction for predicting conservation of entropy production rate“. Journal of Theoretical Biology 523 (21. August 2021): 110716. https://doi.org/10.1016/j.jtbi.2021.110716.
Fossett, Nancy. “Signal Transduction Pathways, Intrinsic Regulators, and the Control of Cell Fate Choice.” Biochimica et Biophysica Acta 1830, no. 2 (February 2013): 2375–84. https://doi.org/10.1016/j.bbagen.2012.06.005.
Mukherjee, Avik, Yanqing Huang, Jens Elgeti, Seungeun Oh, Jose G. Abreu, Anjali Rebecca Neliat, Janik Schüttler, et al. “Membrane Potential as Master Regulator of Cellular Mechano-Transduction.” bioRxiv, May 13, 2024. https://doi.org/10.1101/2023.11.02.565386.
Lavoie, Hugo, Jessica Gagnon, and Marc Therrien. “ERK Signalling: A Master Regulator of Cell Behaviour, Life and Fate.” Nature Reviews. Molecular Cell Biology 21, no. 10 (October 2020): 607–32. https://doi.org/10.1038/s41580-020-0255-7.
Jastrzebska, Beata, Aleksander Debinski, Slawomir Filipek, and Krzysztof Palczewski. “Role of Membrane Integrity on G Protein-Coupled Receptors: Rhodopsin Stability and Function.” Progress in Lipid Research 50, no. 3 (July 2011): 267–77. https://doi.org/10.1016/j.plipres.2011.03.002.
Ammendolia, Dustin A., William M. Bement, and John H. Brumell. “Plasma Membrane Integrity: Implications for Health and Disease.” BMC Biology 19, no. 1 (April 13, 2021): 71. https://doi.org/10.1186/s12915-021-00972-y.
Schlich, Michele, Roberto Palomba, Gabriella Costabile, Shoshy Mizrahy, Martina Pannuzzo, Dan Peer, and Paolo Decuzzi. „Cytosolic delivery of nucleic acids: The case of ionizable lipid nanoparticles“. Bioengineering & Translational Medicine 6, Nr. 2 (20. März 2021): e10213. https://doi.org/10.1002/btm2.10213.
Jörgensen, Arne Matteo, Richard Wibel, and Andreas Bernkop‐Schnürch. „Biodegradable Cationic and Ionizable Cationic Lipids: A Roadmap for Safer Pharmaceutical Excipients“. Small 19, Nr. 17 (April 2023): 2206968. https://doi.org/10.1002/smll.202206968.
Richards, David M., and Timothy E. Saunders. “Spatiotemporal Analysis of Different Mechanisms for Interpreting Morphogen Gradients.” Biophysical Journal 108, no. 8 (April 2015): 2061–73. https://doi.org/10.1016/j.bpj.2015.03.015.
Delgoffe, Greg M., Peter J. Murray, and Dario AA Vignali. “Interpreting Mixed Signals: The Cell’s Cytokine Conundrum.” Current Opinion in Immunology 23, no. 5 (October 2011): 632–38. https://doi.org/10.1016/j.coi.2011.07.013.
Ullo, Maria F., and Lindsay B. Case. “How Cells Sense and Integrate Information from Different Sources.” WIREs Mechanisms of Disease 15, no. 4 (July 2023): e1604. https://doi.org/10.1002/wsbm.1604.
Su, Jimeng, Ying Song, Zhipeng Zhu, Xinyue Huang, Jibiao Fan, Jie Qiao, and Fengbiao Mao. “Cell–Cell Communication: New Insights and Clinical Implications.” Signal Transduction and Targeted Therapy 9, no. 1 (August 7, 2024): 1–52. https://doi.org/10.1038/s41392-024-01888-z.
Zhu, Meng, and Magdalena Zernicka-Goetz. “Principles of Self-Organization of the Mammalian Embryo.” Cell 183, no. 6 (December 2020): 1467–78. https://doi.org/10.1016/j.cell.2020.11.003.
Rosenlehner, Teresa, Stefanie Pennavaria, Batuhan Akçabozan, Shiva Jahani, Thomas J. O’Neill, Daniel Krappmann, Tobias Straub, Jan Kranich, and Reinhard Obst. “Reciprocal Regulation of mTORC1 Signaling and Ribosomal Biosynthesis Determines Cell Cycle Progression in Activated T Cells.” Science Signaling 17, no. 859 (October 22, 2024): eadi8753. https://doi.org/10.1126/scisignal.adi8753.
Xia, Yuhan, Kazuki Inoue, Ting Zheng, Yongli Qin, and Baohong Zhao. “Reciprocal Suppression between TGFβ Signaling and TNF Stimulation Finetunes the Macrophage Inflammatory Response.” FASEB Journal: Official Publication of the Federation of American Societies for Experimental Biology 38, no. 13 (July 15, 2024): e23704. https://doi.org/10.1096/fj.202302230R.
Kirschman, Jonathan L., Sushma Bhosle, Daryll Vanover, Emmeline L. Blanchard, Kristin H. Loomis, Chiara Zurla, Kathryn Murray, Blaine C. Lam, and Philip J. Santangelo. “Characterizing Exogenous mRNA Delivery, Trafficking, Cytoplasmic Release and RNA-Protein Correlations at the Level of Single Cells.” Nucleic Acids Research 45, no. 12 (July 7, 2017): e113. https://doi.org/10.1093/nar/gkx290.
Chong, Zhi Xiong, Swee Keong Yeap, and Wan Yong Ho. “Transfection Types, Methods and Strategies: A Technical Review.” PeerJ 9 (April 21, 2021): e11165. https://doi.org/10.7717/peerj.11165.
Couture-Senécal, Julien, Jagriti Natraj, and Omar F. Khan. “A Cell-Free Kinetic Analysis of Ionizable Lipid Hydrolysis.” Analytical Chemistry 96, no. 43 (October 29, 2024): 17128–34. https://doi.org/10.1021/acs.analchem.4c02399.
Naasani, Imad. „Establishing the Pharmacokinetics of Genetic Vaccines Is Essential for Maximising Their Safety and Efficacy“. Clinical Pharmacokinetics 61, Nr. 7 (1. Juli 2022): 921–27. https://doi.org/10.1007/s40262-022-01149-8.
Luo, Jie, Muge Molbay, Ying Chen, Izabela Horvath, Karoline Kadletz, Benjamin Kick, Shan Zhao, u. a. „Nanocarrier Imaging at Single-Cell Resolution across Entire Mouse Bodies with Deep Learning“. Nature Biotechnology, 14. Januar 2025, 1–14. https://doi.org/10.1038/s41587-024-02528-1.
Kent, Stephen J., Shiyao Li, Thakshila H. Amarasena, Arnold Reynaldi, Wen Shi Lee, Michael G. Leeming, David H. O’Connor, u. a. „Blood Distribution of SARS-CoV-2 Lipid Nanoparticle mRNA Vaccine in Humans“. ACS Nano 18, Nr. 39 (1. Oktober 2024): 27077–89. https://doi.org/10.1021/acsnano.4c11652.
Korzun, Tetiana, Abraham S. Moses, Parham Diba, Ariana L. Sattler, Olena R. Taratula, Gaurav Sahay, Oleh Taratula, and Daniel L. Marks. „From Bench to Bedside: Implications of Lipid Nanoparticle Carrier Reactogenicity for Advancing Nucleic Acid Therapeutics“. Pharmaceuticals 16, Nr. 8 (31. Juli 2023): 1088. https://doi.org/10.3390/ph16081088.
Ci, Lei, Marjie Hard, Hannah Zhang, Srujan Gandham, Serenus Hua, John Wickwire, Tod Wehrman, u. a. „Biodistribution of Lipid 5, mRNA, and Its Translated Protein Following Intravenous Administration of mRNA-Encapsulated Lipid Nanoparticles in Rats“. Drug Metabolism and Disposition: The Biological Fate of Chemicals 51, Nr. 7 (Juli 2023): 813–23. https://doi.org/10.1124/dmd.122.000980.
Guéguen, Claire, Thibaut Ben Chimol, Margaux Briand, Kassandra Renaud, Mélodie Seiler, Morgane Ziesel, Patrick Erbacher, and Malik Hellal. „Evaluating how cationic lipid affects mRNA-LNP physical properties and biodistribution“. European Journal of Pharmaceutics and Biopharmaceutics 195 (1. Februar 2024): 114077. https://doi.org/10.1016/j.ejpb.2023.08.002.
Hosseini-Kharat, Mahboubeh, Kristen E. Bremmell, and Clive A. Prestidge. “Why Do Lipid Nanoparticles Target the Liver? Understanding of Biodistribution and Liver-Specific Tropism.” Molecular Therapy. Methods & Clinical Development 33, no. 1 (February 15, 2025): 101436. https://doi.org/10.1016/j.omtm.2025.101436.
Reiser, A, D Woschée, N Mehrotra, R Krzysztoń, H H Strey, and J O Rädler. “Correlation of mRNA Delivery Timing and Protein Expression in Lipid-Based Transfection.” Integrative Biology 11, no. 9 (December 31, 2019): 362–71. https://doi.org/10.1093/intbio/zyz030.
Cedervall, Tommy, Iseult Lynch, Stina Lindman, Tord Berggård, Eva Thulin, Hanna Nilsson, Kenneth A. Dawson, and Sara Linse. “Understanding the Nanoparticle-Protein Corona Using Methods to Quantify Exchange Rates and Affinities of Proteins for Nanoparticles.” Proceedings of the National Academy of Sciences of the United States of America 104, no. 7 (February 13, 2007): 2050–55. https://doi.org/10.1073/pnas.0608582104.
Caracciolo, G., D. Pozzi, A. L. Capriotti, C. Cavaliere, S. Piovesana, G. La Barbera, A. Amici, and A. Laganà. “The Liposome–Protein Corona in Mice and Humans and Its Implications for in Vivo Delivery.” Journal of Materials Chemistry B 2, no. 42 (October 8, 2014): 7419–28. https://doi.org/10.1039/C4TB01316F.
Palchetti, Sara, Valentina Colapicchioni, Luca Digiacomo, Giulio Caracciolo, Daniela Pozzi, Anna Laura Capriotti, Giorgia La Barbera, and Aldo Laganà. “The Protein Corona of Circulating PEGylated Liposomes.” Biochimica et Biophysica Acta (BBA) - Biomembranes 1858, no. 2 (February 1, 2016): 189–96. https://doi.org/10.1016/j.bbamem.2015.11.012.
Foroozandeh, Parisa, and Azlan Abdul Aziz. “Merging Worlds of Nanomaterials and Biological Environment: Factors Governing Protein Corona Formation on Nanoparticles and Its Biological Consequences.” Nanoscale Research Letters 10, no. 1 (May 16, 2015): 221. https://doi.org/10.1186/s11671-015-0922-3.
Onishchenko, Natalia, Daria Tretiakova, and Elena Vodovozova. “Spotlight on the Protein Corona of Liposomes.” Acta Biomaterialia 134 (October 15, 2021): 57–78. https://doi.org/10.1016/j.actbio.2021.07.074.
Sebastiani, Federica, Marianna Yanez Arteta, Michael Lerche, Lionel Porcar, Christian Lang, Ryan A. Bragg, Charles S. Elmore, et al. “Apolipoprotein E Binding Drives Structural and Compositional Rearrangement of mRNA-Containing Lipid Nanoparticles.” ACS Nano 15, no. 4 (April 27, 2021): 6709–22. https://doi.org/10.1021/acsnano.0c10064.
Straten, Demian van, Helena Sork, Luuk van de Schepop, Rowan Frunt, Kariem Ezzat, and Raymond M. Schiffelers. „Biofluid Specific Protein Coronas Affect Lipid Nanoparticle Behavior in Vitro“. Journal of Controlled Release: Official Journal of the Controlled Release Society 373 (September 2024): 481–92. https://doi.org/10.1016/j.jconrel.2024.07.044.
Liu, Kai, Ralf Nilsson, Elisa Lázaro-Ibáñez, Hanna Duàn, Tasso Miliotis, Marie Strimfors, Michael Lerche, u. a. „Multiomics Analysis of Naturally Efficacious Lipid Nanoparticle Coronas Reveals High-Density Lipoprotein Is Necessary for Their Function“. Nature Communications 14, Nr. 1 (6. Juli 2023): 4007. https://doi.org/10.1038/s41467-023-39768-9.
Naasani, Imad. „Establishing the Pharmacokinetics of Genetic Vaccines Is Essential for Maximising Their Safety and Efficacy“. Clinical Pharmacokinetics 61, Nr. 7 (1. Juli 2022): 921–27. https://doi.org/10.1007/s40262-022-01149-8.
Huang, Ting, Jian Wang, Alexey Stukalov, Margaret K. R. Donovan, Shadi Ferdosi, Lucy Williamson, Seth Just, u. a. „Protein Coronas on Functionalized Nanoparticles Enable Quantitative and Precise Large-Scale Deep Plasma Proteomics“. bioRxiv: The Preprint Server for Biology, 29. August 2023, 2023.08.28.555225. https://doi.org/10.1101/2023.08.28.555225.
Costa Marques, Richard da, Natkritta Hüppe, Kai R. Speth, Jennifer Oberländer, Ingo Lieberwirth, Katharina Landfester, and Volker Mailänder. „Proteomics reveals time-dependent protein corona changes in the intracellular pathway“. Acta Biomaterialia 172 (1. Dezember 2023): 355–68. https://doi.org/10.1016/j.actbio.2023.10.010.
Voke, Elizabeth, Mariah Arral, Henry J. Squire, Teng-Jui Lin, Roxana Coreas, Alison Lui, Anthony T. Iavarone, Rebecca L. Pinals, Kathryn A. Whitehead, and Markita Landry. „Protein Corona Formed on Lipid Nanoparticles Compromises Delivery Efficiency of mRNA Cargo“. bioRxiv: The Preprint Server for Biology, 24. Januar 2025, 2025.01.20.633942. https://doi.org/10.1101/2025.01.20.633942.
Sengottiyan, Selvaraj, Alicja Mikolajczyk, Karolina Jagiełło, Marta Swirog, and Tomasz Puzyn. “Core, Coating, or Corona? The Importance of Considering Protein Coronas in Nano-QSPR Modeling of Zeta Potential.” ACS Nano 17, no. 3 (February 14, 2023): 1989–97. https://doi.org/10.1021/acsnano.2c06977.
Schoenmaker, Linde, Dominik Witzigmann, Jayesh A. Kulkarni, Rein Verbeke, Gideon Kersten, Wim Jiskoot, and Daan J. A. Crommelin. “mRNA-Lipid Nanoparticle COVID-19 Vaccines: Structure and Stability.” International Journal of Pharmaceutics 601 (May 15, 2021): 120586. https://doi.org/10.1016/j.ijpharm.2021.120586.
Verbeke, Rein, Ine Lentacker, Stefaan C. De Smedt, and Heleen Dewitte. “The Dawn of mRNA Vaccines: The COVID-19 Case.” Journal of Controlled Release 333 (May 10, 2021): 511–20. https://doi.org/10.1016/j.jconrel.2021.03.043.
Zhang, Lizhou, Kunal R. More, Amrita Ojha, Cody B. Jackson, Brian D. Quinlan, Hao Li, Wenhui He, Michael Farzan, Norbert Pardi, and Hyeryun Choe. “Effect of mRNA-LNP Components of Two Globally-Marketed COVID-19 Vaccines on Efficacy and Stability.” Npj Vaccines 8, no. 1 (October 11, 2023): 1–14. https://doi.org/10.1038/s41541-023-00751-6.
Tinari, Serena. “The EMA Covid-19 Data Leak, and What It Tells Us about mRNA Instability.” BMJ, March 10, 2021, n627. https://doi.org/10.1136/bmj.n627.
Brader, Mark L., Sean J. Williams, Jessica M. Banks, Wong H. Hui, Z. Hong Zhou, and Lin Jin. “Encapsulation State of Messenger RNA inside Lipid Nanoparticles.” Biophysical Journal 120, no. 14 (July 2021): 2766–70. https://doi.org/10.1016/j.bpj.2021.03.012.
Packer, Meredith, Dipendra Gyawali, Ravikiran Yerabolu, Joseph Schariter, and Phil White. „A Novel Mechanism for the Loss of mRNA Activity in Lipid Nanoparticle Delivery Systems“. Nature Communications 12, Nr. 1 (22. November 2021): 6777. https://doi.org/10.1038/s41467-021-26926-0.
Hashiba, Kazuki, Masamitsu Taguchi, Sachiko Sakamoto, Ayaka Otsu, Yoshiki Maeda, Hirofumi Ebe, Arimichi Okazaki, Hideyoshi Harashima, and Yusuke Sato. “Overcoming Thermostability Challenges in mRNA–Lipid Nanoparticle Systems with Piperidine-Based Ionizable Lipids.” Communications Biology 7, no. 1 (May 10, 2024): 1–13. https://doi.org/10.1038/s42003-024-06235-0.
Lamoot, Alexander, Annemiek Uvyn, Sabah Kasmi, and Bruno G. De Geest. „Covalent Cell Surface Conjugation of Nanoparticles by a Combination of Metabolic Labeling and Click Chemistry“. Angewandte Chemie (International Ed. in English) 60, Nr. 12 (15. März 2021): 6320–25. https://doi.org/10.1002/anie.202015625.
Birdsall, Robert E., Duanduan Han, Kellen DeLaney, Adam Kowalczyk, Razvan Cojocaru, Matthew Lauber, and Jon Le Huray. „Monitoring stability indicating impurities and aldehyde content in lipid nanoparticle raw material and formulated drugs“. Journal of Chromatography B 1234 (15. Februar 2024): 124005. https://doi.org/10.1016/j.jchromb.2024.124005.
“Structural Characterization of the Cationic Lipid Nanoparticle Component, ALC-0315, and Its Impurities Using Electronactivated Dissociation (EAD)-Based MS/MS Fragmentation.” Accessed May 26, 2025. https://sciex.com/tech-notes/biopharma/structural-characterization-of-the-cationic-lipid-nanoparticle-c.
Hermosilla, Jesús, Airan Alonso-García, Antonio Salmerón-García, José Cabeza-Barrera, Antonio L. Medina-Castillo, Raquel Pérez-Robles, and Natalia Navas. “Analysing the In-Use Stability of mRNA-LNP COVID-19 Vaccines ComirnatyTM (Pfizer) and SpikevaxTM (Moderna): A Comparative Study of the Particulate.” Vaccines 11, no. 11 (October 25, 2023): 1635. https://doi.org/10.3390/vaccines11111635.
Liao, Suiyang, Shuangyu Wang, Abishek Wadhwa, Alexandra Birkenshaw, Kevin Fox, Miffy Hok Yan Cheng, Irafasha C. Casmil, u. a. „Transfection Potency of Lipid Nanoparticles Containing mRNA Depends on Relative Loading Levels“. ACS Applied Materials & Interfaces 17, Nr. 2 (15. Januar 2025): 3097–3105. https://doi.org/10.1021/acsami.4c20077.
Renzi, Serena, Luca Digiacomo, Daniela Pozzi, Erica Quagliarini, Elisabetta Vulpis, Maria Valeria Giuli, Angelica Mancusi, u. a. „Structuring Lipid Nanoparticles, DNA, and Protein Corona into Stealth Bionanoarchitectures for in Vivo Gene Delivery“. Nature Communications 15, Nr. 1 (23. Oktober 2024): 9119. https://doi.org/10.1038/s41467-024-53569-8.
Patel, Siddharth, N Ashwanikumar, Emily Robinson, Allison DuRoss, Conroy Sun, Kerry E. Murphy-Benenato, Cosmin Mihai, Örn Almarsson, and Gaurav Sahay. „Boosting intracellular delivery of lipid nanoparticle-encapsulated messenger RNA“. Nano letters 17, Nr. 9 (13. September 2017): 5711–18. https://doi.org/10.1021/acs.nanolett.7b02664.
Di, Jiaxing, Zhili Du, Kangzeng Wu, Shanshan Jin, Xun Wang, Tonglei Li, and Yuhong Xu. „Biodistribution and Non-Linear Gene Expression of mRNA LNPs Affected by Delivery Route and Particle Size“. Pharmaceutical Research 39, Nr. 1 (Januar 2022): 105–14. https://doi.org/10.1007/s11095-022-03166-5.
Zhang, Tian, Han Yin, Yu Li, Haiyin Yang, Kun Ge, Jinchao Zhang, Qing Yuan, u. a. „Optimized lipid nanoparticles (LNPs) for organ-selective nucleic acids delivery in vivo“. iScience 27, Nr. 6 (21. Juni 2024): 109804. https://doi.org/10.1016/j.isci.2024.109804.
Lonez, Caroline, Michel Bessodes, Daniel Scherman, Michel Vandenbranden, Virginie Escriou, and Jean-Marie Ruysschaert. „Cationic lipid nanocarriers activate Toll-like receptor 2 and NLRP3 inflammasome pathways“. Nanomedicine: Nanotechnology, Biology and Medicine 10, Nr. 4 (1. Mai 2014): 775–82. https://doi.org/10.1016/j.nano.2013.12.003.
Lonez, Caroline, Michel Vandenbranden, and Jean-Marie Ruysschaert. „Cationic lipids activate intracellular signaling pathways“. Advanced Drug Delivery Reviews, Nanotoxicity: from bench to bedside, 64, Nr. 15 (1. Dezember 2012): 1749–58. https://doi.org/10.1016/j.addr.2012.05.009.
Akinc, Akin, William Querbes, Soma De, June Qin, Maria Frank-Kamenetsky, K. Narayanannair Jayaprakash, Muthusamy Jayaraman, et al. “Targeted Delivery of RNAi Therapeutics With Endogenous and Exogenous Ligand-Based Mechanisms.” Molecular Therapy 18, no. 7 (July 2010): 1357–64. https://doi.org/10.1038/mt.2010.85.
Paunovska, Kalina, Alejandro J. Da Silva Sanchez, Melissa P. Lokugamage, David Loughrey, Elisa Schrader Echeverri, Ana Cristian, Marine Z. C. Hatit, Philip J. Santangelo, Kun Zhao, and James E. Dahlman. „The Extent to Which Lipid Nanoparticles Require Apolipoprotein E and Low-Density Lipoprotein Receptor for Delivery Changes with Ionizable Lipid Structure“. Nano Letters 22, Nr. 24 (28. Dezember 2022): 10025–33. https://doi.org/10.1021/acs.nanolett.2c03741.
Chaudhary, Namit, Lisa N. Kasiewicz, Alexandra N. Newby, Mariah L. Arral, Saigopalakrishna S. Yerneni, Jilian R. Melamed, Samuel T. LoPresti, et al. “Amine Headgroups in Ionizable Lipids Drive Immune Responses to Lipid Nanoparticles by Binding to the Receptors TLR4 and CD1d.” Nature Biomedical Engineering 8, no. 11 (November 2024): 1483–98. https://doi.org/10.1038/s41551-024-01256-w.
Baimanov, Didar, Jing Wang, Yuchen Liu, Pingping Zheng, Shengtao Yu, Fen Liu, Jian Wang, et al. “Identification of Cell Receptors Responsible for Recognition and Binding of Lipid Nanoparticles.” Journal of the American Chemical Society 147, no. 9 (March 5, 2025): 7604–16. https://doi.org/10.1021/jacs.4c16987.
Massaro, Matteo, Suhong Wu, Gherardo Baudo, Haoran Liu, Scott Collum, Hyunho Lee, Cinzia Stigliano, Victor Segura-Ibarra, Harry Karmouty-Quintana, and Elvin Blanco. „Lipid nanoparticle-mediated mRNA delivery in lung fibrosis“. European Journal of Pharmaceutical Sciences 183 (1. April 2023): 106370. https://doi.org/10.1016/j.ejps.2023.106370.
Liu, Bingxin, Yamato Sajiki, Nicole Littlefield, Yongan Hu, William D. Stuart, Anusha Sridharan, Xuemei Cui, u. a. „PBAE-PEG-Based Lipid Nanoparticles for Lung Cell-Specific Gene Delivery“. Molecular Therapy: The Journal of the American Society of Gene Therapy 33, Nr. 3 (5. März 2025): 1154–65. https://doi.org/10.1016/j.ymthe.2025.01.037.
Khan, Omar F., Edmond W. Zaia, Siddharth Jhunjhunwala, Wen Xue, Wenxin Cai, Dong Soo Yun, Carmen M. Barnes, et al. “Dendrimer-Inspired Nanomaterials for the in Vivo Delivery of siRNA to Lung Vasculature.” Nano Letters 15, no. 5 (May 13, 2015): 3008–16. https://doi.org/10.1021/nl5048972.
Cheng, Qiang, Tuo Wei, Lukas Farbiak, Lindsay T. Johnson, Sean A. Dilliard, and Daniel J. Siegwart. “Selective Organ Targeting (SORT) Nanoparticles for Tissue-Specific mRNA Delivery and CRISPR–Cas Gene Editing.” Nature Nanotechnology 15, no. 4 (April 2020): 313–20. https://doi.org/10.1038/s41565-020-0669-6.
Johnson, Lindsay T., Di Zhang, Kejin Zhou, Sang M. Lee, Shuai Liu, Sean A. Dilliard, Lukas Farbiak, Sumanta Chatterjee, Yu-Hsuan Lin, and Daniel J. Siegwart. “Lipid Nanoparticle (LNP) Chemistry Can Endow Unique In Vivo RNA Delivery Fates within the Liver That Alter Therapeutic Outcomes in a Cancer Model.” Molecular Pharmaceutics 19, no. 11 (November 7, 2022): 3973–86. https://doi.org/10.1021/acs.molpharmaceut.2c00442.
Sakurai, Yu, Himeka Watanabe, Kazuma Nishio, Kohei Hashimoto, Atsuki Harada, Masaki Gomi, Masayoshi Suzuki, u. a. „pH-Responsive Lipid Nanoparticles Achieve Efficient mRNA Transfection in Brain Capillary Endothelial Cells“. Pharmaceutics 14, Nr. 8 (27. Juli 2022): 1560. https://doi.org/10.3390/pharmaceutics14081560.
Żak, Magdalena M., Keerat Kaur, Jimeen Yoo, Ann Anu Kurian, Matthew Adjmi, Gayatri Mainkar, Seonghun Yoon, and Lior Zangi. “Modified mRNA Formulation and Stability for Cardiac and Skeletal Muscle Delivery.” Pharmaceutics 15, no. 9 (August 22, 2023): 2176. https://doi.org/10.3390/pharmaceutics15092176.
Dey, Arindam K., Adrien Nougarède, Flora Clément, Carole Fournier, Evelyne Jouvin-Marche, Marie Escudé, Dorothée Jary, Fabrice P. Navarro, and Patrice N. Marche. “Tuning the Immunostimulation Properties of Cationic Lipid Nanocarriers for Nucleic Acid Delivery.” Frontiers in Immunology 12 (August 23, 2021). https://doi.org/10.3389/fimmu.2021.722411.
Chen, Jingan, Yue Xu, Muye Zhou, Shufen Xu, Andrew James Varley, Alex Golubovic, Rick Xing Ze Lu, et al. “Combinatorial Design of Ionizable Lipid Nanoparticles for Muscle-Selective mRNA Delivery with Minimized off-Target Effects.” Proceedings of the National Academy of Sciences 120, no. 50 (December 12, 2023): e2309472120. https://doi.org/10.1073/pnas.2309472120.
Younis, Mahmoud A., Yusuke Sato, Yaser H. A. Elewa, Yasuhiro Kon, and Hideyoshi Harashima. “Self-Homing Nanocarriers for mRNA Delivery to the Activated Hepatic Stellate Cells in Liver Fibrosis.” Journal of Controlled Release 353 (January 1, 2023): 685–98. https://doi.org/10.1016/j.jconrel.2022.12.020.
Khare, Purva, Sara X. Edgecomb, Christine M. Hamadani, Eden E. L. Tanner, and Devika S Manickam. “Lipid Nanoparticle-Mediated Drug Delivery to the Brain.” Advanced Drug Delivery Reviews 197 (June 2023): 114861. https://doi.org/10.1016/j.addr.2023.114861.
Han, Xuexiang, Hanwen Zhang, Kamila Butowska, Kelsey L. Swingle, Mohamad-Gabriel Alameh, Drew Weissman, and Michael J. Mitchell. “An Ionizable Lipid Toolbox for RNA Delivery.” Nature Communications 12, no. 1 (December 13, 2021): 7233. https://doi.org/10.1038/s41467-021-27493-0.
Swingle, Kelsey L., Hannah C. Safford, Hannah C. Geisler, Alex G. Hamilton, Ajay S. Thatte, Margaret M. Billingsley, Ryann A. Joseph, et al. “Ionizable Lipid Nanoparticles for In Vivo mRNA Delivery to the Placenta during Pregnancy.” Journal of the American Chemical Society 145, no. 8 (March 1, 2023): 4691–4706. https://doi.org/10.1021/jacs.2c12893.
Patel, Pratikkumar, Nurudeen Mohammed Ibrahim, and Kun Cheng. “The Importance of Apparent pKa in the Development of Nanoparticles Encapsulating siRNA and mRNA.” Trends in Pharmacological Sciences 42, no. 6 (June 2021): 448–60. https://doi.org/10.1016/j.tips.2021.03.002.
Ait-Oudhia, Sihem, Donald E. Mager, and Robert M. Straubinger. “Application of Pharmacokinetic and Pharmacodynamic Analysis to the Development of Liposomal Formulations for Oncology.” Pharmaceutics 6, no. 1 (March 18, 2014): 137–74. https://doi.org/10.3390/pharmaceutics6010137.
He, Hua, Dongfen Yuan, Yun Wu, and Yanguang Cao. “Pharmacokinetics and Pharmacodynamics Modeling and Simulation Systems to Support the Development and Regulation of Liposomal Drugs.” Pharmaceutics 11, no. 3 (March 7, 2019): 110. https://doi.org/10.3390/pharmaceutics11030110.
Müller, Judith A., Nathalie Schäffler, Thomas Kellerer, Gerlinde Schwake, Thomas S. Ligon, and Joachim O. Rädler. “Kinetics of RNA-LNP Delivery and Protein Expression.” European Journal of Pharmaceutics and Biopharmaceutics 197 (April 1, 2024): 114222. https://doi.org/10.1016/j.ejpb.2024.114222.
Ermilova, Inna, and Jan Swenson. “Ionizable Lipids Penetrate Phospholipid Bilayers with High Phase Transition Temperatures: Perspectives from Free Energy Calculations.” Chemistry and Physics of Lipids 253 (July 1, 2023): 105294. https://doi.org/10.1016/j.chemphyslip.2023.105294.
Pilkington, Emily H., Estelle J. A. Suys, Natalie L. Trevaskis, Adam K. Wheatley, Danijela Zukancic, Azizah Algarni, Hareth Al-Wassiti, et al. “From Influenza to COVID-19: Lipid Nanoparticle mRNA Vaccines at the Frontiers of Infectious Diseases.” Acta Biomaterialia 131 (September 1, 2021): 16–40. https://doi.org/10.1016/j.actbio.2021.06.023.
Fell, Valentin H. K., Tim Kramer, Andreas Heindl, and Olivia M. Merkel. “Prediction of the Apparent p Ka Value of Lipid Nanoparticles by Density Functional Theory.” ACS Materials Au 5, no. 3 (May 14, 2025): 451–57. https://doi.org/10.1021/acsmaterialsau.4c00158.
Chatterjee, Sushmita, Edo Kon, Preeti Sharma, and Dan Peer. “Endosomal Escape: A Bottleneck for LNP-Mediated Therapeutics.” Proceedings of the National Academy of Sciences 121, no. 11 (March 12, 2024): e2307800120. https://doi.org/10.1073/pnas.2307800120.
Liu, Haiyin, Moore Z. Chen, Thomas Payne, Christopher J.H. Porter, Colin W. Pouton, and Angus P.R. Johnston. “Beyond the Endosomal Bottleneck: Understanding the Efficiency of mRNA/LNP Delivery.” Advanced Functional Materials 34, no. 39 (September 2024): 2404510. https://doi.org/10.1002/adfm.202404510.
Couture-Senécal, Julien, Grayson Tilstra, and Omar F. Khan. “Engineering Ionizable Lipids for Rapid Biodegradation Balances mRNA Vaccine Efficacy and Tolerability.” bioRxiv, August 3, 2024. https://doi.org/10.1101/2024.08.02.606386.
Hald Albertsen, Camilla, Jayesh A. Kulkarni, Dominik Witzigmann, Marianne Lind, Karsten Petersson, and Jens B. Simonsen. “The Role of Lipid Components in Lipid Nanoparticles for Vaccines and Gene Therapy.” Advanced Drug Delivery Reviews 188 (September 2022): 114416. https://doi.org/10.1016/j.addr.2022.114416.
Yartsev, Alex. “Single and Multiple Compartment Models of Drug Distribution | Deranged Physiology.” Accessed May 30, 2025. https://derangedphysiology.com/main/cicm-primary-exam/pharmacokinetics/Chapter-201/single-and-multiple-compartment-models-drug-distribution.
Yashiki, T., T. Matsuzawa, T. Kondo, Y. Uda, and T. Shima. “Studies on the Metabolic Fate and the Pharmacokinetics of 5-n-Butyl-1-Cyclohexyl-2,4,6-Trioxoperhydropyrimidine (BCP) in Man. I. Identification of the Metabolites of BCP in Human Urine.” Chemical & Pharmaceutical Bulletin 19, no. 3 (March 1971): 468–77. https://doi.org/10.1248/cpb.19.468.
Tulliez, J. E., E. F. Durand, and G. F. Bories. “Metabolic Fate and Pharmacokinetics of Tissue Residues of the Anticoccidial Drug Robenidine in the Rabbit. Incidence of Coprophagy on Its Bioavailability.” Journal of Agricultural and Food Chemistry 30, no. 6 (1982): 1071–75. https://doi.org/10.1021/jf00114a016.
Liu, Liu, Yiming Liu, Xiaojie Zhou, Zhiwei Xu, Yehui Zhang, Liting Ji, Chunlan Hong, and Changyu Li. “Analyzing the Metabolic Fate of Oral Administration Drugs: A Review and State-of-the-Art Roadmap.” Frontiers in Pharmacology 13 (October 7, 2022). https://doi.org/10.3389/fphar.2022.962718.
Granados-Riveron, Javier T., and Guillermo Aquino-Jarquin. “Engineering of the Current Nucleoside-Modified mRNA-LNP Vaccines against SARS-CoV-2.” Biomedicine & Pharmacotherapy 142 (October 2021): 111953. https://doi.org/10.1016/j.biopha.2021.111953.
Lai, Chih‐Jen, Dokyun Kim, Seokmin Kang, Kun Li, Inho Cha, Akimi Sasaki, Jose Porras, Tian Xia, and Jae U. Jung. “Viral Codon Optimization on SARS‐CoV‐2 Spike Boosts Immunity in the Development of COVID‐19 mRNA Vaccines.” Journal of Medical Virology 95, no. 10 (October 2023): e29183. https://doi.org/10.1002/jmv.29183.
Nance, Kellie D., and Jordan L. Meier. “Modifications in an Emergency: The Role of N1-Methylpseudouridine in COVID-19 Vaccines.” ACS Central Science 7, no. 5 (May 26, 2021): 748–56. https://doi.org/10.1021/acscentsci.1c00197.
Morais, Pedro, Hironori Adachi, and Yi-Tao Yu. “The Critical Contribution of Pseudouridine to mRNA COVID-19 Vaccines.” Frontiers in Cell and Developmental Biology 9 (November 4, 2021). https://doi.org/10.3389/fcell.2021.789427.
Teo, Shyh Poh. “Review of COVID-19 mRNA Vaccines: BNT162b2 and mRNA-1273.” Journal of Pharmacy Practice 35, no. 6 (December 2022): 947–51. https://doi.org/10.1177/08971900211009650.
Didiano, Dominic, and Oliver Hobert. “Molecular Architecture of a miRNA-Regulated 3′ UTR.” RNA 14, no. 7 (July 2008): 1297–1317. https://doi.org/10.1261/rna.1082708.
Gu, Shuo, Lan Jin, Feijie Zhang, Peter Sarnow, and Mark A. Kay. “The Biological Basis for microRNA Target Restriction to the 3’ Untranslated Region in Mammalian mRNAs.” Nature Structural & Molecular Biology 16, no. 2 (February 2009): 144–50. https://doi.org/10.1038/nsmb.1552.
Zhang, Ying, Chong Tang, Tian Yu, Ruirui Zhang, Huili Zheng, and Wei Yan. “MicroRNAs Control mRNA Fate by Compartmentalization Based on 3′ UTR Length in Male Germ Cells.” Genome Biology 18, no. 1 (June 15, 2017): 105. https://doi.org/10.1186/s13059-017-1243-x.
Hart, Martin, Fabian Kern, Claudia Fecher-Trost, Lena Krammes, Ernesto Aparicio, Annika Engel, Pascal Hirsch, et al. “Experimental Capture of miRNA Targetomes: Disease-Specific 3′UTR Library-Based miRNA Targetomics for Parkinson’s Disease.” Experimental & Molecular Medicine 56, no. 4 (April 2024): 935–45. https://doi.org/10.1038/s12276-024-01202-5.
Hao, Peiqi, Jiaojiao Yu, Richard Ward, Yin Liu, Qiao Hao, Su An, and Tianrui Xu. “Eukaryotic Translation Initiation Factors as Promising Targets in Cancer Therapy.” Cell Communication and Signaling 18, no. 1 (December 2020): 175. https://doi.org/10.1186/s12964-020-00607-9.
Weiss, Benjamin, and Rivka Dikstein. “Unraveling the Landscapes and Regulation of Scanning, Leaky Scanning, and 48S Initiation Complex Conformations.” Cell Reports 43, no. 5 (May 28, 2024): 114126. https://doi.org/10.1016/j.celrep.2024.114126.
Marintchev, Assen. “Exploring the Interaction Dynamics of Eukaryotic Translation Initiation Factor 2.” Biochemical Society Transactions, May 23, 2025, BST20253022. https://doi.org/10.1042/BST20253022.
Mestre-Fos, Santi, Lucas Ferguson, Marena Trinidad, Nicholas T Ingolia, and Jamie Hd Cate. “eIF3 Engages with 3’-UTR Termini of Highly Translated mRNAs,” December 27, 2024. https://doi.org/10.7554/eLife.102977.2.
Orlandini Von Niessen, Alexandra G., Marco A. Poleganov, Corina Rechner, Arianne Plaschke, Lena M. Kranz, Stephanie Fesser, Mustafa Diken, et al. “Improving mRNA-Based Therapeutic Gene Delivery by Expression-Augmenting 3′ UTRs Identified by Cellular Library Screening.” Molecular Therapy 27, no. 4 (April 2019): 824–36. https://doi.org/10.1016/j.ymthe.2018.12.011.
Jee, David, and Eric C. Lai. “Alteration of miRNA Activity via Context-Specific Modifications of Argonaute Proteins.” Trends in Cell Biology 24, no. 9 (September 2014): 546–53. https://doi.org/10.1016/j.tcb.2014.04.008.
Krawczyk, Paweł S., Michał Mazur, Wiktoria Orzeł, Olga Gewartowska, Sebastian Jeleń, Wiktor Antczak, Karolina Kasztelan, et al. “Re-Adenylation by TENT5A Enhances Efficacy of SARS-CoV-2 mRNA Vaccines.” Nature 641, no. 8064 (May 2025): 984–92. https://doi.org/10.1038/s41586-025-08842-1.
Chen, Xiongzhi, and Sanat K. Sarkar. “On Benjamini–Hochberg Procedure Applied to Mid p-Values.” Journal of Statistical Planning and Inference 205 (March 1, 2020): 34–45. https://doi.org/10.1016/j.jspi.2019.06.001.
Warkocki, Zbigniew, Vladyslava Liudkovska, Olga Gewartowska, Seweryn Mroczek, and Andrzej Dziembowski. “Terminal Nucleotidyl Transferases (TENTs) in Mammalian RNA Metabolism.” Philosophical Transactions of the Royal Society B: Biological Sciences 373, no. 1762 (December 19, 2018): 20180162. https://doi.org/10.1098/rstb.2018.0162.
Lim, Jaechul, Dongwan Kim, Young-Suk Lee, Minju Ha, Mihye Lee, Jinah Yeo, Hyeshik Chang, Jaewon Song, Kwangseog Ahn, and V Narry Kim. “Mixed Tailing by TENT4A and TENT4B Shields mRNA from Rapid Deadenylation.” Science (New York, N.Y.) 361, no. 6403 (August 1, 2018): 701–4. https://doi.org/10.1126/science.aam5794.
Kim, Dongwan, Young-suk Lee, Soo-Jin Jung, Jinah Yeo, Jenny J. Seo, Young-Yoon Lee, Jaechul Lim, et al. “Viral Hijacking of the TENT4–ZCCHC14 Complex Protects Viral RNAs via Mixed Tailing.” Nature Structural & Molecular Biology 27, no. 6 (June 2020): 581–88. https://doi.org/10.1038/s41594-020-0427-3.
Kuzmin, Ivan V., Ruben Soto Acosta, Layne Pruitt, Perry T. Wasdin, Kritika Kedarinath, Keziah R. Hernandez, Kristyn A. Gonzales, et al. “Comparison of Uridine and N1-Methylpseudouridine mRNA Platforms in Development of an Andes Virus Vaccine.” Nature Communications 15, no. 1 (July 30, 2024): 6421. https://doi.org/10.1038/s41467-024-50774-3.
Cohn, Waldo E., and Elliot Volkin. “Nucleoside-5′-Phosphates from Ribonucleic Acid.” Nature 167, no. 4247 (March 1951): 483–84. https://doi.org/10.1038/167483a0.
Borchardt, Erin K., Nicole M. Martinez, and Wendy V. Gilbert. “Regulation and Function of RNA Pseudouridylation in Human Cells.” Annual Review of Genetics 54 (November 23, 2020): 309–36. https://doi.org/10.1146/annurev-genet-112618-043830.
Mengel-Jorgensen, J. “Detection of Pseudouridine and Other Modifications in tRNA by Cyanoethylation and MALDI Mass Spectrometry.” Nucleic Acids Research 30, no. 23 (December 1, 2002): 135e–1135. https://doi.org/10.1093/nar/gnf135.
Argoudelis, A. D., and S. A. Mizsak. “1-Methylpseudouridine, a Metabolite of Streptomyces Platensis.” The Journal of Antibiotics 29, no. 8 (August 1976): 818–23. https://doi.org/10.7164/antibiotics.29.818.
Wurm, Jan Philip, Marco Griese, Ute Bahr, Martin Held, Alexander Heckel, Michael Karas, Jörg Soppa, and Jens Wöhnert. “Identification of the Enzyme Responsible for N1-Methylation of Pseudouridine 54 in Archaeal tRNAs.” RNA 18, no. 3 (March 2012): 412–20. https://doi.org/10.1261/rna.028498.111.
Hori, Hiroyuki. “Methylated Nucleosides in tRNA and tRNA Methyltransferases.” Frontiers in Genetics 5 (May 23, 2014). https://doi.org/10.3389/fgene.2014.00144.
Ero, Rya, Lauri Peil, Aivar Liiv, and Jaanus Remme. “Identification of Pseudouridine Methyltransferase in Escherichia Coli.” RNA 14, no. 10 (October 2008): 2223–33. https://doi.org/10.1261/rna.1186608.
Pang, H, M Ihara, Y Kuchino, S Nishimura, R Gupta, C R Woese, and J A McCloskey. “Structure of a Modified Nucleoside in Archaebacterial tRNA Which Replaces Ribosylthymine. 1-Methylpseudouridine.” Journal of Biological Chemistry 257, no. 7 (April 1982): 3589–92. https://doi.org/10.1016/S0021-9258(18)34820-8.
Parr, Callum J C, Shunsuke Wada, Kenjiro Kotake, Shigetoshi Kameda, Satoshi Matsuura, Souhei Sakashita, Soyoung Park, Hiroshi Sugiyama, Yi Kuang, and Hirohide Saito. “N 1-Methylpseudouridine Substitution Enhances the Performance of Synthetic mRNA Switches in Cells.” Nucleic Acids Research 48, no. 6 (April 6, 2020): e35–e35. https://doi.org/10.1093/nar/gkaa070.
Chen, Tien-Hao, Vladimir Potapov, Nan Dai, Jennifer L. Ong, and Bijoyita Roy. “N1-Methyl-Pseudouridine Is Incorporated with Higher Fidelity than Pseudouridine in Synthetic RNAs.” Scientific Reports 12, no. 1 (July 29, 2022): 13017. https://doi.org/10.1038/s41598-022-17249-1.
Monroe, Jeremy, Daniel E. Eyler, Lili Mitchell, Indrajit Deb, Abigail Bojanowski, Pooja Srinivas, Christine M. Dunham, Bijoyita Roy, Aaron T. Frank, and Kristin S. Koutmou. “N1-Methylpseudouridine and Pseudouridine Modifications Modulate mRNA Decoding during Translation.” Nature Communications 15, no. 1 (September 16, 2024): 8119. https://doi.org/10.1038/s41467-024-51301-0.
Sachs, Alan B. “Messenger RNA Degradation in Eukaryotes.” Cell 74, no. 3 (August 13, 1993): 413–21. https://doi.org/10.1016/0092-8674(93)80043-E.
Karikó, Katalin, Prakash Bhuyan, John Capodici, and Drew Weissman. “Small Interfering RNAs Mediate Sequence-Independent Gene Suppression and Induce Immune Activation by Signaling through Toll-Like Receptor 3.” The Journal of Immunology 172, no. 11 (June 1, 2004): 6545–49. https://doi.org/10.4049/jimmunol.172.11.6545.
Karikó, Katalin, Michael Buckstein, Houping Ni, and Drew Weissman. “Suppression of RNA Recognition by Toll-like Receptors: The Impact of Nucleoside Modification and the Evolutionary Origin of RNA.” Immunity 23, no. 2 (August 2005): 165–75. https://doi.org/10.1016/j.immuni.2005.06.008.
Karikó, Katalin, Hiromi Muramatsu, Frank A Welsh, János Ludwig, Hiroki Kato, Shizuo Akira, and Drew Weissman. “Incorporation of Pseudouridine Into mRNA Yields Superior Nonimmunogenic Vector With Increased Translational Capacity and Biological Stability.” Molecular Therapy 16, no. 11 (November 2008): 1833–40. https://doi.org/10.1038/mt.2008.200.
Holtkamp, Silke, Sebastian Kreiter, Abderraouf Selmi, Petra Simon, Michael Koslowski, Christoph Huber, Özlem Türeci, and Ugur Sahin. “Modification of Antigen-Encoding RNA Increases Stability, Translational Efficacy, and T-Cell Stimulatory Capacity of Dendritic Cells.” Blood 108, no. 13 (December 15, 2006): 4009–17. https://doi.org/10.1182/blood-2006-04-015024.
Sahin, Ugur, Katalin Karikó, and Özlem Türeci. “mRNA-Based Therapeutics — Developing a New Class of Drugs.” Nature Reviews Drug Discovery 13, no. 10 (October 2014): 759–80. https://doi.org/10.1038/nrd4278.
Sahin, Ugur, Evelyna Derhovanessian, Matthias Miller, Björn-Philipp Kloke, Petra Simon, Martin Löwer, Valesca Bukur, et al. “Personalized RNA Mutanome Vaccines Mobilize Poly-Specific Therapeutic Immunity against Cancer.” Nature 547, no. 7662 (July 2017): 222–26. https://doi.org/10.1038/nature23003.
Kranz, Lena M., Mustafa Diken, Heinrich Haas, Sebastian Kreiter, Carmen Loquai, Kerstin C. Reuter, Martin Meng, et al. “Systemic RNA Delivery to Dendritic Cells Exploits Antiviral Defence for Cancer Immunotherapy.” Nature 534, no. 7607 (June 2016): 396–401. https://doi.org/10.1038/nature18300.
Pang, H, M Ihara, Y Kuchino, S Nishimura, R Gupta, C R Woese, and J A McCloskey. “Structure of a Modified Nucleoside in Archaebacterial tRNA Which Replaces Ribosylthymine. 1-Methylpseudouridine.” Journal of Biological Chemistry 257, no. 7 (April 1982): 3589–92. https://doi.org/10.1016/S0021-9258(18)34820-8.
Meroueh, May, Patrick J. Grohar, Jian Qiu, John SantaLucia, Stephen A. Scaringe, and Christine S. Chow. “Unique Structural and Stabilizing Roles for the Individual Pseudouridine Residues in the 1920 Region of Escherichia Coli 23S rRNA.” Nucleic Acids Research 28, no. 10 (May 15, 2000): 2075–83. https://doi.org/10.1093/nar/28.10.2075.
Carlile, Thomas M., Maria F. Rojas-Duran, Boris Zinshteyn, Hakyung Shin, Kristen M. Bartoli, and Wendy V. Gilbert. “Pseudouridine Profiling Reveals Regulated mRNA Pseudouridylation in Yeast and Human Cells.” Nature 515, no. 7525 (November 6, 2014): 143–46. https://doi.org/10.1038/nature13802.
Spenkuch, Felix, Yuri Motorin, and Mark Helm. “Pseudouridine: Still Mysterious, but Never a Fake (Uridine)!” RNA Biology 11, no. 12 (January 23, 2015): 1540–54. https://doi.org/10.4161/15476286.2014.992278.
Mauger, David M., B. Joseph Cabral, Vladimir Presnyak, Stephen V. Su, David W. Reid, Brooke Goodman, Kristian Link, et al. “mRNA Structure Regulates Protein Expression through Changes in Functional Half-Life.” Proceedings of the National Academy of Sciences 116, no. 48 (November 26, 2019): 24075–83. https://doi.org/10.1073/pnas.1908052116.
Karikó, Katalin, Houping Ni, John Capodici, Marc Lamphier, and Drew Weissman. “mRNA Is an Endogenous Ligand for Toll-like Receptor 3.” Journal of Biological Chemistry 279, no. 13 (March 2004): 12542–50. https://doi.org/10.1074/jbc.M310175200.
Harada, A., N. Sekido, T. Akahoshi, T. Wada, N. Mukaida, and K. Matsushima. “Essential Involvement of Interleukin-8 (IL-8) in Acute Inflammation.” Journal of Leukocyte Biology 56, no. 5 (November 1994): 559–64.
Medzhitov, R., P. Preston-Hurlburt, and C. A. Janeway. “A Human Homologue of the Drosophila Toll Protein Signals Activation of Adaptive Immunity.” Nature 388, no. 6640 (July 24, 1997): 394–97. https://doi.org/10.1038/41131.
Karimi, Khalil, Hadi Sarir, Esmaeil Mortaz, Joost J. Smit, Hossein Hosseini, Sjef J. De Kimpe, Frans P. Nijkamp, and Gert Folkerts. “Toll-like Receptor-4 Mediates Cigarette Smoke-Induced Cytokine Production by Human Macrophages.” Respiratory Research 7, no. 1 (April 19, 2006): 66. https://doi.org/10.1186/1465-9921-7-66.
He, W., T. Qu, Q. Yu, Z. Wang, H. Lv, J. Zhang, X. Zhao, and P. Wang. “LPS Induces IL ‐8 Expression through TLR 4, M y D 88, NF ‐kappa B and MAPK Pathways in Human Dental Pulp Stem Cells.” International Endodontic Journal 46, no. 2 (February 2013): 128–36. https://doi.org/10.1111/j.1365-2591.2012.02096.x.
Cesta, Maria Candida, Mara Zippoli, Carolina Marsiglia, Elizabeth Marie Gavioli, Flavio Mantelli, Marcello Allegretti, and Robert A. Balk. “The Role of Interleukin-8 in Lung Inflammation and Injury: Implications for the Management of COVID-19 and Hyperinflammatory Acute Respiratory Distress Syndrome.” Frontiers in Pharmacology 12 (January 12, 2022). https://doi.org/10.3389/fphar.2021.808797.
He, Wei, Osmond Rebello, Rocco Savino, Rosa Terracciano, Carole Schuster-Klein, Beatrice Guardiola, and Kathrin Maedler. “TLR4 Triggered Complex Inflammation in Human Pancreatic Islets.” Biochimica Et Biophysica Acta. Molecular Basis of Disease 1865, no. 1 (January 2019): 86–97. https://doi.org/10.1016/j.bbadis.2018.09.030.
Versel, Julia, Adriana Cantos, Monica Feliz R. Castillo, Evangelia Fatourou, James Sinacore, and Angelos Halaris. “Interleukin-8 Is a Potential Inflammation Biomarker in Major Depressive Disorder.” Journal of Affective Disorders Reports 17 (July 1, 2024): 100828. https://doi.org/10.1016/j.jadr.2024.100828.
Svitkin, Yuri V., Yi Min Cheng, Tirtha Chakraborty, Vladimir Presnyak, Matthias John, and Nahum Sonenberg. “N1-Methyl-Pseudouridine in mRNA Enhances Translation through eIF2α-Dependent and Independent Mechanisms by Increasing Ribosome Density.” Nucleic Acids Research 45, no. 10 (June 2, 2017): 6023–36. https://doi.org/10.1093/nar/gkx135.
Liao, Pei-Yu, Prateek Gupta, Alexey N. Petrov, Jonathan D. Dinman, and Kelvin H. Lee. “A New Kinetic Model Reveals the Synergistic Effect of E-, P- and A-Sites on +1 Ribosomal Frameshifting.” Nucleic Acids Research 36, no. 8 (May 2008): 2619–29. https://doi.org/10.1093/nar/gkn100.
Karijolich, John, and Yi-Tao Yu. “Converting Nonsense Codons into Sense Codons by Targeted Pseudouridylation.” Nature 474, no. 7351 (June 15, 2011): 395–98. https://doi.org/10.1038/nature10165.
Atkins, John F., Gary Loughran, Pramod R. Bhatt, Andrew E. Firth, and Pavel V. Baranov. “Ribosomal Frameshifting and Transcriptional Slippage: From Genetic Steganography and Cryptography to Adventitious Use.” Nucleic Acids Research 44, no. 15 (September 6, 2016): 7007–78. https://doi.org/10.1093/nar/gkw530.
Xia, Xuhua. “Detailed Dissection and Critical Evaluation of the Pfizer/BioNTech and Moderna mRNA Vaccines.” Vaccines 9, no. 7 (July 3, 2021): 734. https://doi.org/10.3390/vaccines9070734.
Mulroney, Thomas E., Tuija Pöyry, Juan Carlos Yam-Puc, Maria Rust, Robert F. Harvey, Lajos Kalmar, Emily Horner, et al. “N1-Methylpseudouridylation of mRNA Causes +1 Ribosomal Frameshifting.” Nature 625, no. 7993 (January 2024): 189–94. https://doi.org/10.1038/s41586-023-06800-3.
Cerneckis, Jonas, Qi Cui, Chuan He, Chengqi Yi, and Yanhong Shi. “Decoding Pseudouridine: An Emerging Target for Therapeutic Development.” Trends in Pharmacological Sciences 43, no. 6 (June 1, 2022): 522–35. https://doi.org/10.1016/j.tips.2022.03.008.
Adachi, Hironori, and Yi-Tao Yu. “Pseudouridine-Mediated Stop Codon Readthrough in S. Cerevisiae Is Sequence Context–Independent.” RNA 26, no. 9 (September 2020): 1247–56. https://doi.org/10.1261/rna.076042.120.
Veenstra, Timothy D., Brad Pauley, Elisha Injeti, and Rocco J. Rotello. “In Vitro Characterization of SARS-CoV-2 Protein Translated from the Moderna mRNA-1273 Vaccine.” Allergy and Immunology, March 2, 2022. https://doi.org/10.1101/2022.03.01.22271618.
Lin, Ting‐Yu, Rahul Mehta, and Sebastian Glatt. “Pseudouridines in RNAs: Switching Atoms Means Shifting Paradigms.” FEBS Letters 595, no. 18 (September 2021): 2310–22. https://doi.org/10.1002/1873-3468.14188.
Andries, Oliwia, Séan Mc Cafferty, Stefaan C. De Smedt, Ron Weiss, Niek N. Sanders, and Tasuku Kitada. “N1-Methylpseudouridine-Incorporated mRNA Outperforms Pseudouridine-Incorporated mRNA by Providing Enhanced Protein Expression and Reduced Immunogenicity in Mammalian Cell Lines and Mice.” Journal of Controlled Release 217 (November 2015): 337–44. https://doi.org/10.1016/j.jconrel.2015.08.051.
Ho, Lyana L. Y., Gabriel H. A. Schiess, Pâmella Miranda, Gerald Weber, and Kira Astakhova. “Pseudouridine and N1-Methylpseudouridine as Potent Nucleotide Analogues for RNA Therapy and Vaccine Development.” RSC Chemical Biology 5, no. 5 (n.d.): 418–25. https://doi.org/10.1039/d4cb00022f.
Stockert, Jennifer A, Akriti Gupta, Bryan Herzog, Shalini S Yadav, Ashutosh K Tewari, and Kamlesh K Yadav. “Predictive Value of Pseudouridine in Prostate Cancer.” American Journal of Clinical and Experimental Urology 7, no. 4 (August 25, 2019): 262–72.
Hanna, Nazeeh, Ari Heffes-Doon, Xinhua Lin, Claudia Manzano De Mejia, Bishoy Botros, Ellen Gurzenda, and Amrita Nayak. “Detection of Messenger RNA COVID-19 Vaccines in Human Breast Milk.” JAMA Pediatrics 176, no. 12 (December 1, 2022): 1268. https://doi.org/10.1001/jamapediatrics.2022.3581.
Fertig, Tudor Emanuel, Leona Chitoiu, Daciana Silvia Marta, Victor-Stefan Ionescu, Valeriu Bogdan Cismasiu, Eugen Radu, Giulia Angheluta, et al. “Vaccine mRNA Can Be Detected in Blood at 15 Days Post-Vaccination.” Biomedicines 10, no. 7 (June 28, 2022): 1538. https://doi.org/10.3390/biomedicines10071538.
Castruita, Jose Alfredo Samaniego, Uffe Vest Schneider, Sarah Mollerup, Thomas Daell Leineweber, Nina Weis, Jens Bukh, Martin Schou Pedersen, and Henrik Westh. “SARS‐CoV ‐2 Spike mRNA Vaccine Sequences Circulate in Blood up to 28 Days after COVID ‐19 Vaccination.” APMIS 131, no. 3 (March 2023): 128–32. https://doi.org/10.1111/apm.13294.
Krauson, Aram J., Faye Victoria C. Casimero, Zakir Siddiquee, and James R. Stone. “Duration of SARS-CoV-2 mRNA Vaccine Persistence and Factors Associated with Cardiac Involvement in Recently Vaccinated Patients.” Npj Vaccines 8, no. 1 (September 27, 2023): 1–8. https://doi.org/10.1038/s41541-023-00742-7.
Magen, Eli, Sumit Mukherjee, Mahua Bhattacharya, Rajesh Detroja, Eugene Merzon, Idan Blum, Alejandro Livoff, et al. “Clinical and Molecular Characterization of a Rare Case of BNT162b2 mRNA COVID-19 Vaccine-Associated Myositis.” Vaccines 10, no. 7 (July 16, 2022): 1135. https://doi.org/10.3390/vaccines10071135.
Röltgen, Katharina, Sandra C.A. Nielsen, Oscar Silva, Sheren F. Younes, Maxim Zaslavsky, Cristina Costales, Fan Yang, et al. “Immune Imprinting, Breadth of Variant Recognition, and Germinal Center Response in Human SARS-CoV-2 Infection and Vaccination.” Cell 185, no. 6 (March 2022): 1025-1040.e14. https://doi.org/10.1016/j.cell.2022.01.018.
Lockhart, John, John Canfield, Ezinne Francess Mong, Jeffrey VanWye, and Hana Totary-Jain. “Nucleotide Modification Alters MicroRNA-Dependent Silencing of MicroRNA Switches.” Molecular Therapy. Nucleic Acids 14 (December 18, 2018): 339–50. https://doi.org/10.1016/j.omtn.2018.12.007.
Bak, Rasmus O., and Jacob Giehm Mikkelsen. “miRNA Sponges: Soaking up miRNAs for Regulation of Gene Expression.” WIREs RNA 5, no. 3 (May 2014): 317–33. https://doi.org/10.1002/wrna.1213.
Valinezhad Orang, Ayla, Reza Safaralizadeh, and Mina Kazemzadeh-Bavili. “Mechanisms of miRNA-Mediated Gene Regulation from Common Downregulation to mRNA-Specific Upregulation.” International Journal of Genomics 2014 (2014): 970607. https://doi.org/10.1155/2014/970607.
Georgakopoulos-Soares, Ilias, Guillermo E. Parada, and Martin Hemberg. “Secondary Structures in RNA Synthesis, Splicing and Translation.” Computational and Structural Biotechnology Journal 20 (May 27, 2022): 2871–84. https://doi.org/10.1016/j.csbj.2022.05.041.
Gan, Hin Hark, and Kristin C. Gunsalus. “The Role of Tertiary Structure in MicroRNA Target Recognition.” Methods in Molecular Biology (Clifton, N.J.) 1970 (2019): 43–64. https://doi.org/10.1007/978-1-4939-9207-2_4.
Carlile, Thomas M., Nicole M. Martinez, Cassandra Schaening, Amanda Su, Tristan A. Bell, Boris Zinshteyn, and Wendy V. Gilbert. “mRNA Structure Determines Modification by Pseudouridine Synthase 1.” Nature Chemical Biology 15, no. 10 (October 2019): 966–74. https://doi.org/10.1038/s41589-019-0353-z.



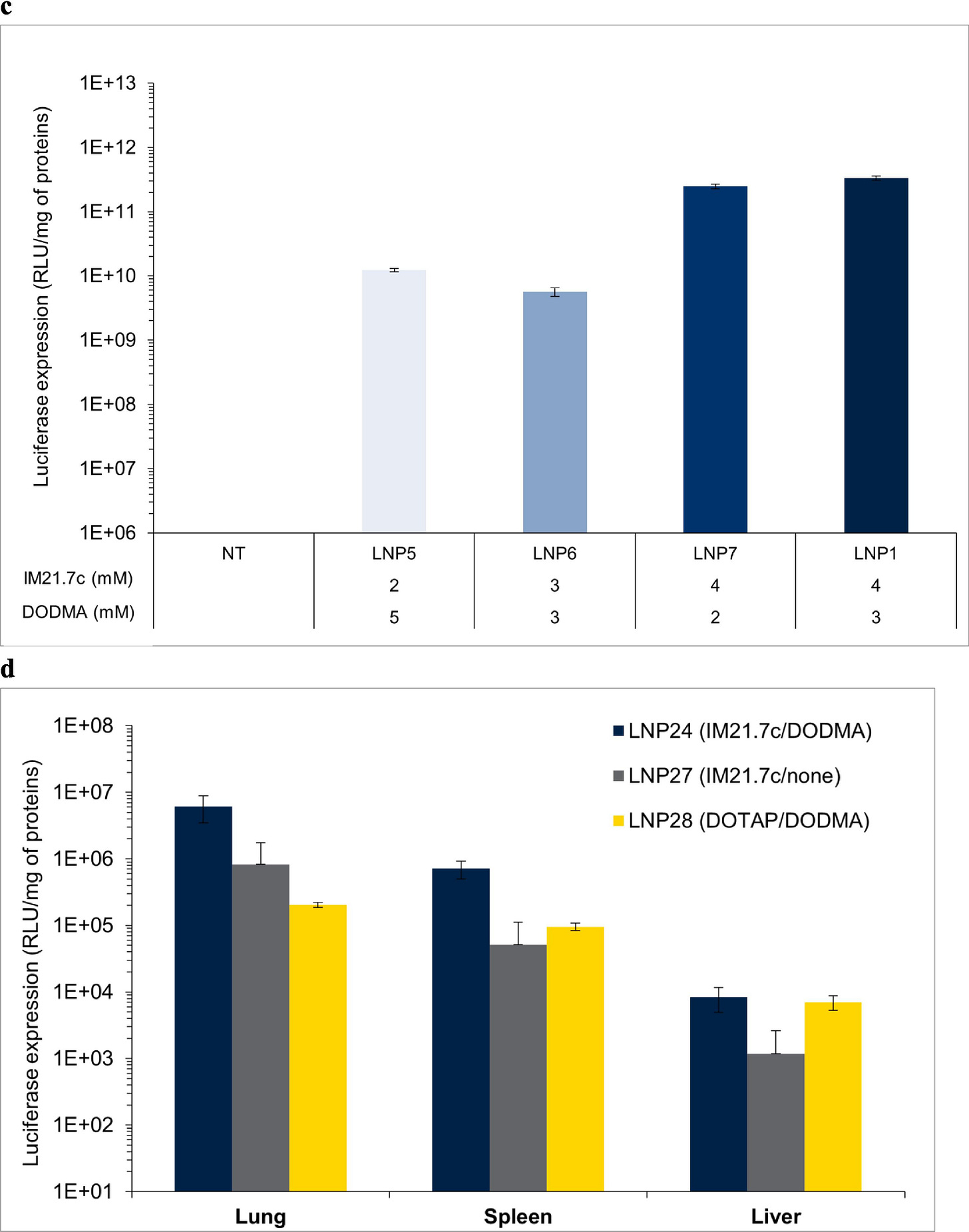
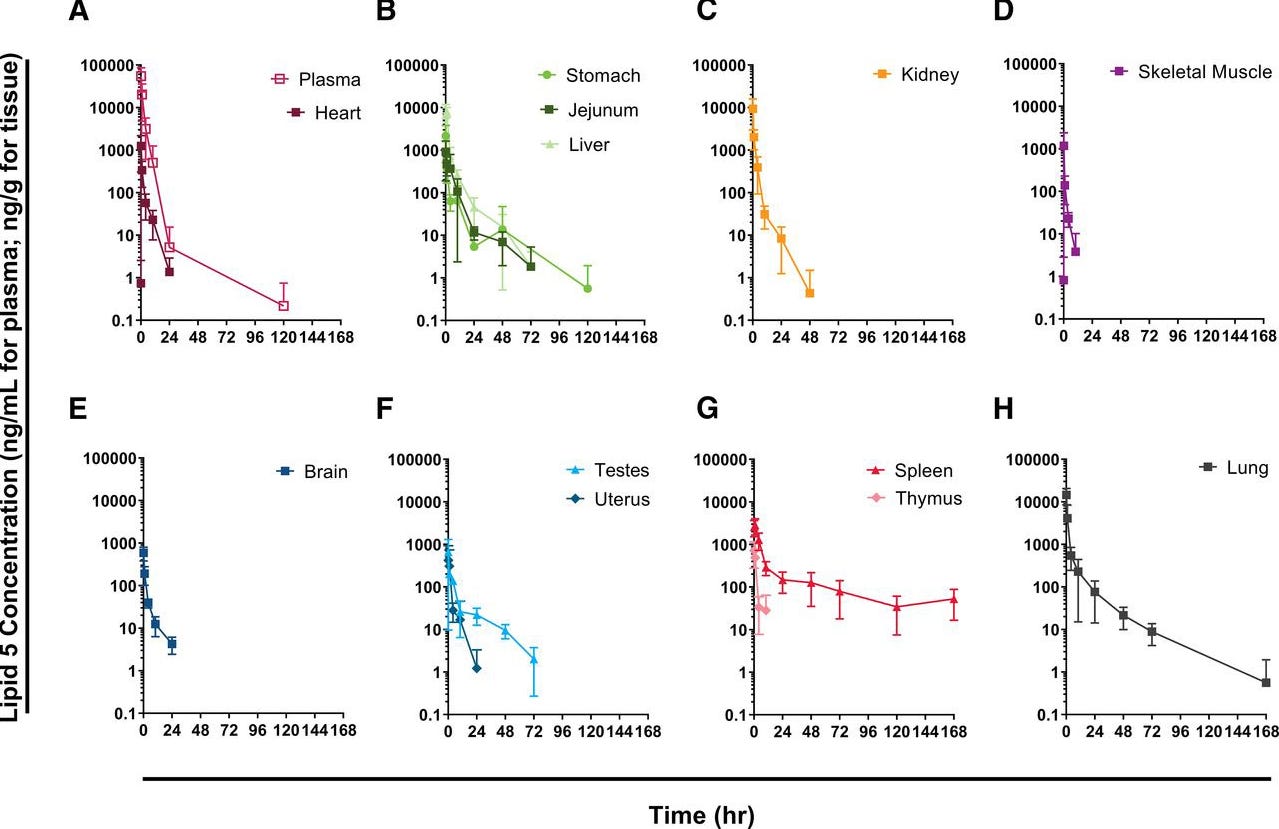
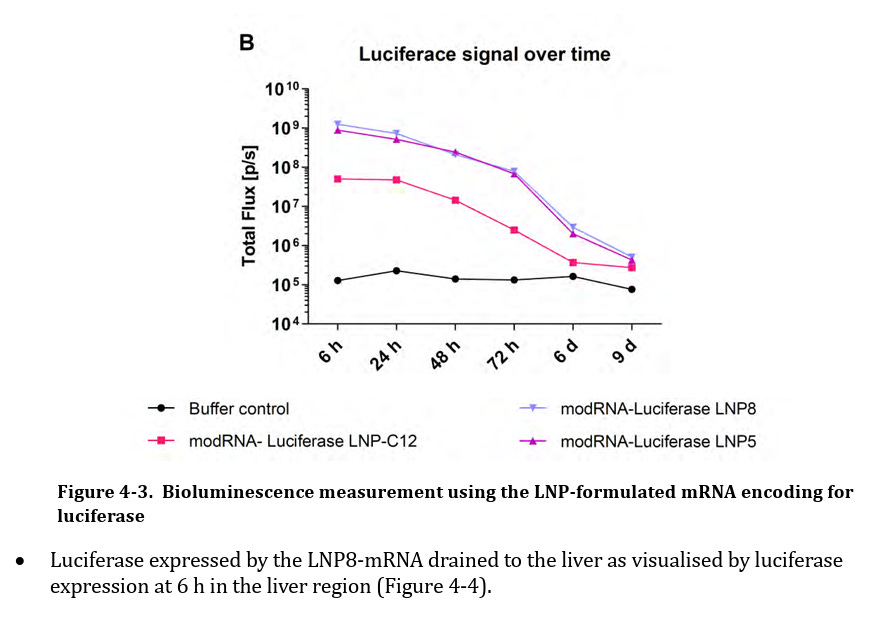
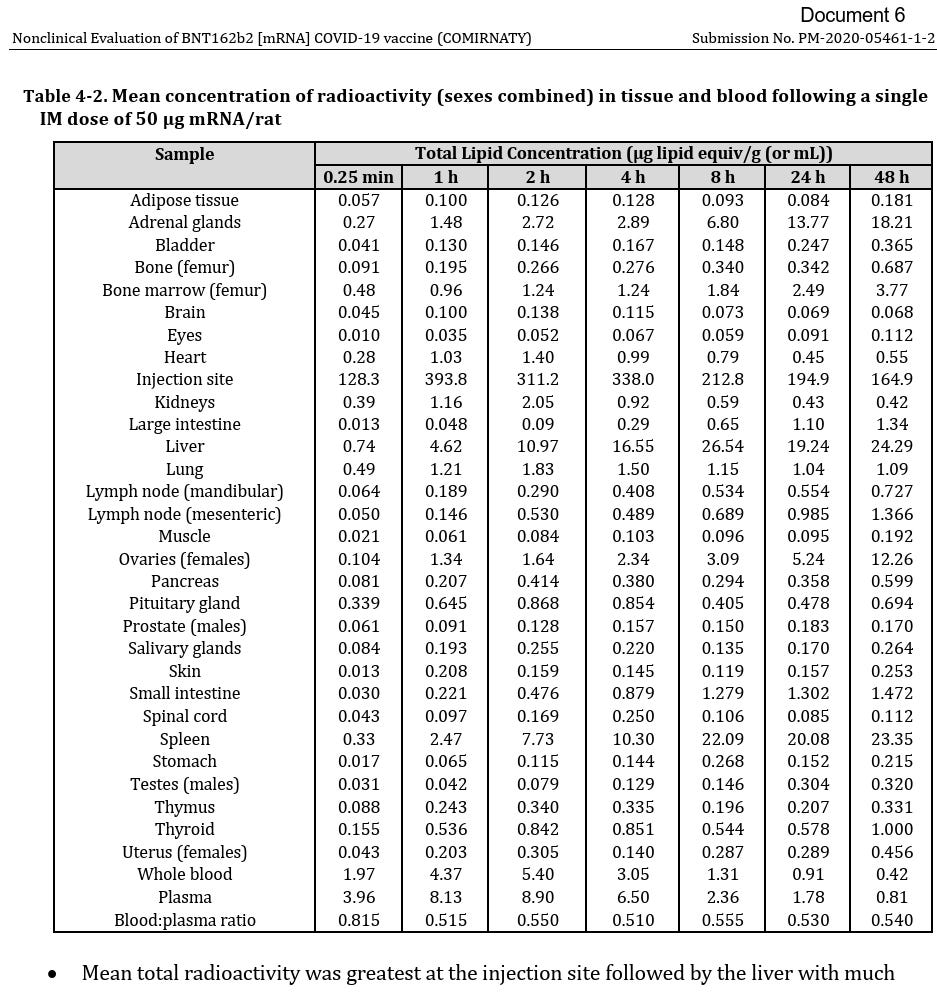



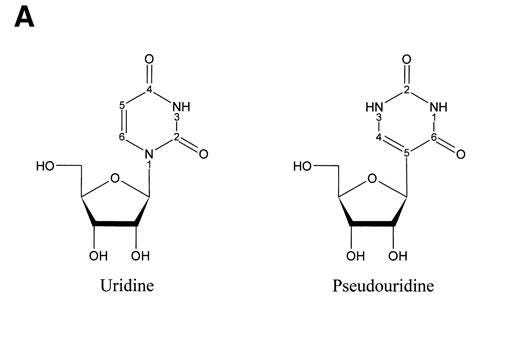
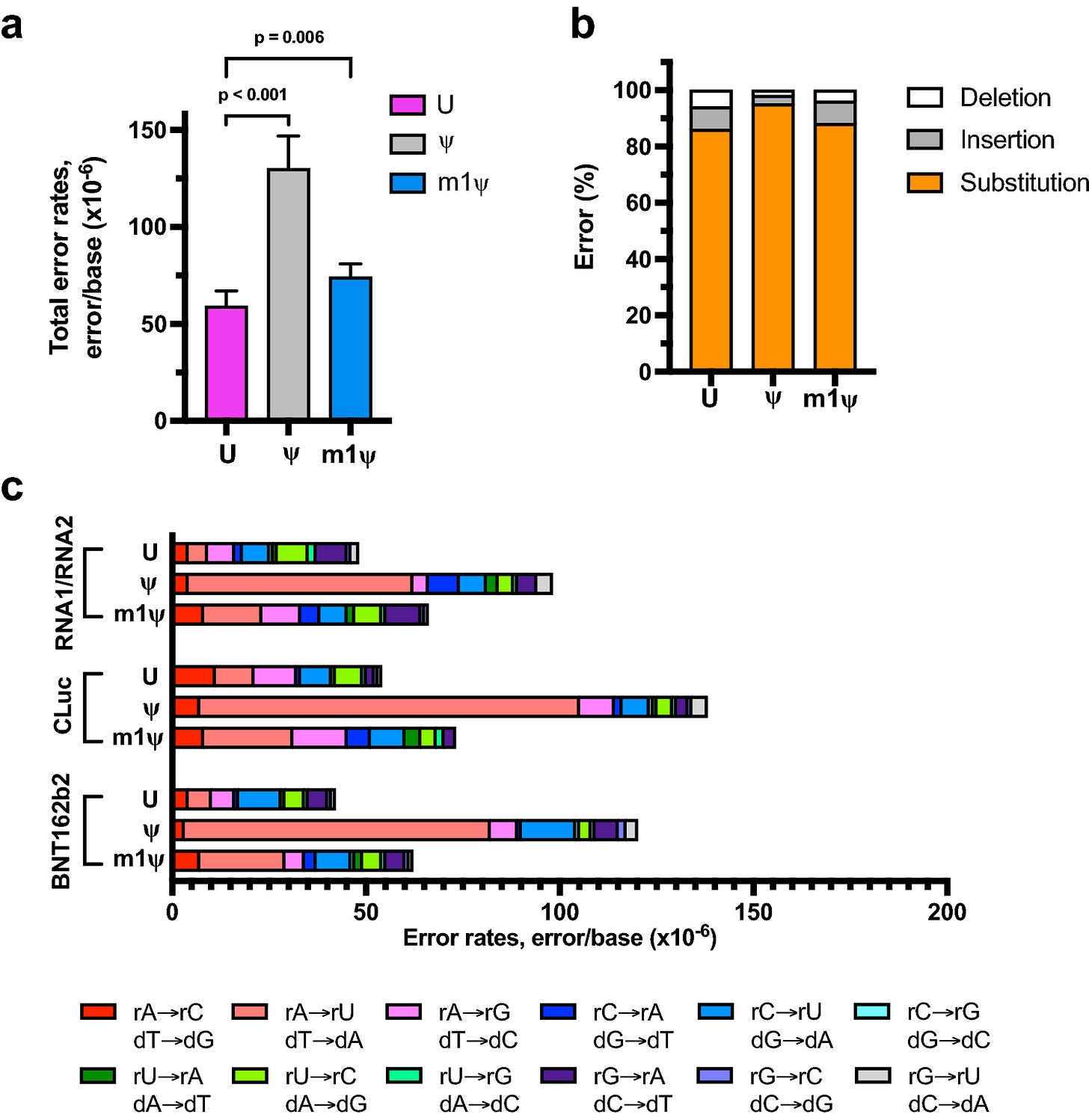
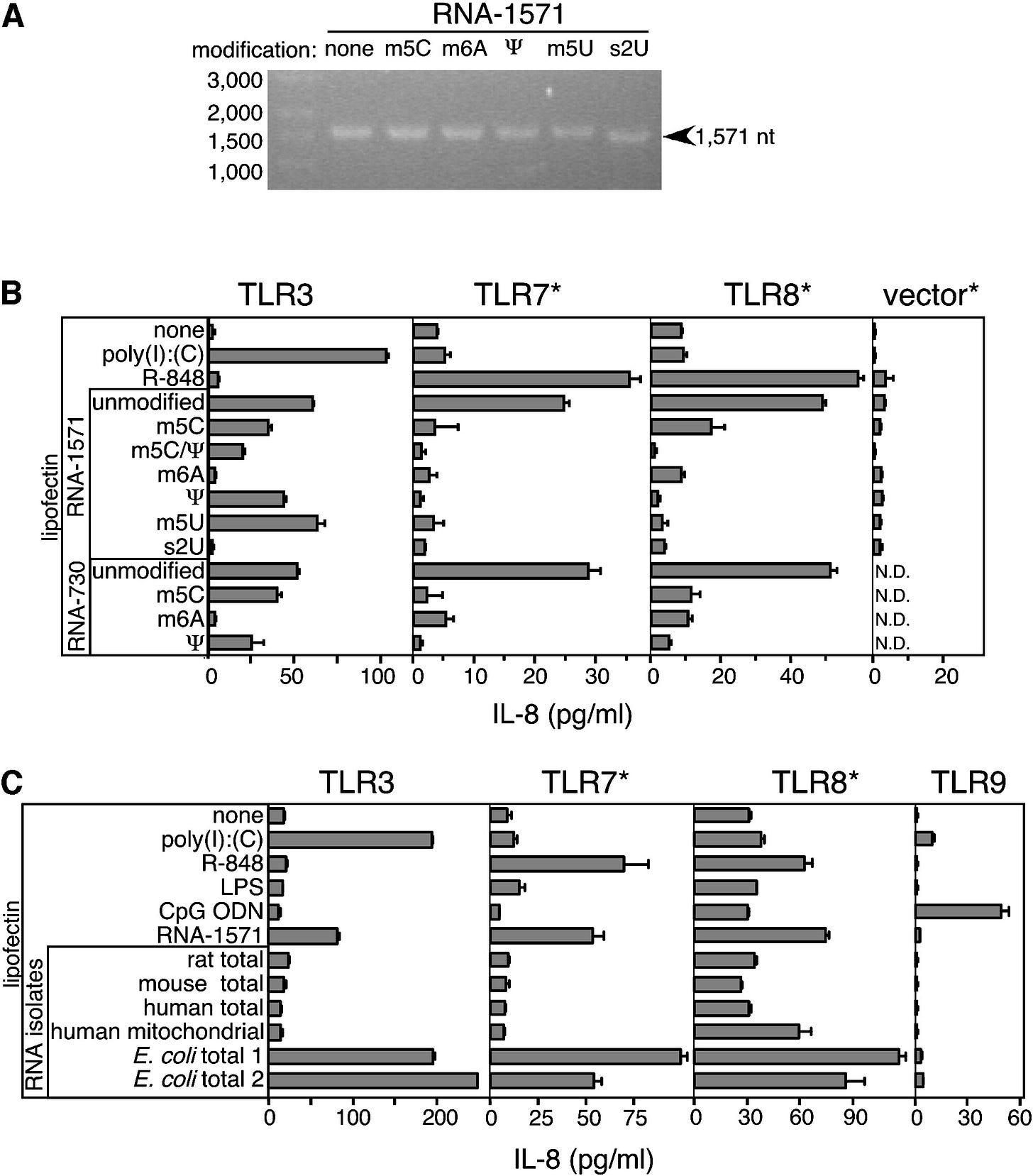


When is Substack gonna add a download to pdf 😂😂🙃
Nur ganz kurz, bevor ich das vergesse während ich den Rest lese, als du die Quelle 46 zitierst war mir eingefallen, das es in der Quelle 48 ja noch die Verteilung mittles radioaktivem Marker von Pfizer durchgeführt wurde und das in dem Summary (Seite 9 des PDF) der Studie steht, das Pfizer wusste, das die LNPs intrazellular sind und auch nicht wieder in die Circulation eintreten.