Vielleicht erinnert sich der ein oder andere noch, als ich noch auf Twitter/X war, bevor zum X Mal gecancelt, lag ich mit dem Hauptautoren der Publikation, die ich hier diskutieren will, im Clinch:
Wir vergleichen hier mal das Preprint und die nun - nach fast 3 Jahren - peer reviewed erschienene Nature-Publikation. Wir beschäftigen uns ein wenig detaillierter damit, dass, wer auch immer das Peer Review machte, zwar schon einige kritische Punkte aufgriff aber dennoch nicht den Kernbeschiss hier begriff. Ich werde mich an einer älteren Version versus Naturepublikation abarbeiten, da ich zu dem Zeitpunkt, nur diese Grafik zur Verfügung hatte. Ich habe dieses Datum (2022) gewählt, weil mir dieser Preprint kurz nach seiner Veröffentlichung aufgefallen ist und mir klar wurde, dass der Autor nur halbgare Scheinargumente und unsinnige Antworten auf meine Frage, wie man den Kern ausschließen könne, liefert und mich als jemanden behandelt, der seine Arbeit nicht gelesen oder nicht verstanden hat.
Es ging um folgenden Schlüsselsatz (der witzigerweise in der Naturepublikation verschwunden ist):
“As the vaccine mRNA is not expected to enter the nucleus, there should be a specific cytoplasmic mechanism causing the extension of vaccine poly(A) tails after delivery to macrophages.”
So, so, ihr haltet es also für ausgeschlossen, obwohl eure eigenen Daten euch Lügen strafen?:
SARS-CoV-2 mRNA vaccine is re-adenylated in vivo, enhancing antigen production and immune response (Preprint)
Fällt euch eigentlich der Skalierungswitz auf, der bereits auf akuten “Wir passen das Ergebnis unserem Text an”-Beschiss hinweist?:
0 bis 4: 4
4 bis 12: 8
12 bis 24: 12
24 bis 48: 12
48 bis 72: 24
72 bis 96: 24
https://www.nature.com/articles/s41586-025-08842-1 (Nature)
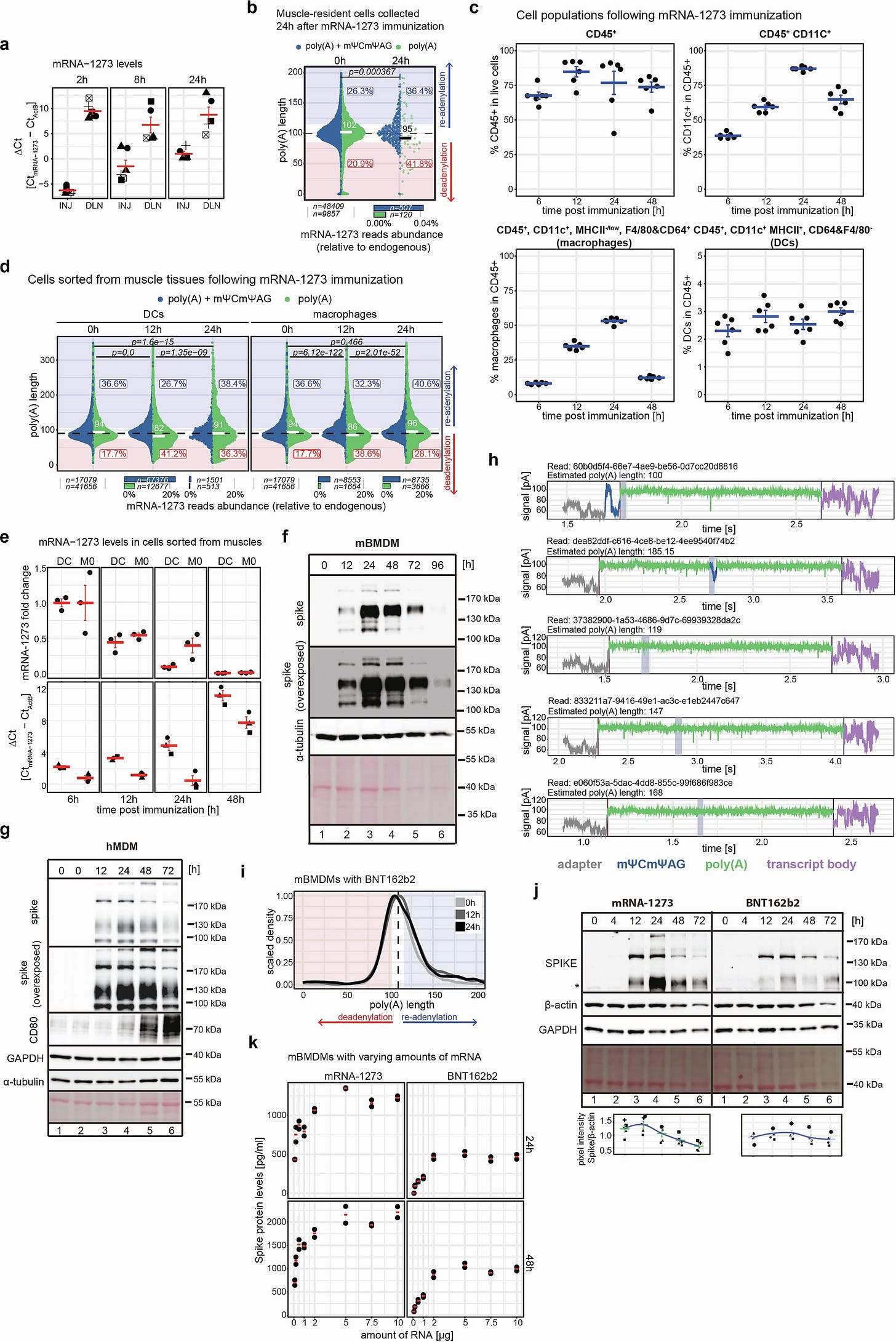
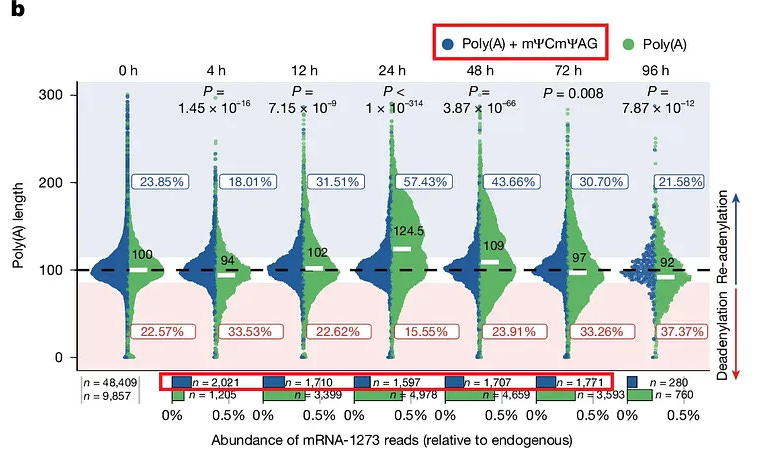
Wir fangen mit Figure 2. (Nature) versus Figure 3 (ursprüngliches Preprint) an:
Uns interessieren die Figuren e und f im Preprint versus b) und c) in der Nature. Übrigens: Interessanterweise widersprechen hier die Grafiken klar denen im Preprint und auch die Reihenfolgen der Figuren:
”e-f, mRNA-1273 poly(A) lengths distribution in BMDMs (e) and DCs (f) treated with vaccine for up to 72 h (DCs) or 96 h (BMDMs). Average distributions for 2 (DCs) or 3 (BMDMs) replicates are shown. Reads are divided into ones with (blue) and without (red) mΨCmΨAG sequence. Lower panel shows the relative abundance of each group. P.values calculated using Wilcoxon test, p. value adjustment with Benjamini-Hochberg method.” (Preprint)
”b,c, mRNA-1273 poly(A) length distribution in mBMDMs (b) or hMDMs (c) incubated with mRNA-1273 LNPs for up to 96 h.” (Nature)
Seht ihr das was ich sehe?: Während die gesamte Readenylierung zwar über die jeweiligen Zeitpunkte der BMDMs und hMDMs (Nature) abnimmt, nimmt dennoch zeitgleich das TCTAG (m1ΨCm1ΨAG)+ Poly-A zu. Was wurde aus den DCs, wo man es noch klarer sieht? Mal gucken, da ich selber gerade noch aktiv in der Nature-Studie hänge, ob ich die irgendwo noch finde:
Extended Data Fig. 3: mRNA-1273 poly(A) tail extension in vivo and in macrophages.
“d, cDNA-seq measured mRNA-1273 poly(A) lengths distribution in muscle-resident DCs (CD45+, I-A/I-E (MHCII)high, CD11c+, F4/80− and CD64−) and macrophages (CD45+, CD11c+, F4/80+, CD64+), sorted from vaccine injection sites 12 h and 24 h after administration. e, Levels of mRNA-1273 in muscle-resident macrophages and DCs, sorted out from injection sites 6 h, 12 h, 24 h and 48 h after mRNA-1273 administration.”
Noch ein kleiner Funfact am Rande, dass der Autor und sein Team sogar die Transfektion von BMDMs (knochenmarkabstammende (derived)) Makrophagen einräumt.
Ganz schön verwirrend, nicht wahr? Und warum vermengten sie hier noch die DCs und Makrophagen so, dass der eigentliche Hammer nicht mehr so heftig auffällt (siehe ursprüngliche Grafik DCs versus Makrophagen aus dem Preprint)? Ich entwirre das Ganze mal ein wenig und stelle die wichtigen Figuren direkt untereinander:
Diesmal starten wir mit der Naturepublikation
Versus Preprint (ältere Version):
Wie ihr also wunderschön sehen könnt, waren meine Pfeile in der Originalfigur bereits korrekt gesetzt und mein Hinweis, dass das TCTAG+POLY-A-Motiv zunahm.
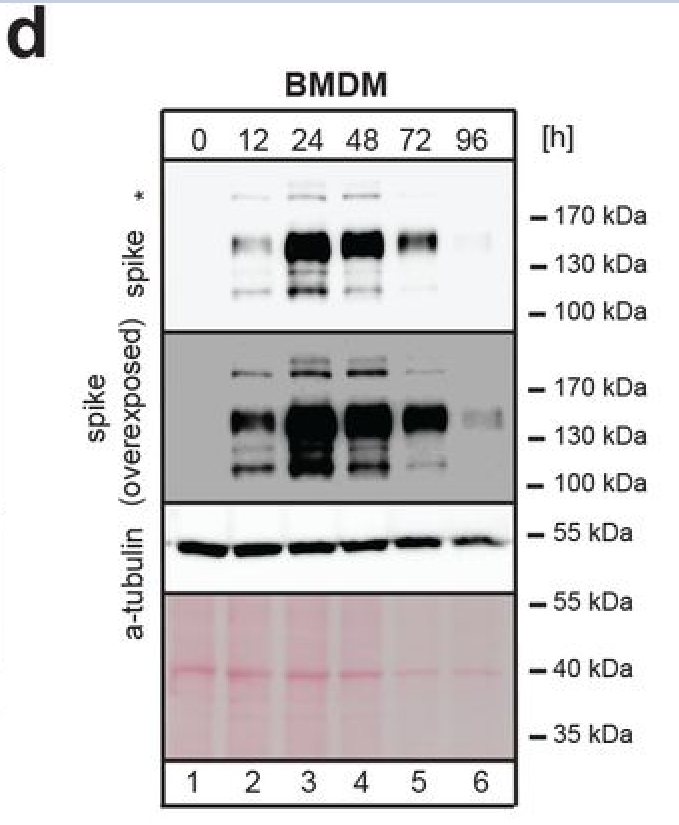
Wir klären gleich noch das Hauptproblem und die dummdreiste Lüge der Autoren. Aber zunächst zerpflücken wir noch folgende Sätze, in dem wir uns dazu die Western Blots anschauen:
“Knowledge about in vivo and in-cell metabolism of mRNA vaccines is extremely limited and is largely based on extrapolation from experiments in established cell lines with generic mRNAs. The paradigm extrapolated from data obtained on endogenous transcripts (or reporters) asserts that the stability of therapeutic mRNAs is determined by the rate of poly(A) tail removal (deadenylation). Notably, almost nothing is known regarding the metabolism of vaccines’ poly(A) tails.”
(…)
“Notably, in the past, the action of the endogenous polyadenylation machinery on mRNA therapeutics has not been reported or even considered.”
(Aus dem Preprint)
Also liebe Autoren: Was auch immer ihr da meint, gesehen zu haben: ENDOGEN war das bestimmt nicht! 3 verschiedene Proteinbänder ist nicht endogen! Ich versuche es verständlich zu erklären:
Mindestens drei verschiedene Isoformen oder Modifikationen eines oder mehrerer Proteine liegen vor. Diese entstehen ‼nicht durch reine endogene Konstitution‼ der Zelle. Folgerichtig ist die Interpretation der Autoren, dass die beobachtete Polyadenylierungsaktivität "endogen" sei, aufgrund der Western Blot-Daten nicht haltbar. Das Auftreten von drei unterschiedlichen Proteinbändern weist auf nicht-physiologische Bedingungen hin, wie sie bei exogener mRNA-Transfektion, induzierter Überexpression oder massiver Zellstressantwort auftreten. Eine rein endogene Aktivität würde typischerweise ein einziges, klar definiertes Band erzeugen. Dieser Schwachsinn wurde auch in der Nature-Publikation übernommen.Soviel also zur Endogenexpression.
Doch zurück zum eigentliche Problem und “TENT5”:
Die Autoren behaupten also, dass ausschließlich TENT5 für die Readenylierung (zytosolisch) in Frage käme. Im Preprint waren sie sogar noch dummdreister und schrieben, wie bereits Oben angemerkt:
“As the vaccine mRNA is not expected to enter the nucleus, there should be a specific cytoplasmic mechanism causing the extension of vaccine poly(A) tails after delivery to macrophages.”
Was eine Übernahme eines Narrativs fernab von wissenschaftlich falsifizierter oder verifizierter Realität ist.
Ganz im Gegenteil: Es gab ja noch folgendes Preprint:
Nuclear translocation of spike mRNA and protein is a novel pathogenic feature of SARS-CoV-2
“In conclusion, the SARS-CoV-2 S protein has a functional pat7 NLS “PRRARSV”, that results in one out of four S proteins translocating into the nucleus in infected cells. S Protein appears to shuttle S mRNA (possibly the genome) into the nucleus as well. Thus, the NLS of the S protein may contribute to the evasion of the host immune response and is a novel pathogenic feature of SARS-CoV-2.”
Üble Geschichte, aber ich will nicht zu sehr abschweifen.
Das eigentliche Problem, wieso dieser Satz der Autoren bereits völliger Schwachsinn ist, ergibt sich aus folgender Figur:
So, so, keine TENT4a - l Aktivität? Keine TENT2 Aktivität genau zu den Zeitpunkten, an denen das TCTAG-Motiv mit POLY-A-Schwanz raufgeht?! Ja ne, ist klar! Wie habt ihr den Nukleus ausgeklammert. Btw: Wo genau sehe ich denn überhaupt TENT4c -l, liebe Autoren?! Habt ihr sicherlich nur, äh, “versehentlich”™ vergessen zu zeigen?Aber hey: Laut dem Autor habe ich ja keine Ahnung, weil TENT4 gar nicht in Frage kommen kann…
Seltsam, wie es zeitlich absolut perfekt mit den Expressionsprofilen von TENT2, 4b und a) korreliert, nicht wahr? Oder vllt. ja doch nicht?
Schauen wir uns das mal noch im Detail an:
Und jetzt genau mitdenken, warum dieser “Nein, nein, nein nicht Tent4”-”Wissenschaftler”™ logischerweise auch TENT5 expressiert sah:
Recycling of uridylated mRNAs in starfish embryos
"Non-A residues are observed more frequently near the 5’ end of poly(A) tails in mouse GV oocytes, as shown using poly(A) inclusive RNA isoform sequencing (PAIso−Seq) (47). Similarly, approximately 30% of the non-A residues were dominantly distributed in the 5’ region of the non-canonical poly(A) tails of the mRNA of the ribosomal protein in starfish embryos (Figure 2C and Figure 3C), suggesting that both animals may utilise similar modification systems. Although the enzymes involved in the 5’ end modifications in mouse oocytes remain to be determined, mammalian TENT4A/4B (48) can mediate mixed tailing of adenylation and guanylation to stabilise mRNAs. Thus, the starfish homologue of TENT4 may generate the 5’ modifications in the non-canonical poly(A) tails. ‼ In addition, TENT5 families mediate the cytoplasmic polyadenylation of collagen mRNAs required for bone mineralisation (49) and immunoglobulin mRNAs (50) in mammals. ‼ These families may also participate in the non-canonical polyadenylation, exhibiting a high percentage of A residues, especially at the 3’ region in the starfish cytoplasm."
Wie witzig! Ausgerechnet bei Uridin?! → Wie war das mit modifizierten Urdin( N1-Methyl-Pseudouridin)?! Oh: Und TENT5 würde also vorrangig Immunoglobuline adenylieren?
“No?! Yes! OHHH?!”
Mach Sachen! Warum sollte man, bei einer Transfektion mit einem mod-Virus-RNA-Strang immunoglobuline Adenylierungen und damit logischerweise auch eine gesteigerte TENT5-Aktivität erwarten, die aber eben nicht eine Steigerung von TCTAG+Poly-A erklären kann?! Übrigens spielt TENT5a hauptsächlich bei Collagenadenylierung und Knochenmarksformation eine entscheidende Rolle. Sollte man sich bei den gezeigten Counts vllt. ein wenig Sorgen machen? Waren ja “nur” Knochenmarkmakrophagen.
Dies schrieb unser Hauptautor übrigens selber in folgender Publikation:
TENT5 cytoplasmic noncanonical poly(A) polymerases regulate the innate immune response in animals
“Mammalian genomes encode four TENT5 proteins (TENT5A to TENT5D, also known as FAM46A to FAM46D), all of which are active ncPAPs (21, 25). Functional analysis revealed that TENT5C is a multiple myeloma growth suppressor (21, 22). In multiple myeloma cell lines, TENT5C polyadenylates and stabilizes numerous mRNAs that encode secreted proteins (21). TENT5C also plays a crucial role in the regulation of immunoglobulin expression and the humoral immune response in mice through polyadenylation of mRNAs that encode immunoglobulins (23, 24). TENT5A polyadenylates mRNAs encoding collagens and is thus required for the proper bone formation (26). However, TENT5 proteins are differentially expressed in mammalian tissues and organs with potential redundancy, which makes study of their functions difficult.”
Na was interessiert mich mein Geschwätz von gestern, wenn ich in meiner eigenen kleinen Perspektivbegrenzung “etwas Neues”™ entdecke und das praktischerweise mit einer absolut seltendämlichen These verbinden kann, die keinerlei Sinn ergibt und zudem Kritikern direkt (aggressiv) ankeifen kann und behaupten kann, diese hätten keine berechtigte Frage gestellt:
Und jetzt bitte anschnallen:
Warum ist TENT4, 2 und die Differenzierungen so wichtig? Ist das nicht einfach nur Rechthaberei? NEIN! Ist es nicht:
Terminal nucleotidyl transferases (TENTs) in mammalian RNA metabolism
“(b) TENT4A, also known as LAK-1, PAPD7, POLK, POLS, TRF4 and TRF4-1
“TENT4A is a human orthologue of the yeast Trf4p protein. Trf4p is a key subunit of the so-called TRAMP complex, within which it specifies mRNAs for surveillance and turnover by the nuclear exosome 3′–5′ ribonuclease complex [45,46], reviewed in [47,48]. However, TENT4A has not been identified as a component of the human TRAMP complex [49].”
“(c) TENT4B, also known as GLD4, PAPD5, TUT3 and TRF4-2
TENT4B is another human orthologue of the yeast Trf4p protein. Initially, TENT4B had been suggested to be involved in uridylation-mediated turnover of replication-dependent histone mRNAs in the cytoplasm [56]. However, this result is controversial and has been challenged by other studies [57,58]. Essentially, human TENT4B has a strong preference for ATP incorporation (in the apparent preference order ATP ≫ GTP > CTP ∼ UTP) with a variety of RNA substrates tested in vitro, ranging from oligo(A) and oligo(U) to different tRNAs as well as the 3′ UTR of histone mRNAs [57]. Additionally, examination of TENT4B-EGFP-expressing cells, as well as immunofluorescence analysis, demonstrated its nuclear localization with nucleolar accumulation [49,57]. These findings suggest that TENT4B function in the mammalian nucleus may be similar to that of the TRAMP complex in yeast [59]. Furthermore, and in contrast to yeast Trf4p which requires the Air2p zinc knuckle protein [45,60], human TENT4B does not require a protein cofactor for its polyadenylation activity, thus demonstrating its mechanistical distinction from its yeast counterpart [57].”
a) Nur TENT 4 kann gemischte AG-Poly-A-Schwänze anhängen.
und
b) Ist es der entscheidende Unterschied zwischen passierte das im Zytosol oder Nukleus?!
Übrigens liefert das Naturepaper auch Hinweise auf micro-RNA-Involvierung, was ebenfalls aus diesem wunderschönen Royalsocietyreview hervorgeht:
Doch genau darum - wenn man sich die Count-Grautöne im Naturepaper anschaut und die verbohrte, um nicht von lächerlich zu reden, Eindimensionalität, mit der der Hauptautor und sein Team sich auf TENT5 versteiften - möchte man nur noch schreien.
Siehe auch:
https://www.ncbi.nlm.nih.gov/gene?Db=gene&Cmd=DetailsSearch&Term=11044
Ich finde es so irre witzig, dass ich seit 2022 recht behalten sollte und auch zeitgleich irre tragisch, dass so ein Musterexemplar aus purer Zirkellogik überhaupt das Review überstehen konnte. Denn in der Nature werden ja nun auch final die Prozentzahlen angegeben. Und bereits daran wird sichtbar, dass die gesamten Schlussfolgerungen des Hauptautoren und seines Teams wissenschaftlich nicht haltbar sind.
Mit einem kräfitigen Narf,
euer Bullshit-Paper-Terminator, äh, Genervter Bürger.












So the mea culpas are coming, but even with that, the FULL extent of what happens IN VIVO with these poly A tails has not been fully elucidated.
Great work again
Ein Hinweis auf sehr niedrigem fachlichem Niveau: Heute ebenfalls in meinem Posteingang war ein Post von Peter McCullough über dasselbe Nature-Papier vom 16. April.
https://www.thefocalpoints.com/p/breaking-endogenous-re-adenylation?publication_id=1119676&post_id=162206833&isFreemail=true&r=1pqqxt&triedRedirect=true
Wenn ich seine Überschrift richtig verstehe, scheint er das "endogen" so zu akzeptieren.
Vermutlich ist das nicht Neues, was ich eben gerade schrieb.
Ich bitte die quasi Doppelzusendung zu entschuldigen.