Downstream Teil 5
Meine erste Folie, mit der ich zufrieden bin und die mir zeitgleich Angst macht.
Downstream! - Wer braucht das schon, wenn er von mutierten Genen schwurbeln kann?
Soweit so schick also erstmal. Verzeiht mir die kleine Anspielung auf Dirk Fasshauer (X ehemals Twitter), die keineswegs die Arbeiten von Kevin McKernan (Anandamide; @kevinmckernan) , David J. Speicher und vielen weiteren großartigen Wissenschaftlern schmälern soll.
Auf Dirk komme ich noch in einem der letzten Teile dieses Substack kurz zu sprechen, wenn ich das Ganze auf die modRNA-Transfektionsbrühen runterbrechen werde. P.s.: Aus irgendeinem Grund hatte ich mir das Ganze wesentlich einfacher vorgestellt, weil ja das Grundprinzip in meinem Kopf längst verankert und begriffen ist.
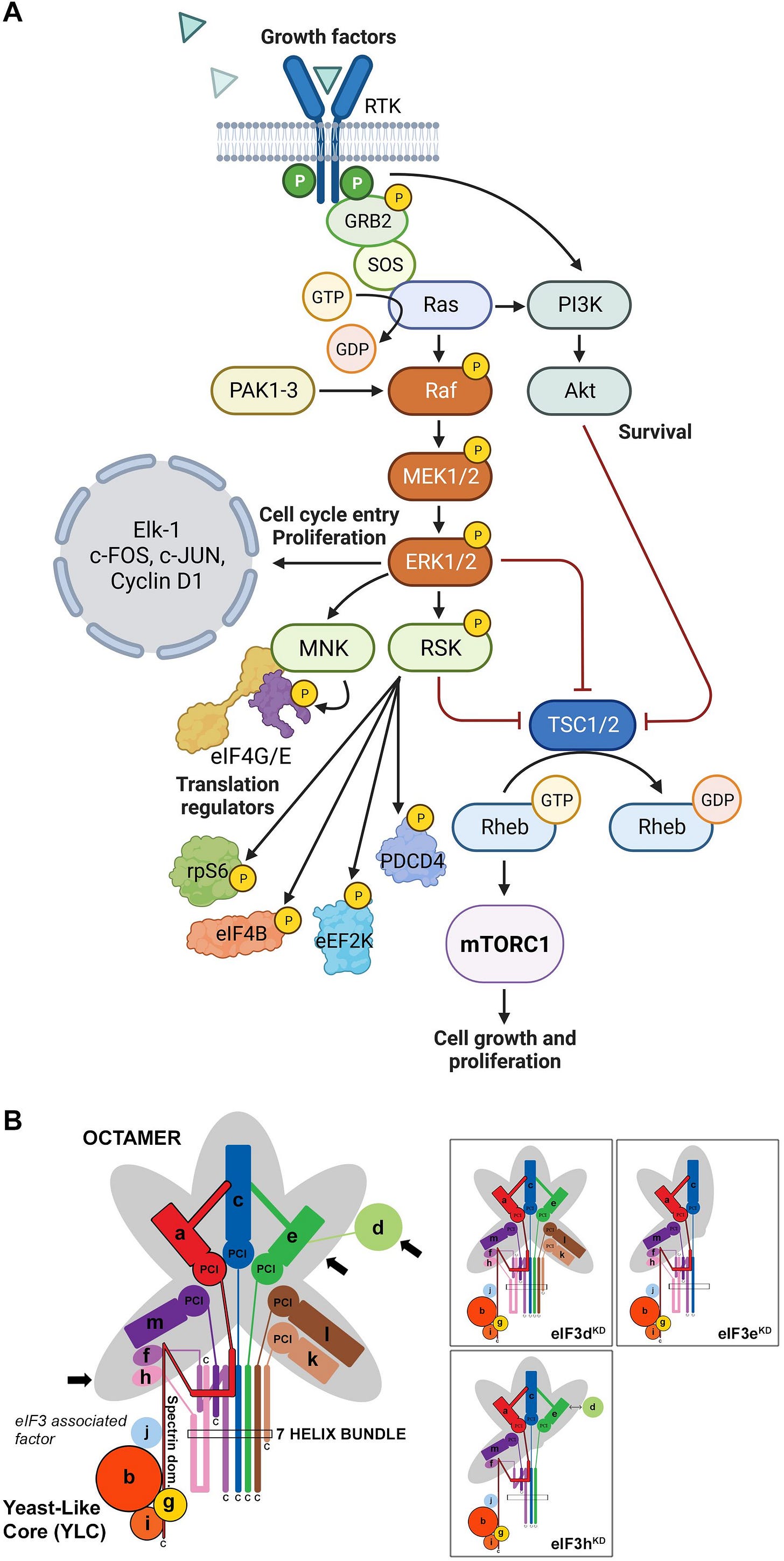
Wir haben jetzt den Einfluss der großen 4 Signalkaskaden auf den PtdIns-Zyklus geklärt (rote Pfeile) und, dass sie untereinander ein komplexes Netzwerk aus gegenseitigen Check - und Balance Crosstalks bilden (grüne Pfeile), die spezifische Downstream-Effekte und regulatorische Prozesse auslösen werden. [p.s.: Hatte 2 blöde Tippfehler in der Folie, die ich mittlerweile ausgebügelt habe.]
Und noch eine kurze Voraberklärung, die vllt. noch nicht so ganz verstanden ist:
Wir müssen Schritt für Schritt der LNPs als engmaschig regulierte Prozesse betrachten. Das heißt es ist ein Riesenunterschied zwischen dem endosomalen Escape (welcher vorrangig von dem PtdIns-Zyklus reguliert wird) und der Transfektion als solche, wo das LNP sich in die Plasmamembran schweißt und damit ein endozytoseähnlicher Prozess ausgelöst wird, welcher erst einen endosomalen Escape forcieren soll. Das ist übrigens der wirklich widerliche Part, wieso ich auch so stinkig auf alle bin, die behaupten, ohne die modRNA kein Schaden: Die Transfektion ist mit dem Kontakt an der Membran bereits Realität. Und auch der Zellschaden. Und die Transfektion - so zumindest mein Verständnis - wird nicht rezeptorabhängig stattfinden, was zu völlig konfusen Reaktionen insgesamt führen wird, da die LNPs randomisiert an unterschiedlichen Stellen (3D-Struktur bedenken) auf der Membran reinkleistern und das in völlig unterschiedlichen Schwingungsmomenten der einzelnen Lipide der Doppelschicht und unterschiedlichen Prozessen(Metabolismen, Aufnahme, Abgabe, etc), die gerade ablaufen. Zeit, Raum, Distanz. Rein hypothetisch müsste ich sogar dasselbe Spiel für den endosomalen Escape noch einmal spielen, da auch dieser engmaschig reguliert wird via PtdIns-Zyklus und davor - und danachgeschalteten Signalen.
Der Part, der also jetzt kommt, gruselt mich offen gestanden am Allermeisten, weil das bisher Diskutierte dagegen ein Kindegeburtstag sein wird. Das ist auch der Grund, wieso ich dieses Substack in “kleine Häppchen” aufteilen musste, da dieser Part der längste sein wird. Ich muss, noch bevor ich auf die Signaltransduktion eingehen kann, zunächst ein halbwegs grundlegendes Verständnis dafür entwickeln, wie eine Translation eingeleitet wird, sprich welche Komponenten involviert sind. s(mall)i(nterfering)RNA und miRNA werde ich später im nächsten Teil nur kurz anreißen. Wer sich für microRNA - die kleinen Powerriesen - noch näher interessiert:
Schauen wir uns in diesem Teil die eukaryotischen Initiationsfaktoren an, welche zwingend an der Translation beteiligt sein müssen. Da ich auf diesen Aspekt bis dato noch so gut wie gar nicht einging, klären wir zunächst einmal, was wir unter diesen Faktoren verstehen und wie sie funktionieren:
A mechanistic overview of translation initiation in eukaryotes
“Die Initiierung der Translation in Eukaryonten ist ein komplexer und stark regulierter Prozess, der die Wirkung von mindestens 12 Proteinfaktoren erfordert. Der Weg zeichnet sich durch die Bildung eines Prä-Initiationskomplexes aus, der das 5′-Ende der mRNA rekrutiert und es abtastet, um das Startcodon zu lokalisieren. In den letzten zehn Jahren hat eine Kombination aus Genetik, Biochemie und Strukturstudien begonnen, die molekularen Schlüsselereignisse in dieser kritischen Phase der Genexpression zu beleuchten. Im Folgenden wird unser derzeitiges Verständnis der eukaryotischen Translationsinitiation skizziert und es werden wichtige offene Fragen diskutiert.”
Ich liebe die Worte “stark regulierter Prozess”, ihr auch?
“Abbildung 1 Schematische Darstellung des Translationsinitiationsweges in Eukaryonten. Die Initiation beginnt mit der Bildung des ternären Komplexes (TC), der eIF2-GTP und die Initiator-tRNA enthält (1). Der ternäre” [drei Komponenten gebunden] “Komplex wird mit Hilfe der eIFs 1, 1A, 3 und 5 an die 40S-Untereinheit rekrutiert und bildet den PIC (2). In der Zwischenzeit wird die mRNA durch die eIF4-Faktoren und das PABP gebunden, um ein aktiviertes mRNP (3a) zu bilden, das dann zum PIC rekrutiert wird (3b). Sobald es am 5′-Ende der mRNA gebunden ist, sucht das PIC nach dem Startcodon (AUG) (4). Die Erkennung des Startcodons löst die Freisetzung von eIF1 und die Umwandlung von eIF2 in seinen GDP-gebundenen Zustand aus, wodurch der Scanning-Prozess gestoppt wird (5). eIF2-GDP und eIF5 dissoziieren und machen den Weg für eIF5B frei, um die Verbindung der 60S-Untereinheit zu vermitteln (6). Nach dem Zusammenschluss der Untereinheiten erfolgt die GTP-Hydrolyse durch eIF5B und die Dissoziation des Faktors zur Bildung des 80S-Initiationskomplexes (IC) (7).”
Ich denke, wenn man sich hier ein wenig Zeit lässt und der Folie folgt, versteht man das grundlegende Prinzip.
Eukaryotic translation initiation factors as promising targets in cancer therapy (Denkt noch jemand an tolle Titel wie “mRNA vaccine shows promise in autoimmunity” 🤡)
“Die Regulierung der Translation von Boten-RNA (mRNA) in eukaryontischen Zellen ist entscheidend für die Genexpression und findet hauptsächlich in der Initiationsphase statt, die hauptsächlich durch eukaryontische Initiationsfaktoren (eIFs) reguliert wird. eIFs sind für die Translation von mRNA von grundlegender Bedeutung und fungieren als solche als primäre Ziele verschiedener Signalwege zur Regulierung der Genexpression. Eine fehlregulierte mRNA-Expression ist ein häufiges Merkmal der Tumorentstehung, und die abnorme Aktivität von eIF-Komplexen, die durch vorgelagerte Signalwege ausgelöst wird, ist in vielen Tumoren nachweisbar. Dies führt zu einer selektiven Translation von mRNA, die für Proteine kodiert, die an der Tumorentstehung, der Metastasierung oder der Resistenz gegen Krebsmedikamente beteiligt sind, und macht eIFs zu einem vielversprechenden therapeutischen Ziel für verschiedene Arten von Krebs. Hier wird kurz unser derzeitiges Verständnis der Biologie der eIFs umrissen, wobei wir uns hauptsächlich auf die Auswirkungen verschiedener Signalwege auf ihre Funktionen konzentrieren und ihren Beitrag zur Auslösung und zum Fortschreiten des Tumorwachstums diskutieren. Außerdem wird ein Überblick über die Fortschritte bei der Entwicklung von Wirkstoffen gegeben, die auf die Komponenten der Translationsmaschinerie zur Krebsbehandlung abzielen.”
Doch dies beantwortet uns ja weder die Frage, wie die eIF-Familie (12 Stück sind momentan bekannt) überhaupt reguliert wird im Sinne von aktiviert, noch wie die Ribosome angesteuert werden. Und nur als kleiner Randfunfact: Es gibt nicht mal eine klare Homologie der Ribosome:
Patterns of ribosomal protein expression specify normal and malignant human cells
„Ribosomen sind hoch konservierte molekulare Maschinen, deren Kernzusammensetzung traditionell als unveränderlich angesehen wurde. Jüngste Studien haben jedoch verblüffende Unterschiede in der Expression einiger ribosomaler Proteine (RP) in verschiedenen Geweben und sehr spezifische Auswirkungen auf die Übersetzung einzelner mRNAs festgestellt.”
“Unsere Studie zeigt eine unerwartete Plastizität der RP-Expression in normalen und bösartigen menschlichen Zelltypen und bietet eine Grundlage für die künftige Charakterisierung zellulärer Verhaltensweisen, die durch spezifische RP gesteuert werden.”
Pah! Das Paper ist doch von 2016. Hat sich bestimmt was daran geändert! Denn schließlich folgen wir “die Wissenschaft”™! - Und die lässt sich ja generell in ihrer Erkenntnisgewinnung mit genügend Müllstudien, PR und “die Experten”™ beschleunigen!
„Es gibt Hinweise darauf, dass einige proteincodierende ribosomale Gene bei verschiedenen Arten, insbesondere bei Wirbeltieren, eine unterschiedliche Rolle spielen, da einige von ihnen an einer Reihe von genetischen Krankheiten beteiligt sind. Auf der Grundlage umfangreicher Sequenzvergleiche und systematischer Kuratierung erstellen wir einen Referenzsatz für ribosomale Proteine (RP) in elf Wirbeltierarten und quantifizieren ihre Sequenzerhaltungsgrade. Darüber hinaus korrelieren wir ihre koordinierten Genexpressionsmuster in bis zu 33 Geweben und bewerten die außergewöhnliche Rolle von Paralogen bei der Gewebespezifität.“
Da der Weg bis hier hin doch sehr trocken war und ich kaum Bezug auf die modRNA-Genplörren nahm, schiebe ich mal fix ein paar “praktische” Sneakpreviews rein, Demnächst auch in Ihrem Kino und in jeder gut sortierten Impfothek:
“Im Gegensatz zu der durch die Chemotherapie mit dem Platin(II)-Komplex verursachten Zerstörung wurde eine fortschreitende Zerstörung des 18S-Ribosoms auch noch 6 Monate nach der Impfung festgestellt.“
„Der Patient Nr. 5 verschlechterte sich plötzlich nach der zweiten Impfung und zeigte Symptome eines leichten Hirninfarkts, wie Gedächtnisverlust, Schwindel, Lähmungen, Schluckstörungen, Dysphonie usw.“
“Die geimpften Personen wiesen eine starke Herabregulierung der ribosomalen Proteine sowie der Gene für die Immunantwort und der Transkriptions-/Translationsmaschinerie auf, die die gesamte Landschaft des angeborenen Immunsystems methodisch in Richtung Immuntoleranz modulierte, ein Merkmal des angeborenen Immungedächtnisses. Diese koordinierte Reaktion wurde durch 17 Transkriptionsfaktoren orchestriert, die bei den Impfdurchbrüchen unterschiedlich exprimiert wurden, darunter epigenetische Modulatoren von CHD1 und LMNB1 sowie mehrere Effektoren der Immunantwort, wobei sich ELF1 als einer der wichtigen Transkriptionsregulatoren der antiviralen angeborenen Immunantwort herausstellte.”
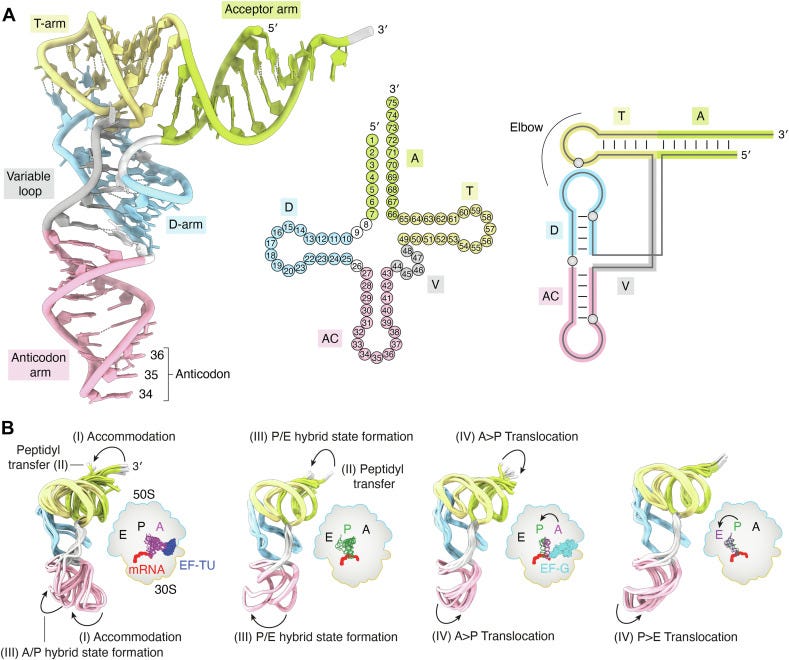
Ribosome sind also die großen Translationsmaschinerien, welche mit absolut codon-spezifischer tRNA (translationsRNA) gefüttert werden und Codon für Codon ablesen und in ein Protein umschreiben. Die Translation, Faltung und Posttranslationale Modifikationen des Proteins geschehen durch verschiedene Subprogramme, wie die eIF, miRNA, siRNA und direkte Interaktionen mit den Signalmolekülen.
The diverse structural modes of tRNA binding and recognition
“Die Anzahl der tRNA-Arten in verschiedenen Organismen schwankt zwischen 23 und 45, die in 20 Gruppen von sogenannten Iso-Akzeptoren zusammengefasst sind. Dabei handelt es sich um tRNAs, die mit derselben Aminosäure geladen sind, obwohl sie unterschiedliche Anticodons haben. Abgesehen von Methionin und Tryptophan haben alle Aminosäuren mehrere Iso-Akzeptoren.”
Und auch hier gilt mal wieder: Load matters.
Uff. Schon wieder so eine ellenlange Einleitung um jetzt endlich, endlich, endlich Vollgas zu geben:
Wir wollten uns ja die Signaltransduktion in Kontext eIF-Regulierung angucken. (Auf die Elongationsfaktoren und weitere Mechanismen verzichte ich mal besser, um es überhaupt noch begreifbar zu gestalten.) Seht es mir nach, dass ich für die Transkriptionsfaktoren einen weiteren Teil machen werde. Häppchen für Häppchen, da meine Folie unglaublich komplex ist. Wie bereits (kurz) erwähnt, stecken da ungefähr 100 - 200 Paper Basisverständnis, ein Erstsemesterbuch Zellbiologie und Kontext aktueller Papers sowie erweiterter Fragestellungen (was dann summiert mind. 1k papers macht) drin. Und ich habe wirklich versucht möglichst einfach strukturiert eine Übersicht für die forcierten Prozesse (noch bevor es zur Translation kommt und was während der Translation abgeht) zu skizzieren und damit aufzuzeigen, was für einem Wahnsinn wir gegenüberstehen.
Also: Attacke und wir starten mit… Trommelwirbel….
Allgemein
Protein Regulation in Signal Transduction
In diesem Review unter 5.3 kurz angerissen. Doch wir wollen ja näher hingucken, richtig?
JAK(1/2) / STAT(1-6)
“Wir stellen fest, dass die globalen Translationsraten in CySCs hoch sind und während der Differenzierung abnehmen, und dass der JAK/STAT-Signalweg die Translation reguliert.“
Wie immer gilt: Das Ganze ist keine Einbahnstraße und ich kann - gerade für die eIF-Regulation nur sehr wenige Beispiele bringen, weil wir von der gesamten Translations- und Proteinsyntheseregulation noch so gut wie gar nichts verstanden haben.
“Die Tyrosinphosphorylierung der Transkriptionsfaktoren Stat1 und Stat3 ist erforderlich, damit sie sich dimerisieren, in den Zellkern verlagern und die Gentranskription auslösen können. Stat1 und Stat3 werden im Kern durch die T-Zell-Protein-Tyrosin-Phosphatase (TC-PTP) dephosphoryliert und deaktiviert, was die Rückkehr beider Proteine in das Zytoplasma erleichtert. Die Proteinkinase PKR spielt durch die Modulation der Phosphorylierung des eukaryotischen Initiationsfaktors-2alpha eine wichtige Rolle bei der Translationskontrolle. Frühere Daten haben gezeigt, dass PKR über die Regulierung von Stat1 und Stat3 an der Zellsignalisierung beteiligt ist, aber die molekularen Mechanismen, die diesen Ereignissen zugrunde liegen, sind noch nicht bekannt. Unter Verwendung von PKR(-/-)-Mausembryonalfibroblasten und einer bedingt aktiven Form der menschlichen PKR zeigen wir hier, dass die Tyrosinphosphorylierung (aber nicht die Serinphosphorylierung) von Stat1 oder Stat3 in Zellen mit aktivierter Kinase beeinträchtigt ist. Diese Verringerung der Stat1- und Stat3-Tyrosinphosphorylierung durch aktive PKR erfolgt über TC-PTP, das sowohl in vitro als auch in vivo ein Substrat der eukaryotischen Initiationsfaktor-2alpha-Kinase ist. Die TC-PTP-Phosphorylierung allein reicht nicht aus, um die Phosphataseaktivität in vivo zu erhöhen, es sei denn, sie geht mit einer Hemmung der Proteinsynthese als Folge der PKR-Aktivierung einher. Diese Daten offenbaren eine neue Funktion von PKR als negativer Regulator von Stat1 und Stat3 mit wichtigen Auswirkungen auf die Zellsignalisierung.”
Keine Einbahnstraße. Einige eIFs geben also Feedbackloops an weitere Signaltransduktionswege.
“Wir zeigen auch, dass PKR als Substrat von aktivierten Jaks wirkt und sowohl in vitro als auch in vivo an Tyr 101 und Tyr 293 phosphoryliert wird. Darüber hinaus liefern wir eindeutige Beweise dafür, dass sowohl die Induktion der eIF2α-Phosphorylierung als auch die Hemmung der Proteinsynthese durch IFN in Zellen, denen Jak1 oder Tyk2 fehlt, beeinträchtigt sind, was einer fehlenden Induktion der PKR-Tyrosinphosphorylierung entspricht.”
+
„Nicht-autonome Aktivierung von PERK in ISCs durch JAK/Stat-Signalisierung
Unsere Ergebnisse deuten darauf hin, dass ISCs {Intestinal Stem Cells] durch die Aktivierung von PERK eine regenerative Reaktion sowohl auf zellautonomen als auch auf gewebsweiten ER-Stress einleiten. Wir bestätigten die Vorstellung einer nicht-autonomen Kontrolle der PERK-Aktivität in ISCs, indem wir die Phosphorylierung von eIF2α in ISCs von Tieren untersuchten, bei denen ER-Stress spezifisch in EBs, ECs, Fettkörper oder Muskel induziert wurde. Um die ER-Proteostase in diesen Zellen und Geweben zu stören, haben wir Xbp1 ausgeschaltet (mit Su(H)Gbe::Gal4, tub::G80ts für EBs, NP1::Gal4, tub::Gal80ts für ECs, ppl::Gal4, tub::G80ts für Fettkörper und How::Gal4, tub::G80ts für Muskeln). Der Knockdown von Xbp1 in EBs oder ECs führt zu einer nicht-autonomen Aktivierung der ISC-Proliferation (Wang et al., 2014) und, im Einklang mit einer ISC-spezifischen Aktivierung von PERK, auch zu einer erhöhten Phosphorylierung von eIF2α in ISCs (Abb. 4A, 4B und S2B, beachten Sie die eIF2α-Phosphorylierung in ISCs, die an GFP-exprimierende EBs in Abb. 4A grenzen, oder in Dl+ ISCs in Abb. 4B). Die Ausschaltung von Xbp1 im Fettkörper oder in der Muskulatur erhöhte jedoch nicht die ISC-Proliferation oder stimulierte die eIF2α-Phosphorylierung in ISCs (Abb. S2C und S2D), was darauf hindeutet, dass die nicht-autonome Regulierung von PERK auf Zell/Zell-Interaktionen innerhalb des Darmepithels beschränkt ist.“
”Jüngste Erkenntnisse haben jedoch gezeigt, dass Stat1 über noch nicht ausreichend verstandene Mechanismen eine Resistenz gegen Antitumor-Medikamente vermitteln kann. Wir zeigen hier die Fähigkeit von Stat1, die Phosphoinositol-3-Kinase γ (PI3Kγ)-Signalübertragung zu induzieren und die allgemeine Proteinsynthese zu hemmen, was zur Übersetzung ausgewählter mRNAs führt, die für Proteine kodieren, die die Zellproliferation hemmen oder Zellen zunehmend resistent gegen Antitumor-Medikamente machen.”
(…)
“Eine verstärkte PI3Kγ-Signalisierung fördert den Abbau des eIF4A-Inhibitors, des programmierten Zelltodproteins 4, was die cap-unabhängige Translation der ausgewählten mRNAs unter den Bedingungen einer allgemeinen Hemmung der Proteinsynthese durch das hochregulierte eIF4E-bindende Protein 1 begünstigt. So hemmt Stat1 die Zellproliferation, macht die Zellen aber auch zunehmend resistent gegen die antiproliferativen Wirkungen pharmakologischer PI3K-Inhibitoren und/oder Mammalian Target of Rapamycin. Stat1 schützt auch Ras-transformierte Zellen vor den genotoxischen Wirkungen von Doxorubicin in Kulturen und immunschwachen Mäusen. Unsere Ergebnisse zeigen, dass die mRNA-Translation eine wichtige Rolle bei den zellautonomen Stat1-Funktionen spielt, was sich auf das Tumorwachstum und die Behandlung mit Chemotherapeutika auswirkt.”
Na bitte. Ist doch ein weiteres hübsches Beispiel, wieso ich immer wieder auf Crosstalks verweise?
Denkt gerade noch jemand an Frankensteinproteine, ohne 5’Cap? Erinnert ihr euch noch an die Integritätslüge? Der Computer sagt nein.
Covid-19: Researchers face wait for patient level data from Pfizer and Moderna vaccine trials
“Notably, ‘Process 2’ batches were shown to have substantially lower mRNA integrity.”
Wobei man natürlich - wenn man richtig nerdig wäre und böse Absichten unterstellt - noch nach versteckten IRES-Mechanismen (Insider für Hardcorenerds) in der modRNA-Brühe fragen würde. Tu’ ich natürlich niemals nie nicht!😶
Zurück zu den eIFs: Ich wäre ja nicht Nerd, würde ich nicht die hübschesten Folien für die Problemstellung finden.
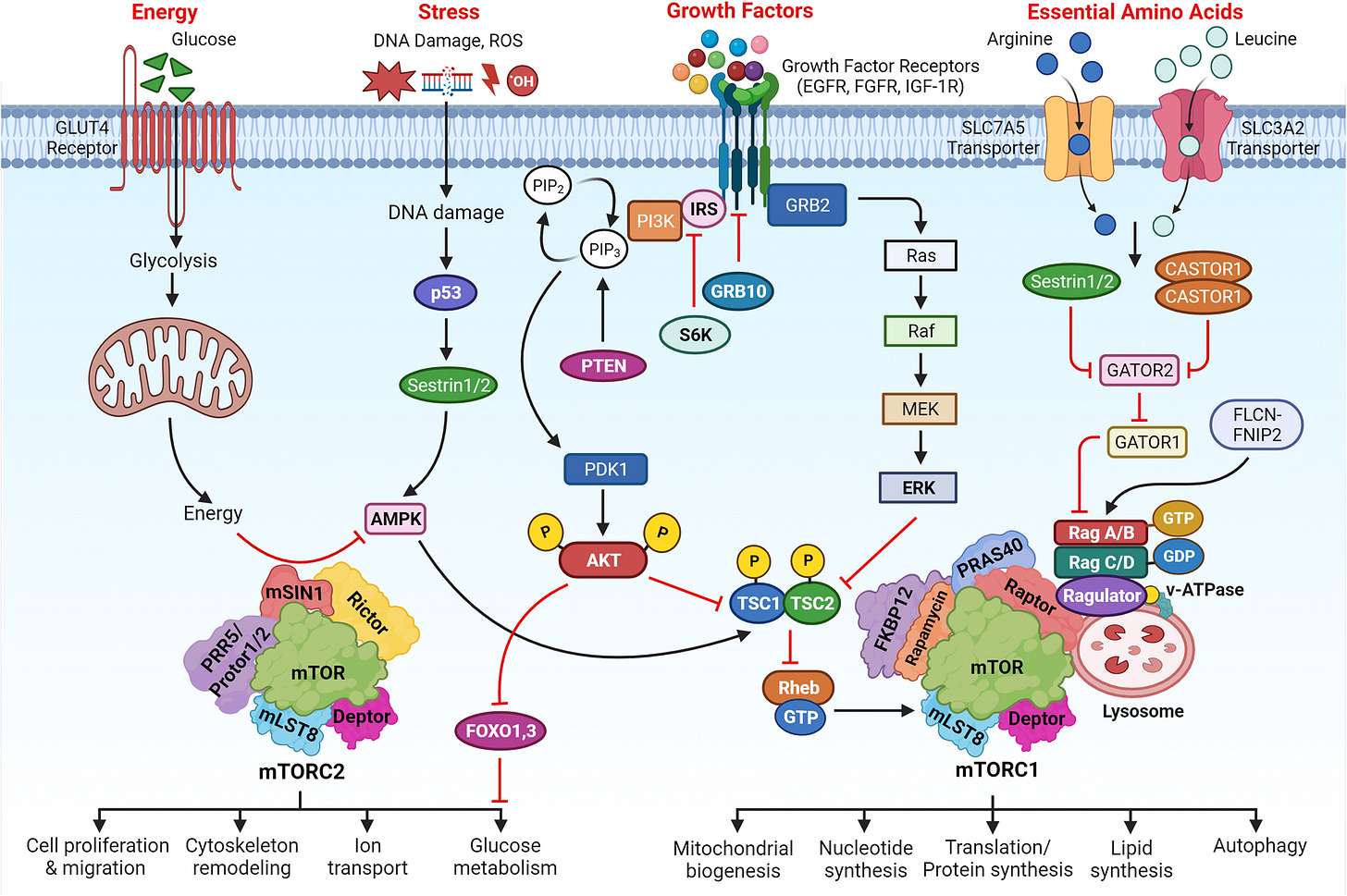
mTOR und MAPk
Ich werde diese beiden Kaskaden zusammenfassen wegen ihren starken Crosstalkaktivitäten.
eIF3-f function in skeletal muscles
Manchmal kann man Signaltransduktion nicht besser erklären, als mit Folien. Ihr braucht blos den “aktiviert” / “inhibiert” Pfeilen zu folgen.
mTOR‐dependent stimulation of the association of eIF4G and eIF3 by insulin
“Insulin stimuliert die Proteinsynthese, indem es die Initiierung der Translation erhöht. Diese Reaktion wird durch mTOR vermittelt und ist vermutlich auf die Phosphorylierung von 4EBP1 zurückzuführen, die es eIF4E ermöglicht, eIF4G zu binden. Hier präsentieren wir Beweise dafür, dass mTOR direkt mit eIF3 interagiert und dass mTOR die Assoziation von eIF3 und eIF4G kontrolliert. “
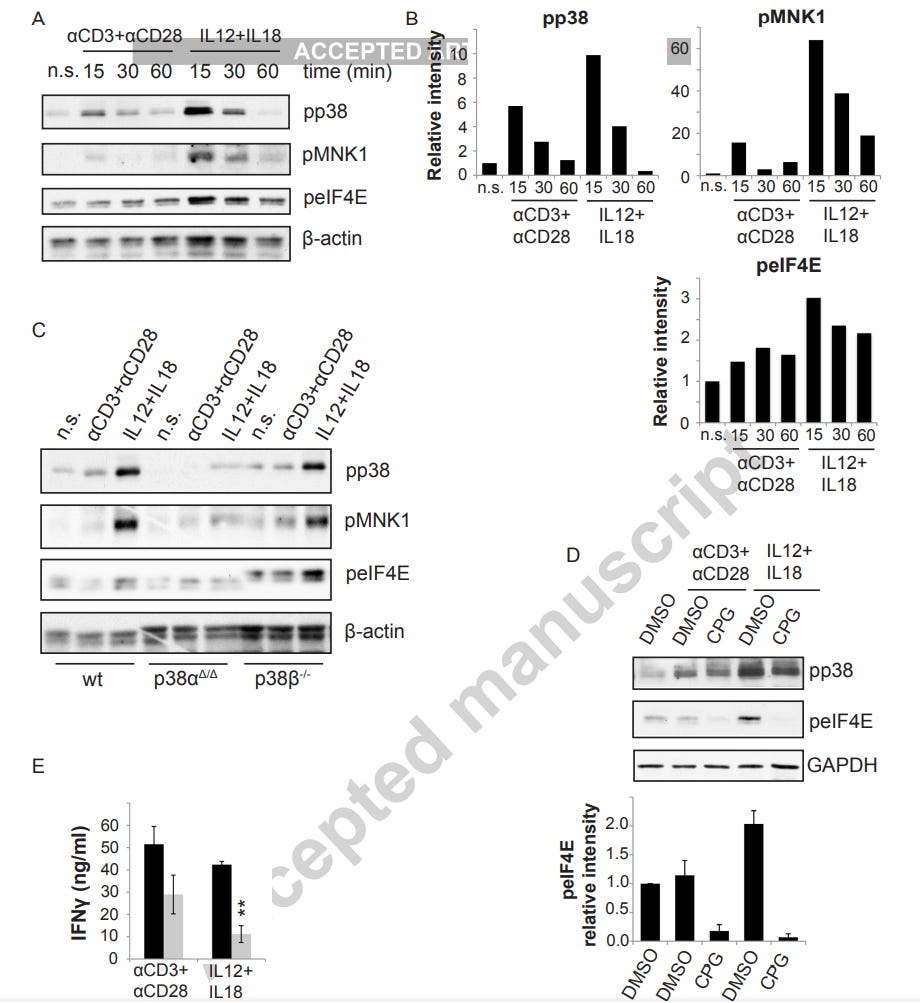
p38α regulates cytokine-induced IFNγ secretion via the Mnk1/eIF4E pathway in Th1 cells (‼maturierte CD4+ Immunzelle TH1‼)
)
“Das Fehlen von p38α in Th1-Zellen führte jedoch zu einer verringerten MNK1-Phosphorylierung nach Zytokinaktivierung, und die MNK1-Hemmung blockierte die IFNγ-Produktion. Unsere Ergebnisse deuten darauf hin, dass p38α die IFNγ-Sekretion durch die Aktivierung des MNK1/eIF4E-Weges der Translationsinitiierung reguliert und spezifische Funktionen für p38α und p38β bei der T-Zell-Proliferation identifiziert.”
Na? Wird euch auch schon warm ums Herz, darüber nachzudenken, dass es keine one size fits on all Regulation gibt und auch hier wieder Ausnahmeregulationen stattfinden?
“Die mitogene Stimulation führte zu einer Phosphorylierung des eukaryotischen Initiationsfaktors (eIF)-4E. Die Hemmung der p38-MAPK-Signalübertragung oder die funktionelle Dysregulierung der Translation durch kleine interferierende doppelsträngige RNA (siRNA) gegen eIF-4E verringerte das von der Verankerung unabhängige Wachstum maligner Cholangiozyten. Zusammenfassend lässt sich sagen, dass diese Studien einen Zusammenhang zwischen der p38-MAPK-Aktivität und der Regulierung der Proteinsynthese während des Wachstums von menschlichen Cholangiokarzinomen aufzeigen. Da die Proteinsynthese eng mit dem Zellwachstum verbunden ist, ist die Dysregulation der Translationsinitiierung ein Mechanismus, durch den die zelluläre p38-MAPK-Signalgebung an der Wachstumsregulation maligner Cholangiozyten beteiligt ist.”
Das folgende paper ist der absolute Kracher!
”Somit scheint die MAPK/ERK-Signalübertragung trotz reduzierter MAPK/ERK-Proteinspiegel aktiviert zu werden, wenn die eIF3-Funktion bei der Translation beeinträchtigt ist.”
“Darüber hinaus beobachteten wir, dass alle drei Knockdowns c-Jun aktivierten, was durch seine erhöhte nukleare Translokation belegt wurde. Obwohl zuvor gezeigt wurde, dass die JUN-mRNA über eine stabile Stammschleife in ihrem 5'-UTR direkt mit eIF3 interagiert, was ihre Translation positiv stimulierte (Lee et al., 2015), beobachteten wir in dieser Studie eine Hochregulierung der JUN-mRNA-Häufigkeit, wobei die Abreicherung von eIF3d, e oder h keine auffälligen Auswirkungen auf ihre Translationseffizienz hatte.“
Kleiner Reminder dazu:
C-JUN-N-Terminal Kinase (JNK → MAPk)
Novel role of c-jun N-terminal kinase in regulating the initiation of cap-dependent translation
“Da JNK an der Entstehung von nicht-kleinzelligem Lungenkrebs (NSCLC) beteiligt ist, stellten wir die Hypothese auf, dass JNK auch an der Aktivierung der cap-abhängigen Translation in NSCLC-Zellen beteiligt sein könnte und einen alternativen Weg zur Regulierung der Translation darstellen könnte. In einer Reihe von NSCLC-Zelllinien führte die Hemmung von JNK mit SP600125 zu einer Hemmung der 4E-BP1-Phosphorylierung und zu einem Rückgang der Bildung des cap-abhängigen Translationskomplexes eIF4F.”
Puh 3 der 4 großen, auf die sich meine Folie fokussiert, geschafft! Entschuldigt, dass ich nicht noch viel näher auf all diese Papers eingehe. Ich kann nur Beispiele liefern, damit - wer es verstehen will - ihr eine grobe Orientierungshilfe habt und euch tiefer in die ganzen Zusammenhänge einlesen könnt. Denn der Irrsinn besteht eben darin, dass wir für 99 Prozent der hier gebrachten Beispiele kein tiefergreifendes, allgemeingültiges Verständnis entwickelt haben. Es ist also eher reine Interpretation und eine - soweit möglich - in sich stringente Überlegung, der ich hier nachgehe, welche - in meinen Augen - noch am plausibelsten wirkt.
Auf zur letzten großen! Auf zu cAMP! Keine Panik! Ihr habt es fast (vorerst *diabolisches Kichern* 😈) geschafft: Da ich zu cAMP direkt nur ein altes Paper fand und AMP(k) nicht noch extra behandeln möchte, welches eiF direkt downstream erwähnt, habe ich auf einen Pfeil zu eIF verzichtet. Ich beanspruche keinerlei Vollständigkeit. Wie ich jedoch in meinen vorangegangenen Teilen schon zeigte, wird cAMP durch die Crosstalks auch die Translation beeinflussen.
“Die Translation in heminhaltigen Retikulozyten-Lysaten wird auch durch zyklische AMP-abhängige Proteinkinasen oder deren katalytische Untereinheiten gehemmt. Sie wirken, indem sie den in den Lysaten vorhandenen Proinhibitor (inaktive eIF-2-Kinase) in Inhibitor (aktive eIF-2-Kinase) umwandeln. Diese Reaktion ist analog zur Umwandlung von inaktiver Phosphorylase-Kinase in aktive Phosphorylase-Kinase.”
Mich würde es also nicht überraschen, wenn cAMP (oder Substrate wie AMP(k)) auch direkt noch regulatorische Einflüsse auf die eIFs ausübt, die wir noch nicht besser charakterisierten. Noch dazu kommen ja all die hübschen Crosstalks.
Also nicht wundern, dass es weh tun könnte, wenn man die Eingangssignalgebung durch Membranpenetration ein wenig, äh, “sicher und wirksam”™ umgestaltet.
Aber wie der Stefan (Bayer AG) ja schon so nett ausführte, ist er nicht an eurer sondern der globalen Gesundheit interessiert. Und ich würde ihm beipflichten, dass diese Brühe nicht nur eine Gentherapie darstellt, sondern eine komplette “Zellumstrukturierungstherapie”.