Downstream Teil 3
Meine erste Folie, mit der ich zufrieden bin und die mir zeitgleich Angst macht.
3. Signaltransduktion: Das Alphabet der Zelle & die Zellmembran
Bevor es also langsam in die Vollen geht und wir uns den Crosstalks und Downstreameffekten widmen, brauchen wir noch ein weiteres Verständnis für die Events, die alleine die LNPs anrichten werden (ob gefüllt oder leer wäre aus dieser Perspektive insoweit vernachlässigbar, dass das Basisprinzip gleich bleibt und lediglich die Komplexität der gestörten Netzwerkinteraktionen steigt).
Fachtermini:
Um meinen Gedanken folgen zu können, müsst ihr zumindest die Grundprinzipien verstehen und unterscheiden können, was Signalmoleküle sind und was Zielgene (mRNA) und Zielproteine wie Transkriptionsfaktoren, Initiationsfaktoren, etc. sind. Dazu empfehle ich dieses Substack von mir und folgendes Review, welches ich auch ein wenig zitieren werde. - Eigentlich erklärt sich hier alles von selbst und ich muss nichts mehr zusammenfassen.
Protein Regulation in Signal Transduction
”Zellsignale steuern die Anzahl der Proteine, die Proteinlokalisierung, die Proteinaktivität und die Protein-Protein-Wechselwirkungen, oft über posttranslationale Modifikationen (z. B. Ubiquitylierung) oder Änderungen der Genexpression oder Proteinsynthese.
Signaltransduktionsprozesse sind in vielerlei Hinsicht proteingesteuerte Ereignisse. Eine gängige Methode zur Beschreibung dieser Schaltkreise besteht darin, verschiedene Proteine oder modulare Domänen innerhalb eines einzelnen Proteins als „Leser“, „Schreiber“ und „Radierer“ zu bezeichnen. In diesem Modell werden katalytische Domänen, die spezifische posttranslationale Modifikationen vornehmen, wie Kinasen und Acetyltransferasen, als „Schreiber“ bezeichnet, weil sie auf den Proteinen, auf die sie einwirken, eine physische Markierung hinterlassen. Umgekehrt sind Phosphatasen, Deacetylasen und andere Enzyme, die diese Modifikationen entfernen, Beispiele für Radiergummis. Neben Schreibern und Radierern gibt es auch Domänen, die an spezifische posttranslational modifizierte oder unmodifizierte Aminosäuresequenzen binden. Diese Arten von Domänen lesen die von Writern und Erasern erzeugten Sequenzen und werden daher Reader genannt. Diese Leser können an Protein-Protein-Wechselwirkungen oder Wechselwirkungen zwischen zwei Teilen desselben Proteins beteiligt sein. Andere Reader können direkt an spezifische Phospho- und neutrale Lipide oder an spezifische Ionen, wie z. B. Kalzium, binden, anstatt an Aminosäuresequenzen. Die Ziele dieser Leser, Schreiber und Radierer sind oft kurze Aminosäuresequenzen (in der Regel drei bis 15 Aminosäuren lang), die als Motive bezeichnet werden. Mit Hilfe dieser modularen Teile kodieren die Zellen dynamisch Informationen als Reaktion auf Umwelt-, chemische oder Entwicklungsreize, um das Signal weiterzuleiten (siehe Tabelle 1).”
Einfacher kann man den Grundgedanken hinter Signaltransduktion nicht erklären, was die Proteinregulierung angeht. Obwohl…. Vllt. eine kleine Analogie: Stellt es euch wie eine Art Alphabet der Zellen vor, mit wesentlich komplizierteren Grundregeln in 3D (Zeit, Raum, Distanz, Ladung, Intensität und Bindungsstelle), um ein Wort zu formen, welches dazu führt, eine bestimmte Aktion zu forcieren. Und weiter im Programm:
“5.2. RNA-Stabilität und miRNAs
Die Signaltransduktion kann auch eine Modulation der mRNA-Verarbeitung oder -Stabilität beinhalten. Mehrere Spleißfaktoren werden als Reaktion auf die Signalübertragung phosphoryliert, und diese Ereignisse können das alternative Spleißen von Prä-mRNAs zu verschiedenen mRNA-Isoformen steuern (Lynch 2007). Zellen können die mRNA-Stabilität regulieren, indem sie die mRNA-Reifung (über 5′-Capping oder 3′-Polyadenylierung) oder die Wechselwirkungen zwischen RNA-bindenden Proteinen und mRNA-Transkripten modulieren (Wu und Brewer 2012). Ein neuer Forschungsbereich ist die Kontrolle der Signalübertragung durch zelluläre Mikro-RNAs (miRNAs) und andere nichtcodierende RNAs, wie lange nichtcodierende RNAs (lnc-RNAs). miRNAs sind kurze (∼21-23 Nukleotide) nichttranslatierte RNAs (Ambros 2001), die typischerweise den Abbau von Ziel-RNAs induzieren oder die mRNA-Translation durch sequenzspezifische Basenpaarungsinteraktionen blockieren. Sie sind wichtige Regulatoren des normalen Entwicklungszeitplans (Ambros 2011). miRNAs wie Lin-4/mir-125 regulieren die zeitlichen Übergänge zwischen pluripotenten und differenzierten Zuständen zahlreicher Stammzellpopulationen.
miRNAs sind auch häufige Regulatoren der Dynamik, Dauer und Empfindlichkeit von Signalisierungsprozessen. Viele Signalwege - darunter beispielsweise der Wnt-, Notch-, Hedgehog- und p53-Signalweg - erreichen ihre Spezifität und Empfindlichkeit durch aktive Repression (d. h. basale Repression oder Standardrepression). In diesem Zusammenhang zielen miRNAs auf Kernkomponenten des Weges oder ihre Transkriptionsziele ab und halten die Zellen in einem inaktiven Zustand, so dass das System zur Aktivierung größere Mengen an Stimuli benötigt und auch die Reaktion auf aktivierende Stimuli verschärft wird. Zum Beispiel zielt miR-125 auf viele Komponenten des p53-Signalwegs ab, und der Verlust von miR-125 führt zu einer spontanen Aktivierung von p53.”
Ich weiß, ich springe jetzt ein wenig hin und her in diesem Review. Mir geht es jedoch um drei wichtige Aspekte, die ich in meiner Folie auch zeige:
“Neben der Veränderung der enzymatischen Aktivität eines Proteins kann die Phosphorylierung auch die Wechselwirkungen zwischen zwei oder mehreren Proteinen stören oder zwei Proteine zur Interaktion veranlassen (siehe unten), wodurch sich häufig der subzelluläre Standort des phosphorylierten Proteins ändert.
Eine zweite häufige reversible posttranslationale Modifikation von Proteinen ist die Acetylierung. In diesem Fall werden die positiv geladenen ɛ-Aminogruppen an Lysinresten durch Zugabe von Acetat in neutrale Amide umgewandelt. Dieser Verlust an positiver Ladung verhindert, dass die acetylierten Lysine elektrostatische Wechselwirkungen mit Phosphatgruppen eingehen können, und ist daher eine wichtige posttranslationale Modifikation, die an DNA-bindenden Histonen zu finden ist. Da das DNA-Grundgerüst aus Phosphat- und Zuckerestern aufgebaut ist, schwächt die Acetylierung die Bindung der Histone in den Nukleosomen an die DNA, so dass andere DNA-bindende Proteine wie Transkriptionsfaktoren und RNA-Polymerase stattdessen binden können. Dies führt häufig zu Veränderungen der Chromatinstruktur und der Transkriptionsaktivität. Die Acetylierung kommt auch bei Enzymen vor, die am Stoffwechsel beteiligt sind (Wang et al. 2010), und funktioniert wahrscheinlich zumindest teilweise durch eine Veränderung der Aktivität des Enzyms, ähnlich wie die Phosphorylierung von Proteinen. Wie die Phosphorylierung kann auch die Acetylierung von Proteinen dazu führen, dass sich zwei Proteine aneinander binden, wenn eines der Proteine eine Domäne besitzt, die spezifisch Acetyl-Lysin erkennt (z. B. eine Brom-Domäne), oder sie kann spezifisch die Dynamik der Rekrutierung anderer Proteine an das acetylierte Protein im Vergleich zur unmodifizierten Form erhöhen. So fördert beispielsweise die Acetylierung der DNA-Schadenskinase ATM durch die Acetyltransferase Tip60 (auch bekannt als Kat5) die Rekrutierung von ATM an Stellen mit DNA-Schäden und ATM-abhängigen Signalen. In ähnlicher Weise sind Mikrotubuli, die acetyliertes Tubulin enthalten, besser in der Lage, molekulare Motoren zu rekrutieren, die den vesikulären Transport innerhalb der Zelle steuern (Perdiz et al. 2011). Wie im Falle der Phosphorylierung wird die Acetylierung durch Deacetylasen ausgeglichen. Früher ging man davon aus, dass Histonproteine die Hauptziele der Acetylierung sind. Acetyltransferasen und Deacetylasen werden daher gemeinhin als Histon-Acetyltransferasen (HAT) bzw. Histon-Deacetylasen (HDAC) bezeichnet. Da jedoch keine dieser Enzymklassen spezifisch für Histonproteine ist, sind die Bezeichnungen Lysin-Acetyltranferase (KAT) und Lysin-Deacetylase (KDAC) angemessener.”
“5.3. Translationskontrolle
Die Translation ist auch ein Ziel von Signaltransduktionswegen. So ist beispielsweise bekannt, dass der mTOR-Komplex (mammalian target of rapamycin) und verwandte Signalwege die Proteintranslation als Reaktion auf Nährstoff-, Wachstumsfaktor- und Aminosäuresignale steuern (Laplante und Sabatini 2012). Ein entscheidendes Ziel, das an der Regulierung der Translation durch den mTORC1-Komplex beteiligt ist, ist das eukaryotische Translationsinitiationsfaktor 4E (eIF4E)-bindende Protein 1 (4E-BP1) (Hara et al. 1997). 4E-BP1 hemmt die cap-abhängige Translation durch Bindung an eIF4E, den Initiationsfaktor, der das 5′-Cap auf mRNA-Molekülen erkennt. Es wird angenommen, dass die Phosphorylierung von 4E-BP1 an T37 oder T46 4E-BP1 für die nachfolgende Phosphorylierung an S65 und T70 vorbereitet, was zum Verlust der Interaktion mit eIF4E führt (Gingras et al. 1999), was zu einer allgemeinen Zunahme der Proteintranslation führt. Der mTORC1-Komplex reguliert auch die Aktivität der p70-S6-Kinase 1 (S6K1), die viele Ziele hat, deren Phosphorylierung die Translation aktiviert (Pullen und Thomas 1997). Eines der wichtigsten Ziele ist die S6-Untereinheit des 40S-Ribosoms, die, wenn sie von S6K1 phosphoryliert wird, die Translation von mRNA-Transkripten fördert, die eine Oligopyrimidin-Sequenz in ihrer 5′ untranslatierten Region (5′-UTR) enthalten. Darüber hinaus kann die Translation spezifischer mRNAs durch Rückkopplungssignale auf der Ebene der Initiierung der Translation gesteuert werden. So wird beispielsweise die Eisenhomöostase zum Teil durch die Regulierung der Translation von Proteinen aufrechterhalten, die am Eisenimport beteiligt sind. In Gegenwart hoher Konzentrationen von intrazellulärem Eisen wird die Transferrinrezeptor-mRNA durch die Eisen-induzierte Bindung des Eisen-Response-Element-bindenden Proteins (IREBP) an ein 5′-UTR-Element in der Transferrinrezeptor-mRNA unterdrückt.”
Ah… Schaut mal: Translation? Check. Direkte Regulation der Transkriptionsfaktoren? Check. miRNA? Check. Leider haben die Autoren doch glatt den endosomalen Escape und die Membranreorganisation via PtdIns-Zyklus, sowie die Regulation von miRNA-Expressionen und Splicing vergessen. Na egal. Dazu haben sie ja mich. Als Erinnerung dazu:
The structural basis for Ras activation of PI3Kα lipid kinase
In meinem Substack über die phospholipide Doppelschicht und die Membranorganisation beschrieb ich ja schon, dass PI3K via RAS aktiviert wird und eine essentielle Rolle im PtdIns-Zyklus und dem Recyclen der Phospholipide spielt.
Pardon für diese lange Vorbereitung. Und jetzt wird es eben echt haarig: Schauen wir uns also die 4 großen Signalkaskaden an und beweisen “kurz” (hust, räusper), dass es zwingende Alterationen nach einer Transfektion geben muss, da faktisch alle die Membran und den endosomalen Escape beeinflussen und von ihr beeinflusst werden. Eine kurze Randbemerkung noch, bevor wir Beispiele für die roten Pfeile in meiner Folie suchen: JAK ist transmembran gebunden. Also ist bereits das Eingangssignal - was logisch erscheint - ebenfalls von der Membranintegrität abhängig.
Siehe:
Cytokine Signaling: STATS IN PLASMA MEMBRANE RAFTS
“Die Daten zeigen eine Vorassoziation von STATs mit Plasmamembran-Rafts in Flotationsfraktionen, die auch Caveolin-1 und Flotillin-1 enthielten, und deuten darauf hin, dass die Tyr-Phosphorylierung an sich nicht ausreicht, um die Abspaltung von PY-STATs von Plasmamembran-Rafts zu bewirken. Methyl-β-Cyclodextrin, das Cholesterin sequestriert und die Plasmamembran-Rafts unterbricht, hemmte die IL-6- und IFN-γ-induzierte STAT-Signalgebung deutlich. Die Signalübertragung durch spezialisierte Raft-Mikrodomänen könnte ein allgemeiner Mechanismus auf der Ebene der Plasmamembran sein, durch den Zytokine und Wachstumsfaktoren STAT-Spezies aktivieren (die „Raft-STAT-Signalisierungshypothese“).”
Uff. PY-STAT = phosphoryliertes Tyrosin gebundenes STAT. Ich glaube, den muss ich kurz erklären. Begriff “RAFTS”:
https://www.sciencedirect.com/topics/agricultural-and-biological-sciences/lipid-raft
”Lipid Rafts sind spezialisierte Glykolipoprotein-Mikrodomänen auf der Zellmembran, die aus einer Kombination von Glykosphingolipiden (hauptsächlich Cholesterin) und Proteinen (hauptsächlich Rezeptoren) bestehen .”
Was ultrakompliziert klingt, heißt eigentlich nichts anderes, als dass bestimmte Formänderungen und die Losbindung von bestimmten Teilen des aktivierten STAT-Signals (was dann downstream zur Kaskade führt), nicht statt fand, wenn Tyrosin phosphoryliert war, sobald diese Rafts unterbrochen wurden.
Gut, gut und los gehts:
Wir widmen uns zunächst dem Einfluss von cAMP als AKT-Inhibitor und folgen den roten Pfeilen:
Wir starten mit dem cAMP. (Dass die MAPks (genauso wie mTOR) ein direkter Regulator des PtdIns Zyklus (beispielsweise RAS als aktivierendes Signalmolekül von PI3K als Effektorprotein) zeigte ich ja schon in meinem verlinkten Substack zur Phospholipiden Doppelschicht. (Das werde ich hier also nicht noch einmal explizit durchkauen.)
In diese Studie müssen wir ein wenig näher gucken:
”Der Gehalt an PtdIns 3,4,5-P3 wird durch mehrere Mitglieder der PtdIns 3-Kinase-Familie stimuliert und durch zwei Phosphatidylinositol-Phosphatasen bekämpft: die Src Homology 2 (SH2) domain-containing inositol 5′ phosphatase (SHIP) und die 3′ inositol phosphatase, phosphatase and tensin homolog (PTEN). PTEN entfernt Phosphat aus der 3′-Position im Inositolring von PtdIns 3,4,5-P3 und wandelt es in Phosphatidylinositol 4,5-bisphosphat um. PTEN besitzt eine C2-Domäne, ein PDZ-Bindungsmotiv und ein N-terminales Phosphatidylinositol-4,5-bisphosphat-Bindungsmotiv, das für die Translokation zur Membran und die Interaktion mit anderen regulatorischen Proteinen wichtig ist. Es gibt Serin- und Threoninreste in PTEN, die nachweislich phosphoryliert werden, deren Rolle bei der Regulierung der Aktivität des Enzyms jedoch unklar ist. Mutationen im PTEN-Protein wurden in vielen Tumoren beobachtet, was auf eine Rolle dieses Enzyms bei Krebserkrankungen hindeutet.
Im Gegensatz dazu dephosphoryliert SHIP die 5′-Position am Inositolring und produziert Phosphatidylinositol-3,4-bisphosphat. Es gibt drei SHIP-Isoformen: das auf hämatopoetische Zellen beschränkte 145-kDa-SHIP (auch bekannt als SHIP1), das auf Stammzellen beschränkte 104-kDa-SHIP (sSHIP) und das weiter verbreitete 150-kDa-SHIP2 (11). SHIP1 ist die wichtigste Inosit-Phosphatase, die PtdIns 3,4,5-P3 in Monozyten, Makrophagen, B-Zellen und T-Zellen reguliert (11). SHIP1 hat drei bekannte strukturelle Merkmale: die N-terminale SH2-Domäne, die zentrale Inositol-5′-Phosphatase-Domäne und zwei NPX-Y-Sequenzenin der C-terminalen Region. Das derzeit akzeptierte Modell für die Regulierung des PtdIns 3,4,5-P3-Spiegels durch SHIP1 sieht eine Translokation von SHIP1 aus dem Zytosol zur Membran vor. Bei Stimulation durch Wachstumsfaktoren, Zytokinrezeptoren oder Immunrezeptoren wird SHIP1 über seine N-terminale SH2-Domäne an phosphorylierte Tyrosinreste in Rezeptorkinasen rekrutiert und baut die erhöhten PtdIns 3,4,5-P3-Konzentration in der Nähe des aktivierten Rezeptors ab. Es wird angenommen, dass SHIP1 während dieses Translokationsprozesses seine 5′-Phosphatase-Aktivität nicht ändert. Obwohl bekannt ist, dass SHIP1 durch die zytoplasmatische Kinase lyn (12) oder nach der Aktivierung des T-Zell-Rezeptors an Tyrosinresten phosphoryliert werden kann, scheinen beide Ereignisse die 5′-Phosphataseaktivität nicht zu beeinflussen. Bislang wurde die direkte Regulierung der SHIP1-Aktivität durch Serin/Threonin-Kinasen noch nicht untersucht.
Es ist bekannt, dass die Aktivierung von G-Protein-gekoppelten Rezeptoren, die cAMP erhöhen (d. h. β-adrenerge Rezeptoren oder Adenosin-A2a-Rezeptoren ), die pro-inflammatorischen Reaktionen abschwächt, die durch Rezeptoren ausgelöst werden, die den PtdIns 3,4,5-P3-Spiegel erhöhen. Daher untersuchten wir die Möglichkeit, dass die Phosphorylierung von SHIP1 durch die zyklische AMP-abhängige Proteinkinase (PKA) die Aktivität von SHIP1 regulieren könnte. Wir fanden heraus, dass SHIP1 sowohl in vitro als auch in Zellen durch PKA phosphoryliert werden kann, was zu einer Stimulation der SHIP1-Aktivität führt. Die Aktivierung von PKA in DT40- und A20-Zellen führte zu einer Abschwächung der Indikatoren für die PtdIns 3,4,5-P3 -Antwort auf die Stimulation durch B-Zell-Rezeptoren. Diese Ergebnisse deuten darauf hin, dass die SHIP1-Aktivität sowohl in vitro als auch in Zellen durch die Aktivierung der zyklischen AMP-abhängigen Proteinkinase reguliert werden kann, und zeigen einen neuen Modus der SHIP-Regulierung durch G-Protein-gekoppelte Rezeptoren auf.”
Cyclic AMP Inhibits Akt Activity by Blocking the Membrane Localization of PDK1
“Hier zeigen wir, dass cAMP hemmende Wirkungen auf den Phosphatidylinositol-3-Kinase/PDK/Akt-Signalweg hat. cAMP hemmt die Phosphorylierung an Threonin 308 und Serin 473 von Akt, was für die Proteinkinase-Aktivitäten von Akt erforderlich ist. cAMP reguliert auch PDK1 negativ, indem es seine Verlagerung zur Plasmamembran hemmt, obwohl es seine Proteinkinase-Aktivitäten nicht beeinträchtigt.”
Das heißt also, dass cAMP einen direkten Einfluss, durch die Interaktionen mit PI3K/AKT auf die PtdIns-Zyklen hat.
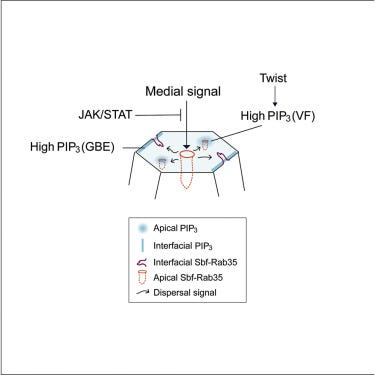
A PtdIns(3,4,5)P3 dispersal switch engages cell ratcheting at specific cell surfaces
JAK beeinflusst demnach auch die Membranstruktur indirekt über ein mittleres Signal, welches ebenfalls PIP3 ansteuert. Man könnte die Hypothese daraus folgerichtig aufstellen, dass es auch direkt den PtdIns-Zyklus zusätzlich regulieren wird. (Leider fand ich keine weitere Forschung in diesem Kontext.)
Spätestens an diesem Punkt, sollte jedem klar sein, der auch nur einen Moment darüber nachdenkt, ob LNPs jemals funktionieren könnten oder nicht:
Falls ihr euch bis hierhin fragt, wieso ich dieses Substack in einzelne Teile aufsplitte. Ab dem kommenden Teil, wird es so haarig und komplex, dass sich die Frage von selbst beantworten wird.
Coming soon:
4. Das Huhn-Ei Problem der Crosstalks: Was war zuerst da?: c(yclic)AMP, MAPk, JAK/STAT oder mTORC1/2?